Creative Proteomics Blog
sekvensen af aminosyrer i et protein eller peptid kan identificeres ved Edman-nedbrydning, som blev udviklet af Pehr Edman. Denne metode kan mærke og spalte peptidet fra N-terminal uden at forstyrre peptidbindingerne mellem andre aminosyrerester. Edman-nedbrydningsreaktionen blev automatiseret i 1967 af Edman og Beggs. I dag anvendes den automatiserede Edman-nedbrydning (proteinsekvensatoren) bredt, og den kan sekvensere peptider op til 50 aminosyrer.
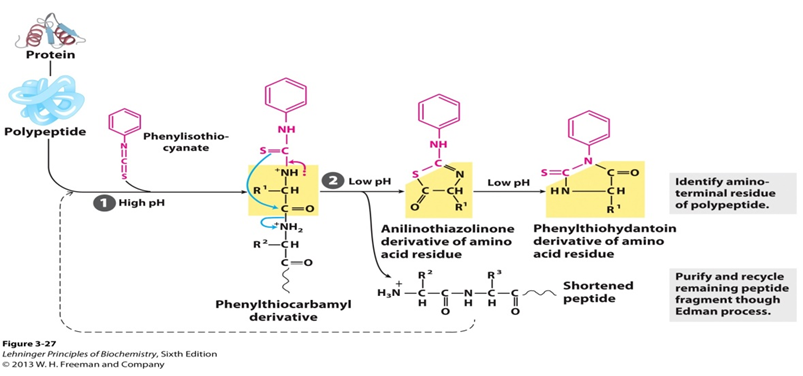
cyklisk nedbrydning af peptider baseret på reaktionen af phenylisothiocyanat med den frie aminogruppe af den N-terminale rest, således at aminosyrer fjernes en ad gangen og identificeres som deres phenylthiohydantoinderivater. Når man taler til den specifikke proces, omsættes et uladet peptid med phenylisothiocyanat (PITC) ved amino-terminalen under mildt alkaliske betingelser for at give et phenylthiocarbamoylderivat (PTC-peptid). Derefter angriber thiocarbonylsulfen af derivatet under sure betingelser carbonylcarbonatet af den N-terminale aminosyre. Den første aminosyre spaltes som anilinothiaminonderivat, og resten af peptidet kan isoleres og underkastes den næste nedbrydningscyklus. Når det er dannet, er dette derivat mere stabilt end phenylthiocarbamylderivat. Aminosyren fjernes derefter ved ekstraktion med ethylacerat og omdannes til et phenylthiohydantoinderivat (PTH-aminosyre). Og kromatografien kan bruges til at identificere den PTH-Rest, der genereres af hver cyklus.
med hensyn til den automatiserede Edman-nedbrydning kan proteiner analyseres ved at anvende dem i opløsning på et TFA-filter og derefter indlæses på Edman-sekventeringsinstrumentet. Proteiner i blandinger adskilles først med 1D-eller 2D-geler og blottes derefter på en PVDF-membran. Proteinerne detekteres ved coomassie blue, amido black eller Poncau s farvning, og proteinerne af interesse skæres ud og PVDF-membranstykket lægges på Edman-sekvensatoren.
med massespektrometri blev udviklet, begyndte brugen af Edman-nedbrydningssekventering at falde. Imidlertid forbliver det stadig metoderne til flere typer applikationer til proteinstrukturel analyse. Det kan bruges til at verificere den N-terminale grænse for rekombinante proteiner eller bestemme N-terminalen for proteaseresistente domæner, især når proteinet eller domænet er >40 til 80 kDa eller ikke let kan renses. Det kan også bruges til at identificere det nye N-terminale og proteolytiske spaltningssted i proteinfragmenterne. Hvad angår nogle nye proteiner og peptider, hvor sekvensdatabaser ikke er tilgængelige til MS/MS-databasesøgning, kan Edman-nedbrydning bruges til analyse.
proteinets N-terminale aminosyre kan spaltes. I processen identificerer den første cyklus således den nøjagtige N-terminale aminosyre. Da de frigivne aminosyrer desuden identificeres og kvantificeres ved kromatografi, kan aminosyrerne med identisk molekylvægt identificeres. For eksempel har isoleucin og leucin en masse på 113 Da, men de har forskellig retentionstid. Desuden kan Edman-sekventering udføres på PVDF-blots fra 1D-og 2D-geler, hvilket muliggør N-terminal sekventering af proteiner i blandingen. Imidlertid vil Edman-nedbrydningssekventering ikke være tilgængelig, når peptidet, hvis N-terminal er blevet kemisk modificeret, såsom acetylering. Og da PITC ikke kan reaktivt med ikke—- – aminosyre, stopper sekventering, hvis der opstår en ikke – – – – – – aminosyre som isoaspartisk syre. Desuden kan større proteiner ikke sekventeres af Edman-sekventeringen.
hos Creative Proteomics leverer vores professionelle proteinsekventeringsplatform N-terminal sekvensanalyse ved Edman-nedbrydning eller massespektrometri (MS) med komplementære fordele til hinanden. Vores team af eksperter med stor erfaring kan hjælpe dig med at forstå, hvad du forsøger at undersøge og opfylde dine krav. Derudover kan vi tilbyde andre proteinidentifikationstjenester, herunder:
- peptid masse fingeraftryk (PMF) service
- sekvensanalyse af peptider eller proteiner
- de novo protein sekvens analyse service
- shotgun protein identifikation service
- membran proteomics service