Biologie für Majors I
Lernergebnisse
- Verstehen Sie den Unterschied zwischen Pre-mRNA und mRNA
Nach der Transkription müssen eukaryotische Prä-mRNAs mehrere Verarbeitungsschritte durchlaufen, bevor sie übersetzt werden können. Eukaryotische (und prokaryotische) tRNAs und rRNAs werden ebenfalls verarbeitet, bevor sie als Komponenten in der Proteinsynthesemaschinerie fungieren können.
mRNA-Verarbeitung
Die eukaryotische Prä-mRNA wird einer umfangreichen Verarbeitung unterzogen, bevor sie übersetzt werden kann. Die zusätzlichen Schritte, die an der eukaryotischen mRNA-Reifung beteiligt sind, erzeugen ein Molekül mit einer viel längeren Halbwertszeit als eine prokaryotische mRNA. Eukaryotische mRNAs dauern mehrere Stunden, während die typische E. coli-mRNA nicht länger als fünf Sekunden dauert.
Pre-mRNAs werden zunächst mit RNA-stabilisierenden Proteinen beschichtet; diese schützen die Pre-mRNA vor dem Abbau, während sie verarbeitet und aus dem Zellkern exportiert wird. Die drei wichtigsten Schritte der Prä-mRNA-Verarbeitung sind die Zugabe von Stabilisierungs- und Signalfaktoren an den 5′- und 3′-Enden des Moleküls und die Entfernung von dazwischenliegenden Sequenzen, die nicht die geeigneten Aminosäuren spezifizieren. In seltenen Fällen kann das mRNA-Transkript nach der Transkription „bearbeitet“ werden.
5′-Capping
Während die Prä-mRNA noch synthetisiert wird, wird dem 5′-Ende des wachsenden Transkripts durch eine Phosphatverknüpfung eine 7-Methylguanosin-Cap hinzugefügt. Diese Einheit (funktionelle Gruppe) schützt die entstehende mRNA vor dem Abbau. Darüber hinaus erkennen Faktoren, die an der Proteinsynthese beteiligt sind, die Cap, um die Translation durch Ribosomen zu initiieren.
3′ Poly-A-Schwanz
Sobald die Elongation abgeschlossen ist, wird die Prä-mRNA durch eine Endonuklease zwischen einer AAUAAA-Konsensussequenz und einer GU-reichen Sequenz gespalten, wobei die AAUAAA-Sequenz auf der Prä-mRNA verbleibt. Ein Enzym namens Poly-A-Polymerase fügt dann eine Reihe von ungefähr 200 A-Resten hinzu, die als Poly-A-Schwanz bezeichnet werden. Diese Modifikation schützt die Prä-mRNA weiter vor dem Abbau und signalisiert den Export der zellulären Faktoren, die das Transkript benötigt, in das Zytoplasma.
Prä-mRNA-Spleißen
Eukaryotische Gene bestehen aus Exons, die proteinkodierenden Sequenzen entsprechen (ex-on bedeutet, dass sie exprimiert werden), und intervenierenden Sequenzen, die Introns genannt werden (Intron bezeichnet ihre intervenierende Rolle), die an der Genregulation beteiligt sein können, aber während der Verarbeitung aus der Prä-mRNA entfernt werden. Intronsequenzen in mRNA kodieren nicht für funktionelle Proteine.
Die Entdeckung von Introns überraschte Forscher in den 1970er Jahren, die erwarteten, dass Prä-mRNAs Proteinsequenzen ohne weitere Verarbeitung spezifizieren würden, wie sie es bei Prokaryoten beobachtet hatten. Die Gene höherer Eukaryoten enthalten sehr oft ein oder mehrere Introns. Diese Regionen können regulatorischen Sequenzen entsprechen; Die biologische Bedeutung vieler Introns oder sehr langer Introns in einem Gen ist jedoch unklar. Es ist möglich, dass Introns die Genexpression verlangsamen, da es länger dauert, Prä-mRNAs mit vielen Introns zu transkribieren. Alternativ können Introns nichtfunktionelle Sequenzreste sein, die von der Fusion alter Gene während der gesamten Evolution übrig geblieben sind. Dies wird durch die Tatsache unterstützt, dass separate Exons häufig separate Proteinuntereinheiten oder Domänen kodieren. Zum größten Teil können die Sequenzen von Introns mutiert werden, ohne letztendlich das Proteinprodukt zu beeinflussen.
Alle Introns einer Prä-mRNA müssen vor der Proteinsynthese vollständig und präzise entfernt werden. Wenn der Prozess auch nur um ein einzelnes Nukleotid fehlschlägt, würde sich der Leserahmen der wieder verbundenen Exons verschieben und das resultierende Protein wäre dysfunktional. Der Vorgang des Entfernens von Introns und des erneuten Verbindens von Exons wird als Spleißen bezeichnet (Abbildung 1). Introns werden entfernt und abgebaut, während sich die Prä-mRNA noch im Zellkern befindet. Das Spleißen erfolgt durch einen sequenzspezifischen Mechanismus, der sicherstellt, dass Introns entfernt und Exons mit der Genauigkeit und Präzision eines einzelnen Nukleotids wieder verbunden werden. Das Spleißen von Prä-mRNAs erfolgt durch Komplexe von Proteinen und RNA-Molekülen, die als Spleißosomen bezeichnet werden.
Übungsfrage
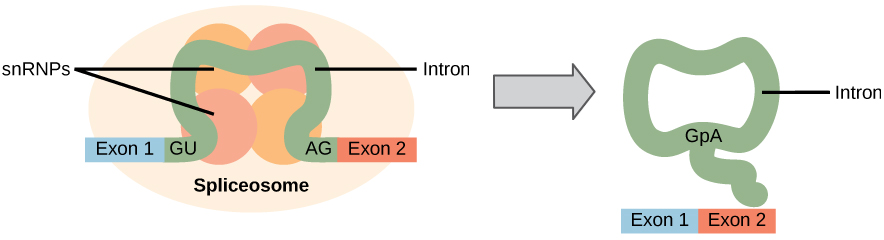
Abbildung 1. Prä-mRNA-Spleißen beinhaltet die präzise Entfernung von Introns aus dem primären RNA-Transkript. Der Spleißprozess wird durch Proteinkomplexe katalysiert, die als Spleißosomen bezeichnet werden und aus Proteinen und RNA-Molekülen bestehen, die als snRNAs bezeichnet werden. Spliceosomen erkennen Sequenzen am 5′- und 3′-Ende des Introns.
Fehler beim Spleißen sind mit Krebs und anderen menschlichen Krankheiten verbunden. Welche Mutationen können zu Spleißfehlern führen?
Beachten Sie, dass mehr als 70 einzelne Introns vorhanden sein können, und jedes muss den Prozess des Spleißens durchlaufen — zusätzlich zum 5′-Capping und der Zugabe eines Poly—A-Schwanzes – nur um ein einzelnes, übersetzbares mRNA-Molekül zu erzeugen.
RNA-Editierung in Trypanosomen
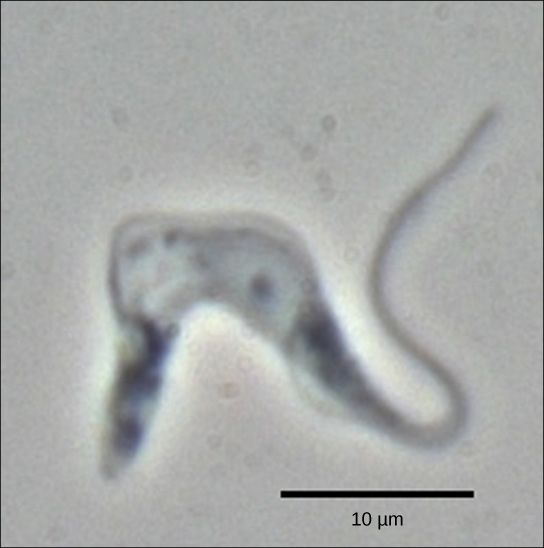
Abbildung 2. Trypanosoma brucei ist der Erreger der Schlafkrankheit beim Menschen. Die mRNAs dieses Pathogens müssen durch Zugabe von Nukleotiden modifiziert werden, bevor eine Proteinsynthese stattfinden kann. (credit: Modifikation der Arbeit von Torsten Ochsenreiter)
Die Trypanosomen sind eine Gruppe von Protozoen, zu denen der Erreger Trypanosoma brucei gehört, der beim Menschen die Schlafkrankheit verursacht (Abbildung 2). Trypanosomen und praktisch alle anderen Eukaryoten haben Organellen, die Mitochondrien genannt werden und die Zelle mit chemischer Energie versorgen. Mitochondrien sind Organellen, die ihre eigene DNA exprimieren und vermutlich die Überreste einer symbiotischen Beziehung zwischen einem Eukaryoten und einem verschlungenen Prokaryoten sind. Die mitochondriale DNA von Trypanosomen weist eine interessante Ausnahme zum zentralen Dogma auf: Ihre Prä-mRNAs haben nicht die richtige Information, um ein funktionelles Protein zu spezifizieren. In der Regel liegt dies daran, dass der mRNA mehrere U-Nukleotide fehlen. Die Zelle führt einen zusätzlichen RNA-Verarbeitungsschritt durch, der als RNA-Editierung bezeichnet wird, um dies zu beheben.
Andere Gene im mitochondrialen Genom kodieren 40- bis 80-Nukleotid-Guide-RNAs. Eines oder mehrere dieser Moleküle interagieren durch komplementäre Basenpaarung mit einigen der Nukleotide im Prä-mRNA-Transkript. Die Führungs-RNA hat jedoch mehr A-Nukleotide als die Prä-mRNA U-Nukleotide zum Binden hat. In diesen Bereichen schlingt die Führungs-RNA aus. Die 3′-Enden von Führungs-RNAs haben einen langen Poly-U-Schwanz, und diese U-Basen werden in Regionen des Prä-mRNA-Transkripts eingefügt, an denen die Führungs-RNAs geschleift sind. Dieser Prozess wird vollständig durch RNA-Moleküle vermittelt. Das heißt, Führer-RNAs — eher als Proteine – dienen als Katalysatoren bei der RNA-Editierung.
RNA-Editierung ist nicht nur ein Phänomen von Trypanosomen. In den Mitochondrien einiger Pflanzen werden fast alle Prä-mRNAs editiert. RNA-Editierung wurde auch bei Säugetieren wie Ratten, Kaninchen und sogar Menschen identifiziert. Was könnte der evolutionäre Grund für diesen zusätzlichen Schritt in der Prä-mRNA-Verarbeitung sein? Eine Möglichkeit ist, dass die Mitochondrien, die Überreste alter Prokaryoten sind, eine ebenso alte RNA-basierte Methode zur Regulierung der Genexpression haben. Zur Unterstützung dieser Hypothese unterscheiden sich die an Prä-mRNAs vorgenommenen Änderungen je nach zellulären Bedingungen. Obwohl spekulativ, kann der Prozess der RNA-Editierung ein Überbleibsel aus einer Urzeit sein, als RNA-Moleküle anstelle von Proteinen für die Katalysierung von Reaktionen verantwortlich waren.
Probieren Sie es aus
Beitragen!
Diese Seite verbessernmehr erfahren