Biologie für Majors II
Lernergebnisse
- Verstehen Sie vergangene und aufkommende Klassifizierungssysteme für Viren
Da sich die meisten Viren wahrscheinlich von verschiedenen Vorfahren entwickelt haben, sind die systematischen Methoden, mit denen Wissenschaftler prokaryotische und eukaryotische Zellen klassifiziert haben, nicht sehr nützlich. Wenn Viren „Überreste“ verschiedener Organismen darstellen, ist selbst eine Genom- oder Proteinanalyse nicht sinnvoll. Warum?, Weil Viren keine gemeinsame genomische Sequenz haben, die sie alle teilen. Zum Beispiel ist die 16S-rRNA-Sequenz, die so nützlich ist, um Prokaryonten-Phylogenien zu konstruieren, für eine Kreatur ohne Ribosomen nutzlos! Biologen haben in der Vergangenheit mehrere Klassifikationssysteme verwendet. Viren wurden zunächst nach gemeinsamer Morphologie gruppiert. Später wurden Gruppen von Viren nach der Art der Nukleinsäure klassifiziert, die sie enthielten, DNA oder RNA, und ob ihre Nukleinsäure einzel- oder doppelsträngig war. Diese früheren Klassifizierungsmethoden gruppierten Viren jedoch unterschiedlich, da sie auf unterschiedlichen Zeichensätzen des Virus basierten. Die heute am häufigsten verwendete Klassifizierungsmethode wird als Baltimore-Klassifizierungsschema bezeichnet und basiert darauf, wie Messenger-RNA (mRNA) in jedem bestimmten Virustyp erzeugt wird.
Frühere Klassifizierungssysteme
Viren enthalten nur wenige Elemente, anhand derer sie klassifiziert werden können: das virale Genom, die Art des Kapsids und die Hüllstruktur für die umhüllten Viren. Alle diese Elemente wurden in der Vergangenheit für die Virusklassifizierung verwendet (Tabelle 1 und Abbildung 1). Virale Genome können in der Art des genetischen Materials (DNA oder RNA) und seiner Organisation (einzel- oder doppelsträngig, linear oder zirkulär und segmentiert oder nicht segmentiert) variieren. Bei einigen Viren sind zusätzliche Proteine, die für die Replikation benötigt werden, direkt mit dem Genom assoziiert oder im viralen Kapsid enthalten.
| Tabelle 1. Virusklassifizierung nach Genomstruktur und Kern | |
|---|---|
| Kernklassifikationen | Beispiele |
| RNA | Tollwutvirus, Retroviren |
| DNA | Herpesviren, Pockenvirus |
| Einzelsträngig | Tollwutvirus, Retroviren |
| Doppelsträngig | Herpesviren, Pockenvirus |
| Linear | Tollwutvirus, Retroviren, Herpesviren, Pockenvirus |
| Rundschreiben | Papillomviren, viele Bakteriophagen |
| Nicht segmentiert: Genom besteht aus einem einzigen Segment genetischen Materials | Parainfluenzaviren |
| Segmentiert: Genom ist in mehrere Segmente unterteilt | Influenzaviren |
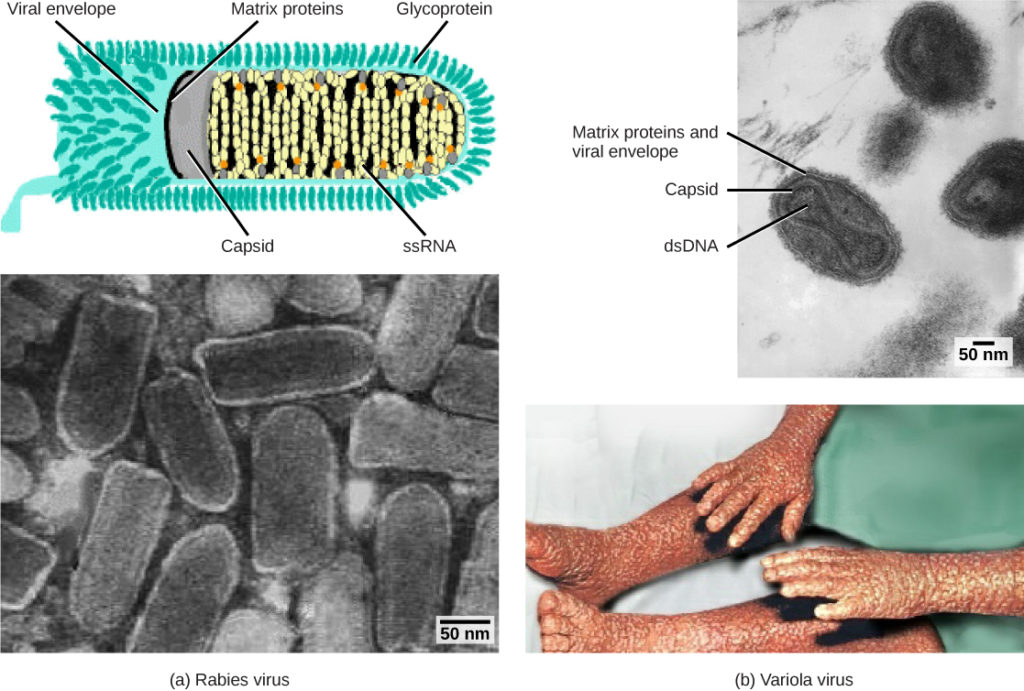
Abbildung 1. Viren werden anhand ihres genetischen Kernmaterials und ihres Kapsid-Designs klassifiziert. (a) Das Tollwutvirus hat einen einzelsträngigen RNA-Kern (ssRNA) und ein umhülltes helikales Kapsid, während (b) das Variola-Virus, der Erreger der Pocken, einen doppelsträngigen DNA-Kern (dsDNA) und ein komplexes Kapsid aufweist. (credit „Tollwut-Diagramm“: Änderung der Arbeit von CDC; „Tollwut-Mikroaufnahme“: Änderung der Arbeit von Dr. Fred Murphy, CDC; credit „Small pox micrograph“: Änderung der Arbeit von Dr. Fred Murphy, Sylvia Whitfield, CDC; credit „Pockenfoto“: Änderung der Arbeit von CDC; scale-bar Daten von Matt Russell)
Viren können auch nach dem Design ihrer Kapside klassifiziert werden (Tabelle 2 und Abbildung 2). Kapside werden als nackte ikosaedrische, umhüllte ikosaedrische, umhüllte helikale, nackte helikale und komplexe klassifiziert. Die Art des genetischen Materials (DNA oder RNA) und seine Struktur (einzel- oder doppelsträngig, linear oder kreisförmig und segmentiert oder nicht segmentiert) werden zur Klassifizierung der Viruskernstrukturen verwendet (Tabelle 2).
| Tabelle 2. Virusklassifizierung nach Kapsid-Struktur | |
|---|---|
| Kapsid-Klassifikation | Beispiele |
| Nacktes Ikosaeder | Hepatitis-A-Virus, Polioviren |
| Umhülltes ikosaedrisches | Epstein-Barr-Virus, Herpes-simplex-Virus, Rötelnvirus, Gelbfiebervirus, HIV-1 |
| Umhüllte helikale | Influenzaviren, Mumpsvirus, Masernvirus, Tollwutvirus |
| Nacktes helikales | Tabakmosaikvirus |
| Komplex mit vielen Proteinen; einige haben Kombinationen von ikosaedrischen und helikalen Kapsid-Strukturen | Herpesviren, Pockenvirus, Hepatitis-B-Virus, T4-Bakteriophagen |

Abbildung 2. Transmissionselektronenmikroskopische Aufnahmen verschiedener Viren zeigen ihre Strukturen. Das Kapsid des (a) Poliovirus ist nackt ikosaedrisch; (b) das Epstein-Barr-Virus-Kapsid ist ikosaedrisch umhüllt; (c) das Mumps-Virus-Kapsid ist eine umhüllte Helix; (d) das Tabakmosaik-Virus-Kapsid ist nackt helikal; und (e) das Herpesvirus-Kapsid ist komplex. (kredit a: Änderung der Arbeit von Dr. Fred Murphy, Sylvia Whitfield; Kredit b: Änderung der Arbeit von Liza Gross; Kredit c: Änderung der Arbeit von Dr. FA Murphy, CDC; Kredit d: Änderung der Arbeit von USDA ARS; Kredit e: modifikation der Arbeit von Linda Stannard, Abteilung für medizinische Mikrobiologie, Universität Kapstadt, Südafrika, NASA; Scale-Bar-Daten von Matt Russell)
Baltimore-Klassifikation
Das am häufigsten verwendete System der Virusklassifizierung wurde Anfang der 1970er Jahre vom Nobelpreisträger David Baltimore entwickelt. Zusätzlich zu den oben erwähnten Unterschieden in Morphologie und Genetik gruppiert das Baltimore-Klassifikationsschema Viren danach, wie die mRNA während des Replikationszyklus des Virus produziert wird.
Viren der Gruppe I enthalten doppelsträngige DNA (dsDNA) als Genom. Ihre mRNA wird durch Transkription ähnlich wie bei zellulärer DNA hergestellt.
Viren der Gruppe II haben einzelsträngige DNA (ssDNA) als Genom. Sie wandeln ihre einzelsträngigen Genome in ein dsDNA-Intermediat um, bevor eine Transkription zu mRNA erfolgen kann.
Viren der Gruppe III verwenden dsRNA als ihr Genom. Die Stränge trennen sich, und einer von ihnen wird als Vorlage für die Erzeugung von mRNA unter Verwendung der vom Virus codierten RNA-abhängigen RNA-Polymerase verwendet.
Viren der Gruppe IV haben ssRNA als ihr Genom mit einer positiven Polarität. Positive Polarität bedeutet, dass die genomische RNA direkt als mRNA dienen kann. Zwischenprodukte von dsRNA, sogenannte replikative Zwischenprodukte, werden beim Kopieren der genomischen RNA hergestellt. Aus diesen Zwischenprodukten werden mehrere RNA-Stränge voller Länge negativer Polarität (komplementär zur positivsträngigen genomischen RNA) gebildet, die dann als Matrizen für die Herstellung von RNA mit positiver Polarität dienen können, einschließlich sowohl genomischer RNA voller Länge als auch kürzerer viraler mRNAs.
Viren der Gruppe V enthalten ssRNA-Genome mit negativer Polarität, was bedeutet, dass ihre Sequenz komplementär zur mRNA ist. Wie bei Viren der Gruppe IV werden dsRNA-Zwischenprodukte verwendet, um Kopien des Genoms herzustellen und mRNA zu produzieren. In diesem Fall kann das negativsträngige Genom direkt in mRNA umgewandelt werden. Zusätzlich werden positive RNA-Stränge in voller Länge hergestellt, um als Schablonen für die Produktion des negativ-gesträngten Genoms zu dienen.
Viren der Gruppe VI haben diploide (zwei Kopien) ssRNA-Genome, die unter Verwendung des Enzyms Reverse Transkriptase in dsDNA umgewandelt werden müssen; die dsDNA wird dann zum Kern der Wirtszelle transportiert und in das Wirtsgenom eingefügt. Dann kann mRNA durch Transkription der viralen DNA hergestellt werden, die in das Wirtsgenom integriert wurde.
Viren der Gruppe VII haben partielle dsDNA-Genome und bilden ssRNA-Zwischenprodukte, die als mRNA fungieren, aber auch durch reverse Transkriptase, die für die Genomreplikation notwendig ist, wieder in dsDNA-Genome umgewandelt werden. Die Merkmale jeder Gruppe in der Baltimore-Klassifikation sind in Tabelle 3 mit Beispielen für jede Gruppe zusammengefasst.
| Tabelle 3. Baltimore Klassifikation | |||
|---|---|---|---|
| Gruppe | Charakteristik | Art der mRNA-Produktion | Beispiel |
| I | Doppelsträngige DNA | mRNA wird direkt von der DNA-Vorlage transkribiert | Herpes simplex (Herpesvirus) |
| II | Einzelsträngige DNA | DNA wird in doppelsträngige Form umgewandelt, bevor RNA transkribiert wird | Canines Parvovirus (Parvovirus) |
| III | Doppelsträngige RNA | mRNA wird transkribiert von das RNA-Genom | Gastroenteritis im Kindesalter (Rotavirus) |
| IV | Einzelsträngige RNA (+) | Genomfunktionen als mRNA | Erkältung (Pircornavirus) |
| V | Einzelsträngige RNA (-) | mRNA wird aus dem RNA-Genom transkribiert | Tollwut (Rhabdovirus) |
| VI | Einzelsträngige RNA-Viren mit Reverse Transkriptase | Reverse Transkriptase macht DNA aus dem RNA-Genom; DNA wird dann in das Wirtsgenom eingebaut; mRNA wird aus der eingebauten DNA transkribiert | Human Immunodeficiency Virus (HIV) |
| VII | Doppelsträngige DNA-Viren mit reverser Transkriptase | Das virale Genom ist doppelsträngige DNA, aber virale DNA wird durch ein RNA-Zwischenprodukt repliziert; Die RNA kann direkt als mRNA oder als Vorlage zur Herstellung von mRNA dienen | Hepatitis-B-Virus (Hepadnavirus) |