Creative Proteomics Blog
Die Sequenz von Aminosäuren in einem Protein oder Peptid kann durch den von Pehr Edman entwickelten Edman-Abbau identifiziert werden. Diese Methode kann das Peptid vom N-Terminal markieren und spalten, ohne die Peptidbindungen zwischen anderen Aminosäureresten zu unterbrechen. Die Edman-Abbaureaktion wurde 1967 von Edman und Beggs automatisiert. Heutzutage ist der automatisierte Edman-Abbau (der Proteinsequenator) weit verbreitet und kann Peptide mit bis zu 50 Aminosäuren sequenzieren.
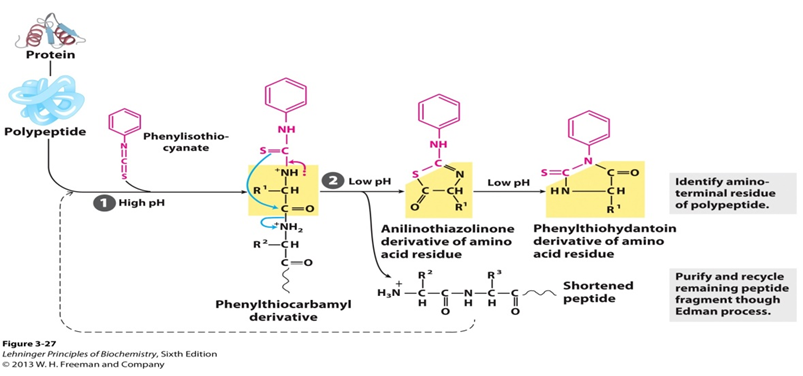
Zyklischer Abbau von Peptiden basierend auf der Reaktion von Phenylisothiocyanat mit der freien Aminogruppe des N-terminalen Restes, so dass Aminosäuren nacheinander entfernt und als ihre Phenylthiohydantoinderivate identifiziert werden. Nach dem spezifischen Verfahren wird ein ungeladenes Peptid mit Phenylisothiocyanat (PITC) am Aminoterminus unter schwach alkalischen Bedingungen zu einem Phenylthiocarbamoylderivat (PTC-Peptid) umgesetzt. Dann greift der Thiocarbonylschwefel des Derivats unter sauren Bedingungen den Carbonylkohlenstoff der N-terminalen Aminosäure an. Die erste Aminosäure wird als Anilinothiazolinonderivat (ATZ-Aminosäure) gespalten und der Rest des Peptids kann isoliert und dem nächsten Abbauzyklus unterworfen werden. Einmal gebildet, ist dieses Thiazolonderivat stabiler als Phenylthiocarbamylderivat. Die ATZ-Aminosäure wird dann durch Extraktion mit Ethylacerat entfernt und in ein Phenylthiohydantoinderivat (PTH-Aminosäure) überführt. Und die Chromatographie kann verwendet werden, um den PTH-Rückstand zu identifizieren, der von jedem Zyklus erzeugt wird.
Hinsichtlich des automatisierten Edman-Abbaus können Proteine analysiert werden, indem sie in Lösung auf einen TFA-Filter aufgetragen und dann auf das Edman-Sequenzierungsinstrument geladen werden. Proteine in Mischungen werden zuerst durch 1D- oder 2D-Gele getrennt und dann auf eine PVDF-Membran geblottet. Die Proteine werden durch Coomassie-Blau-, Amido-Schwarz- oder Poncau-S-Färbung nachgewiesen und die interessierenden Proteine ausgeschnitten und das PVDF-Membranstück auf den Edman-Sequenzer geladen.
Mit Massenspektrometrie entwickelt wurde, begann die Verwendung von Edman-Abbau-Sequenzierung zu verringern. Es bleibt jedoch die Methode für verschiedene Arten von Proteinstrukturanalyseanwendungen. Es kann verwendet werden, um die N-terminale Grenze von rekombinanten Proteinen zu verifizieren oder den N-Terminus von Protease-resistenten Domänen zu bestimmen, insbesondere wenn das Protein oder die Domäne > 40 bis 80 kDa beträgt oder nicht ohne weiteres gereinigt werden kann. Es kann auch verwendet werden, um die neue N-terminale und proteolytische Schnittstelle in den Proteinfragmenten zu identifizieren. Darüber hinaus kann bei einigen neuartigen Proteinen und Peptiden, bei denen Sequenzdatenbanken nicht für die MS / MS-Datenbanksuche verfügbar sind, der Edman-Abbau zur Analyse verwendet werden.
Die N-terminale Aminosäure des Proteins kann abgespalten werden. Somit identifiziert der erste Zyklus im Prozess die genaue N-terminale Aminosäure. Da außerdem die freigesetzten Aminosäuren durch Chromatographie identifiziert und quantifiziert werden, können die Aminosäuren mit identischem Molekulargewicht identifiziert werden. Zum Beispiel haben Isoleucin und Leucin eine Masse von 113 Da, aber sie haben unterschiedliche Retentionszeiten. Darüber hinaus kann die Edman-Sequenzierung an PVDF-Blots aus 1D- und 2D-Gelen durchgeführt werden, was eine N-terminale Sequenzierung von Proteinen in der Mischung ermöglicht. Die Edman-Abbausequenzierung ist jedoch nicht verfügbar, wenn das Peptid, dessen N-Terminus chemisch modifiziert wurde, wie Acetylierung. Und da der PITC nicht mit Nicht-α-Aminosäure reagieren kann, stoppt die Sequenzierung, wenn eine Nicht-α-Aminosäure wie Isoasparaginsäure angetroffen wird. Darüber hinaus können größere Proteine nicht durch die Edman-Sequenzierung sequenziert werden.
Bei Creative Proteomics bietet unsere professionelle Proteinsequenzierungsplattform eine N-terminale Sequenzanalyse durch Edman-Abbau oder Massenspektrometrie (MS) mit komplementären Vorteilen. Unser Expertenteam mit langjähriger Erfahrung kann Ihnen helfen zu verstehen, was Sie untersuchen und Ihre Anforderungen erfüllen möchten. Darüber hinaus können wir andere Proteinidentifikationsdienste anbieten, darunter:
- Peptide Mass Fingerprinting (PMF) Service
- Sequenzanalyse von Peptiden oder Proteinen
- De novo Protein sequence analysis service
- Shotgun Protein Identification service
- Membrane proteomics Service