Frontiers in Microbiology
Introduction
Die Familie der Astroviridae enthält einzelsträngige RNA-Viren mit positivem Sinn innerhalb von zwei Gattungen, Mamastrovirus und Avastrovirus, die Säugetiere bzw. Vögel infizieren. Derzeit erkennt das Internationale Komitee für Taxonomie von Viren (Internationales Komitee für Taxonomie von Viren , 2018) 19 Arten, nämlich Mamastrovirus-1 bis -19, innerhalb der Gattung Mamastrovirus; Es gibt jedoch zahlreiche Stämme, die auf eine Klassifizierung warten, von denen einige vorläufig als neue Arten gelten (Donato und Vijaykrishna, 2017).
Seit 2010 wurden mehrere Astroviren zunehmend als neuroinvasiv bei verschiedenen Säugetierarten, einschließlich Menschen, erkannt (Quan et al., 2010; Naccache et al., 2015), mink (Blomström et al., 2010), Rinder (Li et al., 2013), Schafe (Pfaff et al., 2017) und Schweine (Boros et al., 2017). Nach erstmaliger Erkennung einer bovinen Astrovirus-assoziierten Enzephalitis bei Rindern in den Vereinigten Staaten (Li et al., 2013) ergab eine retrospektive Studie bei sporadischer Rinderenzephalitis unbestimmter Ätiologie aus der Schweiz, dass dieses neuroinvasive Astrovirus jahrzehntelang unentdeckt geblieben war (Selimovic-Hamza et al., 2016). Obwohl die Epidemiologie und die Übertragungswege dieser Astroviren unbekannt sind, wurde eine artübergreifende Übertragung aufgrund des hohen Identitätsniveaus (> 98%) vorgeschlagen, das zwischen neuroinvasiven Astroviren von Rindern und Schafen auf Nukleotid- und Aminosäurespiegel (Boujon et al., 2017).
Bovine Astroviren (BoAstVs), genannt BoAstV-11 (Li et al., 2013) und BoAstV-CH13 (Bouzalas et al., 2014), wurden zunächst im Gehirn von Rindern mit nicht-suppurativer Enzephalitis in den USA bzw. in der Schweiz gefunden. Trotz der unterschiedlichen Nomenklatur repräsentieren beide Viren den gleichen Genotyp (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a), die noch auf die offizielle Klassifizierung durch den ICTV wartet. Im Jahr 2015 wurde ein bisher unbekannter BoAstV-Stamm namens BoAstV-CH15 im Gehirn von Kühen mit Enzephalitis in der Schweiz identifiziert. Der vollständige genomphylogenetische Vergleich ergab eine engere Beziehung von BoAstV-CH15 zu einem Schafastrovirus (OvAstV) als zu BoAstV-CH13 (Seuberlich et al., 2016). Eine Koinfektion mit BoAstV-CH13 und BoAstV-CH15 wurde ebenfalls in einem Fall dokumentiert (Seuberlich et al., 2016). Im selben Jahr in Deutschland, Schlottau et al. (2016) berichteten über ein neuartiges Astrovirus, nämlich BoAstV-BH89 / 14, bei einer Kuh mit Enzephalitis, das am engsten mit OvAstV und BoAstV-CH15 verwandt war. Anschließend wurde BoAstV-CH13 / 11 im Jahr 2017 in Fällen von Rinderenzephalitis in Ost- und Westkanada identifiziert (Spinato et al., 2017; Selimovic-Hamza et al., 2017b). Im Jahr 2018 wurde bei einem Patienten mit nicht-suppurativer Enzephalomyelitis in Japan ein neuartiges neuroinvasives BoAstV identifiziert, das eng mit dem nordamerikanischen und europäischen BoAstV-CH1 / BoAstV-CH13 verwandt ist, und das Auftreten einer intra-genotypischen Rekombination zwischen den nordamerikanischen und europäischen Stämmen wurde vorgeschlagen (Hirashima et al., 2018).
Während Fälle von Astrovirus-assoziierter Enzephalitis in Nordamerika, Europa und Asien berichtet wurden, wurde ihre Anwesenheit in der südlichen Hemisphäre nie dokumentiert. Hier beschreiben wir einen Fall von Astrovirus-assoziierter Enzephalitis bei Rindern in Uruguay, Dies erweitert die geografische Verbreitung und genetische Vielfalt neuroinvasiver Astroviren und liefert phylogeographische Beweise dafür, dass dieses Virus aus Europa nach Amerika eingeführt und später nach Asien verbreitet wurde.
Materialien und Methoden
Geschichte und Signalgebung
Im Juni 2018 entwickelte ein 22 Monate alter Holsteiner Ochse in einer Gruppe von 37 Ochsen auf einer ∼300 Hektar großen Farm in Colonia, Uruguay, progressive neurologische Symptome, darunter ungewöhnliches Verhalten, zielloses Gehen, Kreisen, Ataxie, sich wiederholende und unkoordinierte Zungenbewegungen und Liegeposition. Die Herde graste auf einer jährlichen Haferweide und wurde mit Maissilage ergänzt. Eine vermutliche klinische Diagnose einer zerebralen Listeriose durch den Tierarzt veranlasste die Behandlung mit Penicillin und Streptomycin, das Tier starb jedoch nach einem 3-tägigen klinischen Verlauf spontan.
Pathologische Untersuchung, In-situ-Hybridisierung (ISH) und Immunhistochemie (IHC)
Der Kopf des Ochsen wurde aus dem Schlachtkörper entfernt und dem Veterinärdiagnostiklabor der INIA (Animal Health Platform) zur diagnostischen Aufarbeitung vorgelegt. Die Hälfte des Gehirns, ein kurzes Segment des proximalen zervikalen Rückenmarks (C1), des Trigeminusganglions und der Wurzel des Trigeminusnervs, der Speicheldrüse, des retropharyngealen Lymphknotens, des Oropharynx, der Speiseröhre, der Zunge und des Skelettmuskels wurden 48-72 h lang in 10% neutralem gepuffertem Formalin immersionsfixiert. Gewebe wurden routinemäßig für die Histologie verarbeitet, in Paraffin eingebettet, bei 4-5 µm mikrotomiert und mit Hämatoxylin und Eosin (H & E) und Gram Flecken.
Chromogene ISH wurde manuell an 5 µm-Schnitten von formalinfixiertem, in Paraffin eingebettetem (FFPE) Hirnstamm, Großhirn und Kleinhirn auf Superfrost Plus-Objektträgern (Thermo Fisher Scientific, Pittsburgh, PA, USA) unter Verwendung des RNAscope 2.5 Red Assay Kit (Cat #322360, Advanced Cell Diagnostics, Hayward, CA, USA) und der BoAstV-Sonde Cat durchgeführt. #406921. Die Sonde besteht aus 20 DNA-Paaren, die auf die Region 5232-6180 des Virus abzielen (GenBank KF233994.1). Jeder 5 µm Gewebeabschnitt wurde vor der Sondenhybridisierung für 2 h bei 40 °C mit Wärme und Protease vorbehandelt und gemäß den Empfehlungen der Hersteller verarbeitet. Negativkontrollen, die zur Validierung des Signals verwendet wurden, umfassten eine nicht verwandte (GC-content matched) Sonde, die an seriellen Schnitten durchgeführt wurde und Gewebe von nicht infizierten Tieren untersuchte. Objektträger wurden mit Hämatoxylin gegengefärbt und mit EcoMount (Biocare Medical, Concord, CA, USA) montiert.
Zusätzlich wurde IHC in FFPE-Abschnitten von Hirnstamm, Großhirn und Kleinhirn durchgeführt, wie zuvor beschrieben, zur Identifizierung des West-Nil-Virus (WNV, Flavivirus) (Palmieri et al., 2011), Tollwutvirus (Lyssavirus) (Stein et al., 2010) und Chlamydia spp. (Giannitti et al., 2016) Antigene.
Molekulare Virologie
Die Nukleinsäureextraktion wurde aus einer gepoolten Probe von gefrorenem (-20 ° C) Gehirn unter Verwendung des MagMAX Nucleic Acid Isolation Kit® (Thermo Fisher Scientific) durchgeführt. Für den Astrovirus-Nachweis wurde die reverse Transkription (RT) mit RevertAid Reverse Transkriptase® (Thermo Fisher Scientific) und Random Hexamer Primern (Qiagen) durchgeführt. Die PCR wurde aus cDNA unter Verwendung von MangoMix® (Bioline) und Primern durchgeführt, die ein 432-Nukleotidfragment des Astrovirus-Polymerase-Gens amplifizieren (Tse et al., 2011). Das PCR-Produkt wurde in 2% igem Agarosegel visualisiert, mit PureLink® Quick Gel Extraction und PCR Purification Combo Kit (Invitrogen) gereinigt und bei Macrogen Inc. sequenziert. (Seoul, Südkorea). Für die Astrovirus-Ganzgenomamplifikation, Maxima H Minus Reverse Transkriptase (Thermo Fisher Scientific) und oligo(dT)18 zur Gewinnung von cDNA und MangoMix® (Bioline) oder Ranger DNA Polymerase (Bioline) mit Primern, die von Hirashima et al., 2018, verwendet wurden. Die PCR-Produkte wurden in 1-2%igem Agarosegel visualisiert, gereinigt und wie oben erwähnt sequenziert. Die Sequenzmontage wurde mit SeqMan (Lasergene 8, DNASTAR) durchgeführt. Sechsundzwanzig vollständige Genomsequenzen des neuroinvasiven Astrovirus von Rindern, Schafen, Schweinen, Menschen und Nerzen sowie des enterischen bovinen Astrovirus, die in der GenBank verfügbar sind, wurden mit der Clustal W in MEGA 7-Software heruntergeladen und ausgerichtet (Kumar et al., 2016). W-IQ-BAUM1 (Trifinopoulos et al., 2016) wurde verwendet, um das Best-Fit-Modell der Sequenzevolution (SYM + I + G4) zu bestimmen und einen phylogenetischen Baum mit maximaler Wahrscheinlichkeit mit den fast vollständigen Sequenzen des in diesem Fall nachgewiesenen BoAstV und den vollständigen Sequenzen, die von der GenBank heruntergeladen wurden, zu konstruieren Bootstrap als statistische Methode zur Bewertung der Robustheit von Kladen. Ähnlichkeitsdiagramm wurde mit SimPlot-Software durchgeführt (Lole et al., 1999). P-Distanzen auf Aminosäurespiegel des ORF2 wurden mit MEGA 7 Software abgeschätzt (Kumar et al., 2016).
Zusätzlich wurde eine Bayes-phylogeographische Analyse mit dem BEAST v1.8.4-Paket durchgeführt (Drummond et al., 2012), mit: die vollständige kodierende Region der BoAstV CH13 / NeuroS1-Linie, ORF1ab (nicht-strukturelle Gene), ORF2 (strukturelles Gen), ORF1a (Protease) und eine partielle ORF1b (Polymerase-genomische Region, für die kanadische Stämme verfügbar waren) mit allen in GenBank verfügbaren Sequenzen (letzter Zugriff April 18, 2019), um die Evolutionsrate, das Alter / die Jahre der gemeinsamen Vorfahren und den wahrscheinlichsten Weg der Viruszirkulation nach Ländern (Schweiz, Uruguay, USA, Kanada und Japan) zu bestimmen. Der Mangel an Rekombination im Datensatz wurde unter Verwendung des Rekombinationserkennungsprogramms 4 bestimmt. Das Substitutionsmodell, das am besten zu jeder Ausrichtung passt, wurde mithilfe der MEGA 7-Software anhand von Bayesian Information Criterion (BIC) -Werten bestimmt, und die zeitliche Struktur jedes Datensatzes wurde mithilfe von TempEst (Rambaut et al., 2016). Die lognormal relaxed molecular Clock mit Bayes’scher Strukturanalyse wurde von Bayes Factor unter den verschiedenen Kombinationen von molekularen Uhren und koaleszierenden Baumprioren ausgewählt. Das Land der Entdeckung wurde als Merkmal verwendet. Die Markov-Kette Monte Carlo Länge betrug 100 Millionen Generationen, die Konvergenz der Analyse zu gewährleisten, in Tracer v1.6 ausgewertet.0, und die posteriore Wahrscheinlichkeit wurde verwendet, um Clades zu bewerten. Der maximale Clade Credibility Tree (MCCT) wurde mit der TreeAnnotator-Software von BEAST ermittelt und in FigTree v1.4.3 visualisiert.
Schließlich wurde DNA, die aus gefrorenem Gehirn extrahiert wurde, durch PCR zum Nachweis der bovinen Herpesviren 1 und 5 (BHV-1 und -5) verarbeitet, wie zuvor beschrieben (Ashbaugh et al., 1997).
Bakteriologie
Frische Proben von Großhirn und Hirnstamm wurden routinemäßig für aerobe Bakterienkulturen in Blut und MacConkey-Agaren und selektive Kultur für Listeria monocytogenes (Al-Zoreky und Sandine, 1990) verarbeitet.
Ergebnisse und Diskussion
Die klinischen Symptome und epidemiologischen Befunde in dem hier beschriebenen Fall waren, wenn auch unspezifisch, ähnlich denen, die in anderen Fällen einer bovinen Astrovirus-assoziierten Enzephalitis beschrieben wurden, die normalerweise als sporadisch beschrieben wird (Selimovic-Hamza et al., 2016), mit einer Vielzahl von neurologischen Defiziten (Deiss et al., 2017), mit einer Dauer der klinischen Symptome, die typischerweise von 1 Tag bis 3 Wochen reicht (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
Die makroskopische Untersuchung des Gehirns, des C1-Segments des Rückenmarks und anderer Gewebe des Kopfes ergab keine signifikanten groben anatomischen Läsionen. Histologisch gab es eine mittelschwere bis schwere lymphozytäre, histiozytäre und plasmazytäre Meningoenzephalomyelitis, die das Telencephalon (einschließlich der Gehirnhälfte und des Hippocampus), den Hirnstamm und das einzige untersuchte Segment des Rückenmarks betraf. Die Läsionen waren überwiegend in der grauen Substanz und den begrenzenden Bereichen der weißen Substanz verteilt. In den betroffenen Gebieten gab es perivaskuläre Cuffing und lymphoplasmazytische und histiozytische Entzündung und neuronale Nekrose / Neuronophagie mit Gliose in der angrenzenden neuropil. Es gab eine Satellitose betroffener, nekrotischer Neuronen (Abbildungen 1A–D). Die Läsionen waren im Kleinhirnparenchym viel seltener und schwerer, obwohl eine multifokale moderate Kleinhirn-Leptomeningitis auftrat. Es wurden keine intraläsionalen Bakterien mit H & E- und Gram-Flecken gefunden. In den anderen untersuchten Geweben wurden keine signifikanten histologischen Veränderungen festgestellt.

Abbildung 1. Histologische Läsionen im Hirnstamm (A,B) und der Großhirnrinde (C,D) und Nachweis von BoAstV-RNA in der Großhirnrinde (E, G). Bilder (A–D) sind Abschnitte des Gehirns, die mit H & E gefärbt sind; die Bilder E und G sind Abschnitte der Großhirnrinde, die die Hybridisierung unter Verwendung von chromogenem ISH unter Verwendung der BoAstV-spezifischen Sonde demonstrieren, mit Hämatoxylin gegengefärbt; Bild F ist ein serieller Abschnitt der DAPB-Sonde der Großhirnrinde (Negativkontrolle), mit Hämatoxylin gegengefärbt. (A) Ein perivenulärer Raum wird durch Entzündungszellen (meist Lymphozyten und Histiozyten), die auch das angrenzende Neuropil infiltrieren, deutlich erweitert. (B) Das Neuron in der Mitte weist hypereosinophiles Perikaryon und Karyorrhexis (Nekrose) auf, und der neuronale Körper ist von einer erhöhten Anzahl von Gliazellen (Satellitose) und Entzündungszellen umgeben. (C) Die Leptomeninge wird von Lymphozyten und Histiozyten infiltriert. (D) Eine Region der Großhirnrinde mit mehreren hypereosinophilen (nekrotischen) Neuronen und einem großen Gefäß mit perivaskulären lymphozytären Manschetten. (E) In einem seriellen Abschnitt der Großhirnrinde wird die reichlich vorhandene intrazytoplasmatische BoAstV-RNA-Markierung durch starke, körnige rote Chromogenablagerung innerhalb des neuronalen Zytoplasmas des Somas und der neuronalen Erweiterungen (E, G) dargestellt, die bei Hybridisierung unter Verwendung der negativen Kontrollsonde (F) nicht vorhanden ist.
Bei histologischer Untersuchung des Zentralnervensystems wurde eine neuroinvasive Virusinfektion vermutet. Rinder mit Enzephalitis sind besorgniserregend, da viele Neuropathogene von Wiederkäuern zoonotisch sind (Cantile und Youssef, 2016); daher sollte eine Diagnose einer Enzephalitis umfangreiche Labortests veranlassen, um nach Möglichkeit nach Infektionserregern zu suchen. In dem hier beschriebenen Fall werden IHCs für WNV, Tollwutvirus und Chlamydia spp., und PCR für BHV-1 und -5 waren alle negativ, und keine pathogenen Bakterien wurden vom Gehirngewebe kultiviert. Da das Tier < 2 Jahre alt war und keine spongiformen Veränderungen im Hirnstamm beobachtet wurden, wurde das Tier nicht auf bovine spongiforme Enzephalopathie (BSE) getestet, eine exotische Krankheit ausgewachsener Rinder, über die in Uruguay noch nie berichtet wurde. Darüber hinaus ist BSE nicht entzündlich (Cantile und Youssef, 2016).
Die In-situ–Hybridisierung wurde unter Verwendung einer aus BoAstV-NeuroS1 erzeugten Sonde durchgeführt, und es gab eine Sondenhybridisierung, die innerhalb des Zytoplasmas von Neuronen in der Gehirnhälfte und im Hippocampus reichlich vorhanden und auf diese beschränkt war (Abbildungen 1E-G). In diesen Bereichen kolokalisierte die Sondenhybridisierung mit nekrotischen Neuronen und Regionen der Gliose, wobei keine Sondenhybridisierung in den Gliazellen oder Entzündungszellen des perivaskulären Systems nachweisbar war. Im Kleinhirn, das nur minimale entzündliche Läsionen im Parenchym, aber mäßige Leptomeningitis aufwies, oder im Hirnstamm, einschließlich Abschnitten mit schwerer Entzündung, wurde keine virale Nukleinsäure durch ISH nachgewiesen. Dies bedeutet, dass der topographische Nachweis der Virusverteilung durch ISH in den untersuchten Abschnitten eingeschränkter war als die Enzephalitis, die gelegentlich bei BoAstV-CH13 / CH1-assoziierter Enzephalitis bei Rindern beschrieben wurde (Selimovic-Hamza et al., 2017a,b). Ein Grund für diesen gelegentlichen Mangel an viralem RNA-Nachweis in geschädigten Hirnarealen könnte die Nachweisgrenze des ISH oder die Clearance des Virus in entzündeten Hirnarealen zum Zeitpunkt des Todes sein, wie zuvor vorgeschlagen (Selimovic-Hamza et al., 2017b). Wie erwartet wurde keine Sondenhybridisierung durch ISH in dem als Negativkontrolle verwendeten Hirngewebe nachgewiesen.
Astrovirus wurde im Gehirn mittels RT-PCR nachgewiesen. Die nahezu vollständige Genomsequenzanalyse ergab einen Mamastrovirus-Stamm innerhalb der CH13 / NeuroS1-Klasse, den wir BoAstV-Neuro-Uy nannten, die Sequenz wurde in der GenBank unter der Zugangsnummer MK386569 hinterlegt. Die phylogenetische Analyse ergab die Nähe zu anderen neuroinvasiven Astroviren innerhalb der Virginia / Human-Mink-Schaf (VA / HMO) -Klade (Abbildung 2), die die meisten bekannten neuroinvasiven Astroviren enthält (Hirashima et al., 2018; Reuter et al., 2018). Die fast vollständige Sequenz von BoAstV-Neuro-Uy ist 6427 bp lang und hat eine Sequenzidentität von 94% mit KagoshimaSR28-462 Stamm. BoAstV-Neuro-Uy hat ähnliche Eigenschaften wie andere Stämme der Linie CH13 / NeuroS1: eine 5’UTR-Region von 51 nt, ORF1a (Protease) von 861 Aminosäuren (aa), ORF1b von 523 aa (RNA-abhängige RNA-Polymerase) und ORF2 von 758 aa (Kapsidprotein). Leider konnte die 3’UTR nicht sequenziert werden, aber es wird angenommen, dass ein Poly (A) -Schwanz vorhanden ist, da oligo (dT) 18 verwendet wurde, um cDNA zu erhalten. Zusätzlich ist die heptamere AAAAAAC-Sequenz, ein ribosomales Frameshift-Signal, vorhanden. P-Abstände auf Aminosäurespiegel des ORF2 bestätigten die Zuordnung dieses Stammes zur CH13/CH1-Klasse. P-Abstände < 0.35 zwischen BoAstV-Neuro-Uy und anderen Mitgliedern dieser Klasse (Tabelle 1) würde eine Klassifizierung dieser Virusstämme innerhalb derselben Spezies unterstützen; Mamastrovirus-13 wurde kürzlich von anderen Autoren vorgeschlagen (Donato und Vijaykrishna, 2017; Hirashima et al., 2018), obwohl eine definitive Artenzuweisung durch das ICTV noch aussteht. Die für die ISH verwendete Sonde, die aus BoAstV-NeuroS1 erzeugt wurde, hatte eine Sequenzidentität von 92,7% mit BoAstV-Neuro-Uy.
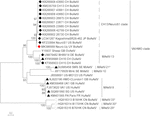
Abbildung 2. Phylogenetische Analyse von Nukleotidsequenzen voller Länge nach der Maximum-Likelihood-Methode. BoAstV-Neuro-Uy ist mit einer roten Raute markiert. Andere Sequenzen von bovinen neuroinvasiven Astroviren sind mit schwarzen Rauten markiert, Sequenzen von nicht-bovinen neuroinvasiven Astroviren sind mit schwarzen Dreiecken markiert, und Sequenzen von enterischen bovinen Astroviren sind mit weißen Kreisen markiert. Mamastrovirus-Arten werden gezeigt; Sternchen weisen auf Arten hin, die vom ICTV noch nicht erkannt wurden. Stämme innerhalb der CH13 / CH1-Klasse, denen vom ICTV noch keine Art zugewiesen wurde, sind mit einer gestrichelten Linie gekennzeichnet. Bootstrap-Werte werden in Knoten angegeben. CH, Schweiz; JP, Japan; US, Vereinigte Staaten von Amerika; UY, Uruguay; GB, Vereinigtes Königreich von Großbritannien; DE, Deutschland; SE, Schweden; FR, Frankreich; CN, China. BoAstV, Rinder-Astrovirus; OvAstV, Schaf-Astrovirus; PoAstV, Schweine-Astrovirus; HuAstV, menschliches Astrovirus; MiAstV, Nerz-Astrovirus.
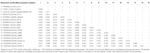
Tabelle 1. Schätzungen der evolutionären Divergenz auf Aminosäurespiegel der gesamten ORF2-Region zwischen Sequenzen von Mamastrovirus-13 von Rindern und Schafen und Stämmen innerhalb der CH13 / CH1-Klasse, die vom Internationalen Komitee für Taxonomie noch nicht einer Art zugeordnet wurden von Viren (ICTV).
Studien, die auf neuropathologischen Untersuchungen und dem Nachweis von Astrovirus-Nukleinsäuren und -proteinen basieren, haben ergeben, dass ein wahrscheinlicher Kausalzusammenhang zwischen Astrovirus-Infektion und neurologischen Erkrankungen und Läsionen bei Rindern besteht (Selimovic-Hamza et al., 2017a; Reuter et al., 2018). Nach unserem besten Wissen wurde die Astrovirus-assoziierte Enzephalitis noch nicht experimentell reproduziert. Dies würde die Isolierung neuroinvasiver Astroviren aus klinischen Fällen erfordern, was in unserem Fall nicht versucht wurde.
Die Quelle von BoAstV-Neuro-Uy konnte in diesem Fall nicht bestimmt werden. Reservoirrinder und Wildtiere sollten jedoch berücksichtigt werden, da die Rinder unter ausgedehnten Außenbedingungen aufgezogen wurden. Das betroffene Tier war im Februar 2018 zusammen mit anderen 9 Ochsen gekauft und auf den Hof gebracht worden. Leider lehnte der Eigentümer weitere Probenahmen und Tests anderer Tiere auf dem Grundstück sowie eine detailliertere epidemiologische Untersuchung ab. Keines der anderen Tiere in der Gruppe hatte bis August 2018, dem letzten Kontakt mit dem Tierarzt, eine neurologische Erkrankung entwickelt. Für Fälle von Astrovirus-assoziierter Enzephalitis in der Schweiz wurde eine Saisonalität vom Beginn des Winters bis zum Ende des Frühlings vorgeschlagen (Selimovic-Hamza et al., 2016). Interessanterweise ereignete sich der hier beschriebene Fall im Juni, entsprechend der Herbst-Winter-Übergangszeit in der südlichen Hemisphäre.
Während in Nordamerika neurotrope Astroviren identifiziert wurden (Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) und Asien (Hirashima et al., 2018) wurde ihre Anwesenheit auf der südlichen Hemisphäre noch nie berichtet, so dass diese Mitteilung die geografische Verbreitung der Astrovirus-assoziierten Enzephalitis erweitert. Um zu beurteilen, ob der in Uruguay nachgewiesene Virusstamm möglicherweise aus Europa stammt, Nordamerika, oder Asien, Wir schätzten die Evolutionsrate und führten eine phylogeographische Analyse unter Verwendung neuroinvasiver BoAstV-Sequenzen durch, die in der GenBank verfügbar sind. Die unter Verwendung der vollständigen Codierungsregion geschätzte Evolutionsrate betrug 4.27 × 10-4 (95% höchste Wahrscheinlichkeitsdichte -HPD-, 2,19–6,46 × 10-4) Nukleotidsubstitutionen / Stelle / Jahr, die für ein RNA-Virus erwartet werden (Jenkins et al., 2002), aber niedriger als für enterische humane Astroviren geschätzt (Babkin et al., 2012, 2014). Die ORF1ab–Region zeigte eine ähnliche Evolutionsrate (4,20 × 10-4, 95% HPD 1,66-6,46 × 10-4 Substitutionen / Stelle / Jahr) wie die vollständige kodierende Region, während die ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6,46 × 10-4 Substitutionen / Stelle / Jahr) und ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 Substitutionen / Stelle / Jahr) zeigten eine etwas schnellere Evolutionsrate, und die partielle Polymerase-Genomregion (ORF1b) zeigte eine etwas langsamere Evolutionsrate (5,39 × 10-4, 95% HPD 6,41 × 10-7-1,10 × 10-3 Substitutionen / Stelle / Jahr).
Wie durch die phylogeographische Analyse mit der vollständigen kodierenden Region bestimmt und im MCTT gezeigt (Abbildung 3), gibt es zwei Unterlinien (CH13 und CH1) basierend auf Referenzstämmen, die einen gemeinsamen Vorfahren haben. Der jüngste gemeinsame Vorfahr dieser Unterlinien (Linie CH13 / CH1) entstand ungefähr 1885 in Europa (95% HPD, 1794-1940). Zu Beginn der 1900er Jahre gingen die beiden Unterlinien auseinander, die CH13-Unterlinie blieb in Europa im Umlauf, während sich die CH1-Unterlinie nach Amerika und Asien ausbreitete. Das wahrscheinlichste Szenario ist, dass die H1-Unterlinie in Uruguay um das Jahr 1921 aus Europa eingeführt wurde (95% HPD, 1849-1967), vermutlich durch Viehhandel, und sich dann nach Nordamerika und später nach Japan ausbreitete (Abbildung 3). Aufgrund der Begrenzung der Anzahl der in GenBank verfügbaren Sequenzen, die die Analyse verzerrt haben könnte, wurden die Ergebnisse, die unter Verwendung der vollständigen kodierenden Region erhalten wurden, mit denen verglichen, die mit anderen genomischen Regionen (ORF1ab, ORF2, ORF1a und ORF1b) für eine größere Anzahl von Stämmen (d. H. Kanadischen Stämmen) erhalten wurden. In allen Analysen ist das wahrscheinlichste Szenario, dass die Einschleppung des Virus aus Europa nach Uruguay erfolgte (Ergänzende Abbildungen S1A–D). Darüber hinaus war das geschätzte Datum für diese Einführung, das mit der ORF1ab- und partiellen Polymerase-Genomregion (ORF1b) erhalten wurde (ergänzende Abbildungen S1A, D), ähnlich dem, das mit der vollständigen kodierenden Region erhalten wurde, während das geschätzte Datum der Einführung, das mit ORF2 und ORF1a erhalten wurde, früher war, jedoch mit einem breiteren 95% HPD-Intervall (ergänzende Abbildungen S1B, C). Eine Einführung der Unterlinie S1 direkt nach Kanada aus Europa, mit anschließender Ausbreitung in den Vereinigten Staaten und Japan, ist ebenfalls plausibel, s in ergänzender Abbildung S1D gezeigt.

Abbildung 3. Maximaler Clade-Glaubwürdigkeitsbaum, der durch Analyse des Codierungsbereichs in voller Länge erhalten wird. Die Farbe der Zweige repräsentiert das wahrscheinlichste Land, in dem die Vorfahren zirkulierten, die Wahrscheinlichkeitswerte werden in den Zweigen angezeigt und die Zahlen in jedem Knoten repräsentieren die Herkunftsjahre für jede Gruppe mit dem 95% HPD-Intervall. Unterlinien sind mit Etiketten gekennzeichnet.
Weitere Untersuchungen sind erforderlich, um die geografische Verteilung, pathogene Mechanismen (insbesondere Mechanismen der Übertragung und des Eintritts), molekulare Epidemiologie und mögliche Interspezies-Übertragung von neuroinvasiven Astroviren zu bewerten.
Datenverfügbarkeit
Die für diese Studie generierten Datensätze finden Sie in GenBank, MK386569.
Autorenbeiträge
FG, RDC und MC haben an der Konzeption der Studie mitgewirkt. FG und RDC führten die pathologische Untersuchung und Probenahme durch. PP führte die in situ Hybridisierung durch. FU führte die Immunhistochemie durch. LM, RC und MC führten molekulare virologische Tests durch. MC führte die Sequenz und phylogeographische Analysen und die damit verbundenen Zahlen. FG und PP erhielten die histologischen Bilder. MF führte die Bakterienkulturen durch. FG und MC schrieben den ersten Entwurf des Manuskripts. RDC, PP, FU, LM, MF und RC schrieben Abschnitte des Manuskripts. Alle Autoren haben zur Überarbeitung des Manuskripts beigetragen, die eingereichte Version gelesen und genehmigt.
Finanzierung
Diese Arbeit wurde durch Zuschüsse PL-015 N-15156 von INIA und 158 aus dem „Programa de Iniciación a la Investigación 2017 „von“ Comisión Sectorial de Investigación Científica “ (CSIC) finanziert. MC und RDC erkennen die Unterstützung der „National Agency for Research and Innovation “ (ANII) bzw. der INIA durch Doktorandenstipendien an. FG erkennt Unterstützung von ANII durch Mobilitätszuschuss MOV_CA_2018_1_150021.
Interessenkonflikterklärung
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Der Rezensent TS hat gegenüber dem Herausgeber eine frühere Koautorschaft mit einem der Autoren PP erklärt.
Danksagung
Die Autoren danken Yisell Perdomo und Cecilia Monesiglio von INIA sowie Karen Sverlow und Juliann Beingesser von CAHFS für die technische Unterstützung.
Ergänzungsmaterial
Das Ergänzungsmaterial zu diesem Artikel finden Sie online unter: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
ABBILDUNG S1 / Maximum Clade Credibility Trees (MCCTs), erhalten durch Analyse des ORF1ab (A) in voller Länge, des ORF2 (B) in voller Länge, des ORF1a (C) in voller Länge und des partiellen ORF1b (D) in voller Länge. Die Farbe der Zweige repräsentiert das wahrscheinlichste Land, in dem die Vorfahren zirkulierten, die Wahrscheinlichkeitswerte werden in den Zweigen angezeigt, und die Zahlen in jedem Knoten repräsentieren die Herkunftsjahre für jede Gruppe mit dem 95% HPD-Intervall. Unterlinien sind mit Etiketten gekennzeichnet.
Fußnoten
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N. und Sandine, W. E. (1990). Hochselektives Medium zur Isolierung von Listeria monocytogenes aus Lebensmitteln. Appl. ENVIRON. Microbiol. 56, 3154–3157.
PubMed Zusammenfassung / Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chowdhury, S. und Collins, J. K. (1997). Spezifischer Nachweis von Shedding und Latenz des bovinen Herpesvirus 1 und 5 mittels einer verschachtelten Polymerase-Kettenreaktion. J. Tierarzt. Diagn. Investieren. 9, 387–394. doi: 10.1177/104063879700900408
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V. und Tikunova, N. V. (2014). Rekombinationsanalyse basierend auf den HAstV-2- und HastV-4-Komplettgenomen. Infizieren. Genet. In: Evol. 22, 94–102. modell: 10.1016/j.meegid.2014.01.010
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V. und Tikunova, N. V. (2012). Hohe Evolutionsrate des menschlichen Astrovirus. Infizieren. Genet. In: Evol. 12, 435–442. modell: 10.1016/j.meegid.2012.01.019
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Blomström, A. L., Widén, F., Hammer, A. S., Belák, S. und Berg, M. (2010). Nachweis eines neuartigen Astrovirus im Hirngewebe von Nerzen mit Shaking Mink Syndrom mittels viraler Metagenomik. J. Clin. Microbiol. 48, 4392–4396. doi: 10.1128/JCM.01040-10
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Es sind keine Frei zugänglichen ergänzenden Materialien verfügbar Zitation Boros,Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., et al. (2017). Ausbrüche des neuroinvasiven Astrovirus im Zusammenhang mit Enzephalomyelitis, Schwäche und Lähmung bei entwöhnten Schweinen, Ungarn. Emerg. Infizieren. Dis. 23, 1982–1993. doi: 10.3201/eid2312.170804
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Wüthrich, D., Jakupovic, D., Bruggmann, R., Wüthrich, D., Brüggmann, R., Wüthrich, D., et al. (2017). Hinweis auf eine artübergreifende Übertragung des Astrovirus im Zusammenhang mit Enzephalitis bei Schafen und Rindern. Emerg. Infizieren. Dis. 23, 1604–1608. doi: 10.3201/eid2309.170168
PubMed Abstract | CrossRef Full Text | Google Scholar
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., and Seuberlich, T. (2016). Full-genome based molecular characterization of encephalitis-associated bovine astroviruses. Infect. Genet. Evol. 44, 162–168. doi: 10.1016/j.meegid.2016.06.052
PubMed Abstract | CrossRef Full Text | Google Scholar
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C., Zurbriggen, A., Vandevelde, M., et al. (2014). Neurotropes Astrovirus bei Rindern mit nicht suppurativer Enzephalitis in Europa. J. Clin. Microbiol. 52, 3318–3324. doi: 10.1128/JCM.01195-14
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Cantile, C. und Youssef, S. (2016). „Nervensystem“, in Jubb, Kennedy und Palmers Pathologie von Haustieren, 6. Aufl., Hrsg. M. G. Maxie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T. und Meylan, M. (2017). Neurologische klinische Symptome bei Rindern mit Astrovirus-assoziierter Enzephalitis. J. Tierarzt. Praktikant. Med. 31, 1209–1214. doi: 10.1111/jvim.14728
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Donato, C. und Vijaykrishna, D. (2017). Das breite Wirtsspektrum und die genetische Vielfalt von Säugetier- und Vogel-Astroviren. Kapitel 9:E102. doi: 10.3390/v9050102
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Drummond, AJ, Suchard, MA, Xie, D. und Rambaut, A. (2012). Bayesianische Phylogenetik mit BEAUti und dem BIEST 1.7. Mol. Biol. In: Evol. 29, 1969–1973. doi: 10.1093/molbev/mss075
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Es sind keine frei zugänglichen ergänzenden Materialien verfügbar Zitation Giannitti, F., Anderson, M., Miller, M., Rowe, J., Vasquez, M., et al. (2016). Chlamydia pecorum: Fetale und plazentare Läsionen bei sporadischem Ziegenabbruch. J. Tierarzt. Diagn. Investieren. 28, 184–189. doi: 10.1177/1040638715625729
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Vollständige Genomanalyse eines neuartigen neurotropen bovinen Astrovirus, das in einem japanischen schwarzen Ochsen mit nicht-suppurativer Enzephalomyelitis in Japan nachgewiesen wurde. Bogen. Virol. 163, 2805–2810. ust-idnr.: 10.1007/s00705-018-3898-3
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Internationales Komitee für Taxonomie von Viren (2018). Master-Artenliste (MSL32), Version 1. Verfügbar unter: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (Zugriff März, 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G. und Holmes, E. C. (2002). Raten der molekularen Evolution in RNA-Viren: eine quantitative phylogenetische Analyse. In: J. Mol. In: Evol. 54, 156–165. ust-idnr.: 10.1007/s00239-001-0064-3
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Kumar, S., Stecher, G. und Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis Version 7.0 für größere Datensätze. Mol. Biol. In: Evol. 33, 1870–1874. doi: 10.1093/molbev/msw054
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Es sind keine frei zugänglichen ergänzenden Materialien verfügbar Zitation Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Divergentes Astrovirus im Zusammenhang mit neurologischen Erkrankungen bei Rindern. Emerg. Infizieren. Dis. 19, 1385–1392. doi: 10.3201/eid1909.130682
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Full-length human immunodeficiency Virus Typ 1 Genome von Subtyp C-infizierten Serokonverter in Indien, mit dem Nachweis von Intersubtyp-Rekombination. In: J. Virol. 73, 152–160.
PubMed Zusammenfassung / Google Scholar
Naccache, S. N., Peggs, KS, Mattes, FM, Phadke, R., Garson, JA, Grant, P., et al. (2015). Diagnose einer neuroinvasiven Astrovirus-Infektion bei einem immungeschwächten Erwachsenen mit Enzephalitis durch unvoreingenommene Sequenzierung der nächsten Generation. Clin. Infizieren. Dis. 60, 919–923. doi: 10.1093/cid/ciu912
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Pathologie und immunhistochemische Befunde der West-Nil-Virus-Infektion in psittaciformes. Tierarzt. Pathologisch. 48, 975–984. doi: 10.1177/0300985810391112
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Es sind keine frei zugänglichen ergänzenden Materialien verfügbar Zitationen Pfaff, F., Höper, D., Scholes, S., Hoffmann, B., et al. (2017). Ein neuartiges Astrovirus, das mit Enzephalitis und Ganglioneuritis bei Hausschafen assoziiert ist. Transbound. Emerg. Dis. 64, 677–682. Ursprungsbezeichnung: 10.1111/tbed.12623
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Taschmuchamedowa, A., et al. (2010). Astrovirus-Enzephalitis bei Jungen mit X-chromosomaler Agammaglobulinämie. Emerg. Infizieren. Dis. 16, 918–925. doi: 10.3201/eid1606.091536
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L. und Pybus, O. G. (2016). Erforschung der zeitlichen Struktur heterochroner Sequenzen mit TempEst (ehemals Path-O-Gen). In: Virus Evol. 2:vew007. doi: 10.1093/ve/vew007
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Reuter, G., Pankovics, P. und Boros, A. (2018). Nicht-suppurative (aseptische) Meningoenzephalomyelitis im Zusammenhang mit neurovirulenten Astrovirus-Infektionen bei Mensch und Tier. Clin. Microbiol. Offb. 31, e00040-18. doi: 10,1128/CMR.00040-18
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Es sind keine frei zugänglichen ergänzenden Materialien verfügbar Zitationen Schulze, C., Höper, D., Beer, M., et al. (2016). Nachweis eines neuartigen bovinen Astrovirus bei einer Kuh mit Enzephalitis. Transbound. Emerg. Dis. 63, 253–259. Ursprungsbezeichnung: 10.1111/tbed.12493
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A. und Seuberlich, T. (2017a). Häufigkeit und pathologischer Phänotyp der bovinen Astrovirus CH13 / NeuroS1-Infektion bei neurologisch erkrankten Rindern: zur Beurteilung der Kausalität. Kapitel 9:E12. doi: 10.3390/v9010012
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G. und Seuberlich, T. (2017b). Bovine Astrovirus-Infektion bei Feedlot-Rindern mit neurologischen Erkrankungen in Westkanada. Können. Tierarzt. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A. und Seuberlich, T. (2016). Nachweis von Astrovirus in historischen Fällen der europäischen sporadischen Rinderenzephalitis, Schweiz 1958-1976. Front. Tierarzt. Sci. 3:91. Ursprungsbezeichnung: 10.3389/fvets.2016.00091
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identifizierung eines zweiten Enzephalitis-assoziierten Astrovirus bei Rindern. Emerg. Mikroben infizieren. 5:e5. doi: 10.1038/ems.2016.5
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Spinato, MT, Vince, A., Cai, H. und Ojkic, D. (2017). Identifizierung des bovinen Astrovirus bei nicht suppurativer boviner Enzephalitis in Ostkanada. Können. Tierarzt. J. 58, 607-609.
Google Scholar
Stein, LT, Rech, RR, Harrison, L. und Brown, CC (2010). Immunhistochemische Untersuchung des Tollwutvirus im zentralen Nervensystem von Haus- und Wildtierarten. Tierarzt. Pathologisch. 47, 630–633. doi: 10.1177/0300985810370013
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A. und Minh, B. Q. (2016). W-IQ-TREE: ein schnelles Online-phylogenetisches Tool für die Maximum-Likelihood-Analyse. Nukleinsäuren Res. 44, W232-W235. doi: 10.1093/nar/gkw256
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Wiederentdeckung und genomische Charakterisierung von bovinen Astroviren. In: J. Gen. Virol. 92(Pt 8), 1888-1998. doi: 10.1099/vir.0.030817-0
PubMed Zusammenfassung / CrossRef Volltext / Google Scholar