Biología para las Carreras I
Resultados de aprendizaje
- Comprender la diferencia entre el pre-ARNm y el ARNm
Después de la transcripción, los pre-ARNm eucarióticos deben someterse a varios pasos de procesamiento antes de que puedan ser traducidos. Los ARNt y ARNR eucarióticos (y procarióticos) también se procesan antes de que puedan funcionar como componentes en la maquinaria de síntesis de proteínas.
Procesamiento de ARNm
El pre-ARNm eucariótico se somete a un procesamiento extenso antes de que esté listo para ser traducido. Los pasos adicionales involucrados en la maduración del ARNm eucariótico crean una molécula con una vida media mucho más larga que un ARNm procariótico. Los ARNm eucarióticos duran varias horas, mientras que el ARNm típico de E. coli no dura más de cinco segundos.
Los Pre-ARNm se recubren primero en proteínas estabilizadoras de ARN; estas protegen al pre-ARNm de la degradación mientras se procesa y exporta fuera del núcleo. Los tres pasos más importantes del procesamiento previo al ARNm son la adición de factores estabilizadores y de señalización en los extremos 5′ y 3′ de la molécula, y la eliminación de secuencias intermedias que no especifican los aminoácidos apropiados. En casos raros, la transcripción del ARNm puede ser «editada» después de ser transcrita.
Tapado de 5′
Mientras el pre-ARNm todavía se está sintetizando, se agrega una tapa de 7-metilguanosina al extremo de 5′ de la transcripción creciente mediante un enlace de fosfato. Esta fracción (grupo funcional) protege el ARNm naciente de la degradación. Además, los factores involucrados en la síntesis de proteínas reconocen la tapa para ayudar a iniciar la traducción por los ribosomas.
Cola de Poli-A de 3′
Una vez que se completa la elongación, el pre-ARNm es escindido por una endonucleasa entre una secuencia de consenso AAUAAA y una secuencia rica en GU, dejando la secuencia AAUAAA en el pre-ARNm. Una enzima llamada polimerasa poli-A luego agrega una cadena de aproximadamente 200 residuos A, llamada cola poli-A. Esta modificación protege aún más el pre-ARNm de la degradación y señala la exportación de los factores celulares que la transcripción necesita al citoplasma.
Empalme pre-ARNm
Los genes eucarióticos se componen de exones, que corresponden a secuencias codificantes de proteínas (ex-on significa que se expresan), y secuencias intervinientes llamadas intrones (intrón denota su papel interviniente), que pueden estar involucrados en la regulación de los genes, pero se eliminan del pre-ARNm durante el procesamiento. Las secuencias de intrones en el ARNm no codifican proteínas funcionales.
El descubrimiento de intrones fue una sorpresa para los investigadores en la década de 1970 que esperaban que los pre-ARNm especificaran secuencias de proteínas sin procesamiento adicional, como habían observado en procariotas. Los genes de los eucariotas superiores a menudo contienen uno o más intrones. Estas regiones pueden corresponder a secuencias reguladoras; sin embargo, la importancia biológica de tener muchos intrones o tener intrones muy largos en un gen no está clara. Es posible que los intrones ralenticen la expresión génica porque se necesita más tiempo para transcribir pre-ARNm con muchos intrones. Alternativamente, los intrones pueden ser restos de secuencias no funcionales que quedan de la fusión de genes antiguos a lo largo de la evolución. Esto se apoya en el hecho de que los exones separados a menudo codifican subunidades o dominios de proteínas separados. En su mayor parte, las secuencias de intrones pueden mutarse sin afectar en última instancia al producto proteínico.
Todos los intrones de un pre-ARNm deben eliminarse por completo y con precisión antes de la síntesis de proteínas. Si el proceso falla incluso en un solo nucleótido, el marco de lectura de los exones reincorporados cambiaría, y la proteína resultante sería disfuncional. El proceso de extracción de intrones y reconexión de exones se denomina empalme (Figura 1). Los intrones se eliminan y degradan mientras el pre-ARNm todavía está en el núcleo. El empalme se produce por un mecanismo de secuencia específica que asegura que se eliminarán los intrones y se unirán los exones con la precisión y exactitud de un solo nucleótido. El empalme de pre-ARNm se lleva a cabo por complejos de proteínas y moléculas de ARN llamados empalmes.
Pregunta práctica
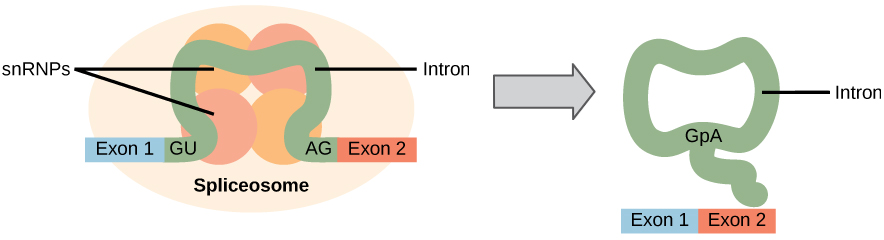
Figura 1. El empalme previo al ARNm implica la eliminación precisa de intrones de la transcripción primaria del ARN. El proceso de empalme es catalizado por complejos de proteínas llamados spliceosomas que están compuestos de proteínas y moléculas de ARN llamadas snRNAs. Los spliceosomas reconocen secuencias en el extremo 5′ y 3′ del intrón.
Los errores en el empalme están implicados en cánceres y otras enfermedades humanas. ¿Qué tipos de mutaciones pueden llevar a errores de empalme?
Tenga en cuenta que pueden estar presentes más de 70 intrones individuales, y cada uno tiene que someterse al proceso de empalme, además del tapado de 5′ y la adición de una cola poli—A, solo para generar una sola molécula de ARNm traducible.
La edición de ARN en Tripanosomas
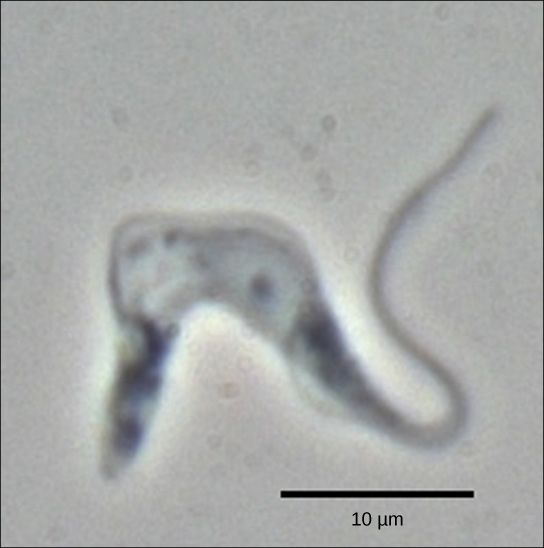
Figura 2. El Trypanosoma brucei es el agente causal de la enfermedad del sueño en los seres humanos. Los ARNM de este patógeno deben modificarse mediante la adición de nucleótidos antes de que pueda producirse la síntesis de proteínas. (crédito: modificación de la obra de Torsten Ochsenreiter)
Los tripanosomas son un grupo de protozoos que incluyen el patógeno Trypanosoma brucei, que causa la enfermedad del sueño en los seres humanos (Figura 2). Los tripanosomas, y prácticamente todos los demás eucariotas, tienen orgánulos llamados mitocondrias que suministran energía química a la célula. Las mitocondrias son orgánulos que expresan su propio ADN y se cree que son los restos de una relación simbiótica entre un eucariota y un procariota envuelto. El ADN mitocondrial de los tripanosomas exhibe una interesante excepción al Dogma Central: sus pre-ARNm no tienen la información correcta para especificar una proteína funcional. Por lo general, esto se debe a que al ARNm le faltan varios nucleótidos U. La célula realiza un paso adicional de procesamiento de ARN llamado edición de ARN para remediar esto.
Otros genes del genoma mitocondrial codifican ARN guía de 40 a 80 nucleótidos. Una o más de estas moléculas interactúan por emparejamiento de bases complementarias con algunos de los nucleótidos en la transcripción pre-ARNm. Sin embargo, el ARN guía tiene más nucleótidos A que el pre-ARNm tiene nucleótidos U con los que unirse. En estas regiones, el ARN guía se enrolla. Los extremos de 3′ de los ARN guía tienen una larga cola poli-U, y estas bases U se insertan en regiones de la transcripción pre-ARNm en la que se enrollan los ARN guía. Este proceso está completamente mediado por moléculas de ARN. Es decir, los ARN guía, en lugar de las proteínas, sirven como catalizadores en la edición de ARN.
La edición de ARN no es solo un fenómeno de tripanosomas. En las mitocondrias de algunas plantas, casi todos los pre-ARNm son editados. La edición de ARN también se ha identificado en mamíferos como ratas, conejos e incluso humanos. ¿Cuál podría ser la razón evolutiva para este paso adicional en el procesamiento previo al ARNm? Una posibilidad es que las mitocondrias, que son restos de procariotas antiguos, tengan un método igualmente antiguo basado en ARN para regular la expresión génica. En apoyo de esta hipótesis, las ediciones hechas a pre-ARNm difieren dependiendo de las condiciones celulares. Aunque especulativo, el proceso de edición de ARN puede ser un remanente de un tiempo primordial en el que las moléculas de ARN, en lugar de las proteínas, eran responsables de catalizar las reacciones.
Pruébalo
Contribuir!
Mejora esta pagaprender más