Biología para las Carreras II
Resultados de aprendizaje
- Comprender los sistemas de clasificación de virus anteriores y emergentes
Debido a que la mayoría de los virus probablemente evolucionaron de diferentes antepasados, los métodos sistemáticos que los científicos han utilizado para clasificar las células procariotas y eucariotas no son muy útiles. Si los virus representan «restos» de diferentes organismos, incluso el análisis genómico o de proteínas no es útil. ¿Por qué?, porque los virus no tienen una secuencia genómica común que todos compartan. Por ejemplo, la secuencia de ARNr 16S, tan útil para construir filogenias procariotas, no sirve de nada para una criatura sin ribosomas. Los biólogos han utilizado varios sistemas de clasificación en el pasado. Los virus se agruparon inicialmente por morfología compartida. Más tarde, los grupos de virus se clasificaron por el tipo de ácido nucleico que contenían, ADN o ARN, y si su ácido nucleico era de cadena simple o doble. Sin embargo, estos métodos de clasificación anteriores agrupaban los virus de manera diferente, porque se basaban en diferentes conjuntos de caracteres del virus. El método de clasificación más comúnmente utilizado hoy en día se llama el esquema de clasificación de Baltimore, y se basa en cómo se genera el ARN mensajero (ARNm) en cada tipo particular de virus.
Sistemas de clasificación anteriores
Los virus contienen solo unos pocos elementos por los que se pueden clasificar: el genoma viral, el tipo de cápside y la estructura de la envoltura de los virus envueltos. Todos estos elementos se han utilizado en el pasado para la clasificación viral (Tabla 1 y Figura 1). Los genomas virales pueden variar en el tipo de material genético (ADN o ARN) y su organización (de una o dos cadenas, lineales o circulares, segmentadas o no segmentadas). En algunos virus, las proteínas adicionales necesarias para la replicación están asociadas directamente con el genoma o contenidas dentro de la cápside viral.
| Cuadro 1 Clasificación de Virus por Estructura y Núcleo del Genoma | |
|---|---|
| Clasificaciones básicas | Ejemplos |
| ARN | Virus de la rabia, retrovirus |
| ADN | Virus de la viruela, virus de la viruela |
| Virus de la rabia monocatenario | , retrovirus |
| Virus de la viruela, virus de la viruela de doble cadena | |
| Linear | Virus de la rabia, retrovirus, virus herpesvirus, virus de la viruela |
| Circular | Virus del papiloma, muchos bacteriófagos |
| No segmentado: el genoma consiste en un único segmento de material genético | virus de la parainfluenza |
| Segmentado: el genoma se divide en múltiples segmentos | Virus de la gripe |
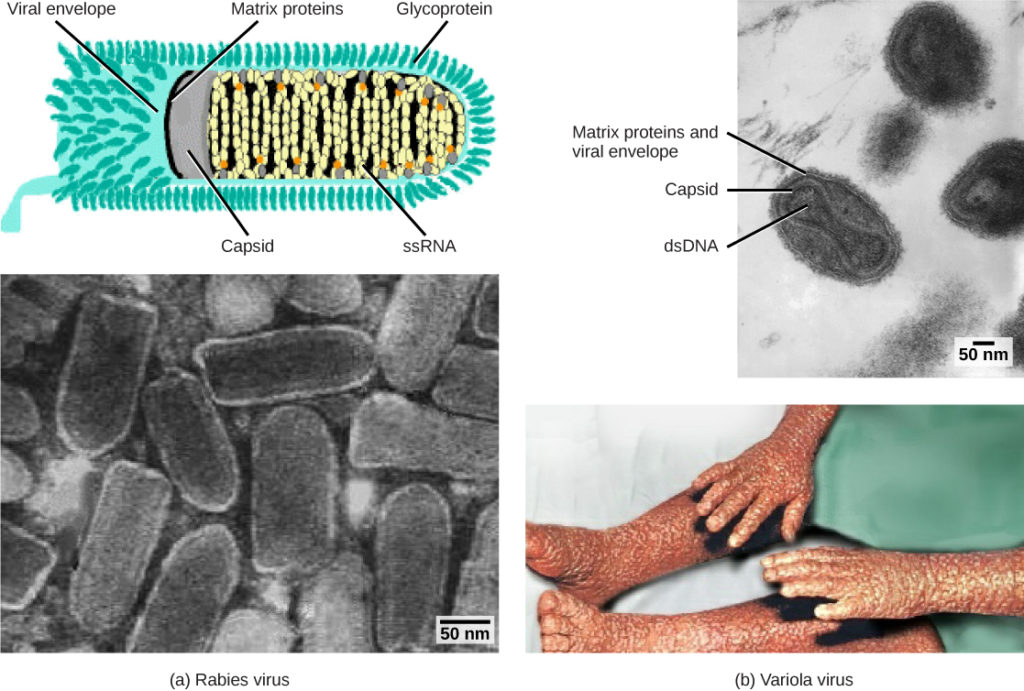
Figura 1. Los virus se clasifican según su material genético central y el diseño de la cápside. a) El virus de la rabia tiene un núcleo de ARN monocatenario (ARNSRS) y una cápside helicoidal envuelta, mientras que b) el virus de la variola, el agente causante de la viruela, tiene un núcleo de ADN bicatenario (arNDD) y una cápside compleja. (crédito «diagrama de la rabia»: modificación del trabajo de los CDC; «micrografía de la rabia»: modificación del trabajo del Dr. Fred Murphy, CDC; crédito «micrografía de la viruela pequeña»: modificación del trabajo del Dr. Fred Murphy, Sylvia Whitfield, CDC; crédito «foto de la viruela»: modificación del trabajo de los CDC; datos de barras de escala de Matt Russell)
Los virus también pueden clasificarse por el diseño de sus cápsidos (Tabla 2 y Figura 2). Los capsidos se clasifican como icosaédricos desnudos, icosaédricos envueltos, helicoidales envueltos, helicoidales desnudos y complejos. El tipo de material genético (ADN o ARN) y su estructura (monocatenaria o bicatenaria, lineal o circular, segmentada o no segmentada) se utilizan para clasificar las estructuras básicas del virus (Tabla 2).
| Cuadro 2 Clasificación de Virus por Estructura de Cápside | |
|---|---|
| Clasificación de la cápside | Ejemplos |
| Virus icosaédrico desnudo | Hepatitis A, poliovirus |
| Icosaédrico envuelto | Virus de Epstein-Barr, virus del herpes simple, virus de la rubéola, virus de la fiebre amarilla, VIH-1 |
| Helicoidal con envoltura | Virus de la gripe, virus de las paperas, virus del sarampión, virus de la rabia |
| Virus del mosaico del tabaco helicoidal desnudo | |
| Complejo con muchas proteínas; algunos tienen combinaciones de estructuras de la cápside icosaédrica y helicoidal | Virus de la viruela, virus de la hepatitis B, bacteriófago T4 |

Figura 2. Micrografías electrónicas de transmisión de varios virus muestran sus estructuras. La cápside del virus de la polio (a) es icosaédrica desnuda; (b) la cápside del virus de Epstein-Barr es icosaédrica envuelta; (c) la cápside del virus de las paperas es una hélice envuelta; (d) la cápside del virus del mosaico del tabaco es helicoidal desnuda; y (e) la cápside del virus del herpesvirus es compleja. (crédito a: modificación del trabajo del Dr. Fred Murphy, Sylvia Whitfield; crédito b: modificación del trabajo de Liza Gross; crédito c: modificación del trabajo del Dr. F. A. Murphy, CDC; crédito d: modificación del trabajo del USDA ARS; crédito e: modificación del trabajo de Linda Stannard, Departamento de Microbiología Médica, Universidad de Ciudad del Cabo, Sudáfrica, NASA; datos de barras a escala de Matt Russell)
Clasificación de Baltimore
El sistema de clasificación de virus más utilizado fue desarrollado por el biólogo ganador del Premio Nobel David Baltimore a principios de la década de 1970. Además de las diferencias en morfología y genética mencionadas anteriormente, el esquema de clasificación de Baltimore agrupa los virus de acuerdo con la forma en que se produce el ARNm durante el ciclo replicativo del virus.
Los virus del grupo I contienen ADN de doble cadena (dsDNA) como genoma. Su ARNm se produce por transcripción de la misma manera que con el ADN celular.
Los virus del grupo II tienen ADN monocatenario (ADNss) como genoma. Convierten sus genomas monocatenarios en un intermediario de dsDNA antes de que pueda ocurrir la transcripción a ARNm.
Los virus del grupo III utilizan el dsRNA como genoma. Las hebras se separan, y una de ellas se utiliza como plantilla para la generación de ARNm utilizando la ARN polimerasa dependiente de ARN codificada por el virus.
Los virus del grupo IV tienen un ARNSRS como genoma con una polaridad positiva. La polaridad positiva significa que el ARN genómico puede servir directamente como ARNm. Los intermedios del dsRNA, llamados intermedios replicativos, se fabrican en el proceso de copiar el ARN genómico. Múltiples hebras de ARN de longitud completa de polaridad negativa (complementarias al ARN genómico de cadena positiva) se forman a partir de estos intermedios, que luego pueden servir como plantillas para la producción de ARN con polaridad positiva, incluyendo tanto ARN genómico de longitud completa como ARNm virales más cortos.
Los virus del grupo V contienen genomas de ARNSRS con una polaridad negativa, lo que significa que su secuencia es complementaria al ARNm. Al igual que con los virus del Grupo IV, los intermediarios del dsRNA se utilizan para hacer copias del genoma y producir ARNm. En este caso, el genoma de cadena negativa se puede convertir directamente en ARNm. Además, las hebras de ARN positivo de longitud completa están hechas para servir como plantillas para la producción del genoma de cadena negativa.
Los virus del grupo VI tienen genomas diploides (dos copias) de ARNSRS que deben convertirse, utilizando la enzima transcriptasa inversa, en arNDD; el dsDNA se transporta al núcleo de la célula huésped y se inserta en el genoma del huésped. Luego, el ARNm se puede producir mediante la transcripción del ADN viral que se integró en el genoma del huésped.
Los virus del grupo VII tienen genomas parciales de dsDNA y fabrican intermediarios de ssRNA que actúan como ARNm, pero también se convierten de nuevo en genomas de dsDNA mediante transcriptasa inversa, necesaria para la replicación del genoma. Las características de cada grupo en la clasificación de Baltimore se resumen en la Tabla 3 con ejemplos de cada grupo.
| Cuadro 3 Clasificación de Baltimore | |||
|---|---|---|---|
| Grupo | Características | Modo de producción de ARNm | Ejemplo |
| I | ADN de doble cadena | El ARNm se transcribe directamente de la plantilla de ADN | Herpes simple (herpesvirus) |
| II | ADN monocatenario | El ADN se convierte a una forma bicatenaria antes de transcribir el ARN | parvovirus canino (parvovirus) |
| III | ARN de doble cadena | El ARNm se transcribe de el genoma del ARN | Gastroenteritis infantil (rotavirus) |
| IV | ARN monocatenario (+) | El genoma funciona como ARNm | Resfriado común (pircornavirus) |
| V | ARN monocatenario ( – ) | El ARNm se transcribe del genoma del ARN | Rabia (rabdovirus) |
| VI | Virus de ARN monocatenario con transcriptasa inversa | La transcriptasa inversa produce ADN a partir del genoma del ARN; el ADN se incorpora al genoma del huésped; El ARNm se transcribe del ADN incorporado | Virus de inmunodeficiencia humana (VIH) |
| Virus de ADN de doble cadena con transcriptasa inversa | El genoma viral es ADN de doble cadena, pero el ADN viral se replica a través de un intermediario de ARN; el ARN puede servir directamente como ARNm o como una plantilla para hacer ARNm | Virus de la hepatitis B (hepadnavirus) | |