Blog Creativo de Proteómica
La secuencia de aminoácidos en una proteína o péptido se puede identificar mediante la degradación de Edman, que fue desarrollada por Pehr Edman. Este método puede etiquetar y escindir el péptido del terminal N sin interrumpir los enlaces peptídicos entre otros residuos de aminoácidos. La reacción de degradación de Edman fue automatizada en 1967 por Edman y Beggs. Hoy en día, la degradación automatizada de Edman (secuenciador de proteínas) se usa ampliamente y puede secuenciar péptidos de hasta 50 aminoácidos.
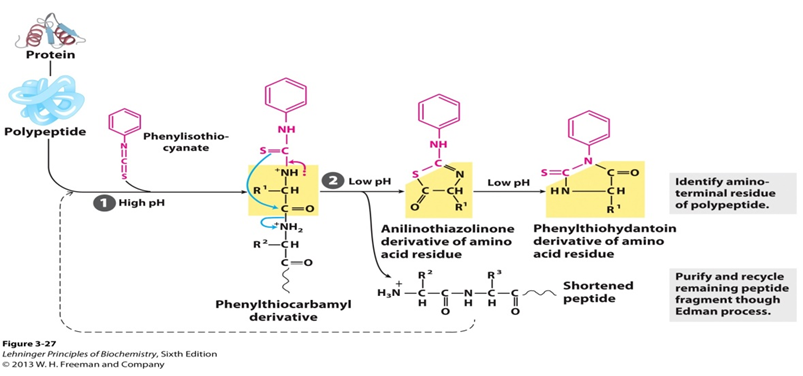
Degradación cíclica de péptidos basada en la reacción de fenilisotiocianato con el grupo amino libre del residuo N-terminal, de modo que los aminoácidos se eliminan uno a la vez y se identifican como sus derivados de feniltiohidantoína. Hablando del proceso específico, un péptido sin carga reacciona con fenilisotiocianato (PITC) en el terminal amino en condiciones ligeramente alcalinas para dar un derivado feniltiocarbamoil (péptido PTC). Luego, en condiciones ácidas, el azufre tiocarbonilo del derivado ataca el carbono carbonilo del aminoácido N-terminal. El primer aminoácido se escinde como derivado de anilinotiazolinona (aminoácido ATZ) y el resto del péptido se puede aislar y someter al siguiente ciclo de degradación. Una vez formado, este derivado de tiazolona es más estable que el derivado de feniltiocarbamilo. El aminoácido ATZ se elimina por extracción con ácido etílico y se convierte en un derivado de feniltiohidantoína (aminoácido PTH). Y la cromatografía se puede utilizar para identificar los residuos de PTH generados por cada ciclo.
En cuanto a la degradación automatizada de Edman, las proteínas se pueden analizar aplicándolas en solución en un filtro TFA y luego cargarlas en el instrumento de secuenciación Edman. Las proteínas en mezclas se separan primero por geles 1D o 2D y luego se secan en una membrana de PVDF. Las proteínas se detectan mediante tinción de azul de Coomassie, negro de Amido o Poncau y las proteínas de interés se cortan y la pieza de membrana de PVDF se carga en el secuenciador Edman.
Con la espectrometría de masas desarrollada, el uso de la secuenciación de degradación de Edman comenzó a disminuir. Sin embargo, sigue siendo los métodos para varios tipos de aplicaciones de análisis estructural de proteínas. Se puede usar para verificar el límite N-terminal de proteínas recombinantes o para determinar el N-terminal de dominios resistentes a la proteasa, particularmente cuando la proteína o dominio es >40 a 80 kDa o no puede purificarse fácilmente. También se puede utilizar para identificar el nuevo sitio de escisión proteolítica y N-terminal en los fragmentos de proteína. Además, en cuanto a algunas proteínas y péptidos novedosos donde las bases de datos de secuencias no están disponibles para la búsqueda de bases de datos MS/MS, la degradación de Edman se puede usar para el análisis.
El aminoácido N-terminal de la proteína puede ser escindido. Por lo tanto, en el proceso, el primer ciclo identifica el aminoácido N-terminal exacto. Además, debido a que los aminoácidos liberados se identifican y cuantifican por cromatografía, se pueden identificar los aminoácidos con un peso molecular idéntico. Por ejemplo, la isoleucina y la leucina tienen una masa de 113 Da, pero tienen un tiempo de retención diferente. Además, la secuenciación de Edman se puede realizar en blots de PVDF de geles 1D y 2D, lo que permite la secuenciación N-terminal de proteínas en la mezcla. Sin embargo, la secuenciación de degradación de Edman no estará disponible cuando el péptido cuyo N-terminal ha sido modificado químicamente, como la acetilación. Y como el PITC no puede reaccionar con ácido no α-aminoácido, la secuenciación se detendrá si se encuentra un ácido no α-aminoácido como el ácido isoaspártico. Además, las proteínas más grandes no pueden ser secuenciadas por la secuenciación de Edman.
En Creative Proteomics, nuestra plataforma profesional de secuenciación de proteínas proporciona análisis de secuencias N-terminal mediante degradación Edman o espectrometría de masas (MS), con ventajas complementarias entre sí. Nuestro equipo de expertos con amplia experiencia puede ayudarlo a comprender lo que está tratando de investigar y cumplir con sus requisitos. Además, podemos proporcionar otros servicios de identificación de proteínas, que incluyen:
- Servicio de huellas dactilares de masa de péptidos (PMF)
- Análisis de secuencias de péptidos o proteínas
- Servicio de análisis de secuencias de proteínas de novo
- Servicio de identificación de proteínas de escopeta
- Servicio de proteómica de membrana