Estructura molecular Revela Cómo Funciona el Antibiótico Estreptomicina / Sala de prensa de BNL
Estructura molecular Revela Cómo funciona el Antibiótico Estreptomicina
5 de septiembre de 2013
La estreptomicina fue el primer antibiótico desarrollado para tratar la tuberculosis. Sabían que la estreptomicina bloquea un proceso crítico, la síntesis de proteínas por los ribosomas que conduce a la muerte celular bacteriana, pero ciertos detalles de la interacción permanecieron sin descubrir. En la Fuente de Luz Sincrotrón Nacional del Laboratorio Nacional Brookhaven, los investigadores han utilizado cristalografía de rayos X para completar la imagen.
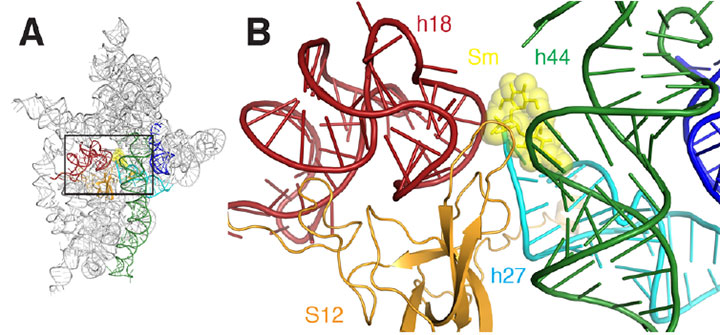
A) Un diagrama de cinta del sitio de unión de estreptomicina del ribosoma. B) Un primer plano del área rectangular delineada en A. Estreptomicina Se representa como esferas y esferas amarillas, las hélices son de color rojo, verde oscuro, cian, naranja y azul.
La estreptomicina es un miembro de una familia de antibióticos que funcionan interrumpiendo la función de los ribosomas de las células bacterianas, las complejas máquinas moleculares que crean proteínas al unir aminoácidos. Los ribosomas, un objetivo principal de los antibióticos que actúan inhibiendo la síntesis de proteínas, tienen dos partes principales o «subunidades».»
La subunidad más grande hace la construcción de la proteína, guiada por un tipo de ARN llamado ARN mensajero (ARNm), que se une a ella. La subunidad pequeña » lee » el ARNm y selecciona la molécula de ARN de transferencia (ARNt) correspondiente, que selecciona y entrega el siguiente aminoácido al ribosoma. Aquí es donde la estreptomicina juega un papel. Se une cerca de la subunidad pequeña, causando que malinterprete gravemente la secuencia. Esto resulta en la síntesis de proteínas aleatorias, que en última instancia matan a las bacterias. Pero cómo ocurrió esta lectura errónea siguió siendo un misterio, hasta un estudio reciente realizado por investigadores de la Universidad Brown y el Equipo de Acceso Colaborativo del Noreste de múltiples instituciones en el Laboratorio Nacional Argonne (administrado por la Universidad de Cornell).
Al crear un cristal-una estructura ordenada de unidades idénticas – de la pequeña subunidad ribosómica unida al ARNm en presencia de estreptomicina, los investigadores generaron varias «instantáneas» detalladas que revelaron detalles clave a nivel molecular de la interacción, mostrando finalmente cómo la estreptomicina deteriora la función de la subunidad. En la línea de haz NSLS X25, utilizaron una técnica llamada cristalografía de rayos X, en la que un haz de rayos X se dirige al cristal, interactúa con las moléculas y produce un intrincado patrón de difracción. A partir del patrón, con la ayuda de programas informáticos, el grupo construyó representaciones visuales del complejo de subunidad-ARNm-estreptomicina.
En resumen, los investigadores pudieron » ver » por primera vez las formas sutiles en que la estreptomicina distorsiona la estructura del sitio de decodificación de la subunidad, haciendo que lea incorrectamente el ARNm. Por ejemplo, la unión de estreptomicina reduce la distancia entre dos de las muchas hélices que componen la estructura molecular de la subunidad. Esto es particularmente significativo porque estas hélices forman el sitio de decodificación real, y la decodificación solo se lleva a cabo correctamente si estos elementos están orientados exactamente correctamente con respecto al ARNm y al ARNt seleccionado. La unión de estreptomicina también induce un cambio en la relación entre una de estas dos hélices y una tercera hélice, causando que una se retraiga de la otra o se «desacople».»
El resultado final de todas estas ligeras alteraciones es que la estreptomicina desestabiliza la unión entre la subunidad y el ARNt «correcto», al mismo tiempo que estabiliza la unión de la subunidad al ARNt «incorrecto», eliminando así la discriminación entre el ARNt correcto y el incorrecto. Esto causa estragos en la cadena de suministro bacteriana de nuevas proteínas, interrumpiendo la reproducción y el ciclo de vida de las bacterias.
«Nuestros estudios estructurales revelaron que la estreptomicina induce distorsiones sorprendentemente grandes en el ribosoma bacteriano, lo que nos ayuda a comprender cómo este antibiótico interfiere con la síntesis de proteínas en las bacterias», dijo el investigador principal Gerwald Jogl, profesor asociado de biología en el Departamento de Biología Molecular de Brown, Biología Celular & Bioquímica. «Continuando con nuestros hallazgos actuales, ahora estamos estudiando cómo las mutaciones en los ribosomas bacterianos pueden contrarrestar estos reordenamientos estructurales y permitir que las bacterias sobrevivan a la acción letal de la estreptomicina.»
Esta investigación fue publicada en la edición del 15 de enero de 2013 de Nature Communications, bajo el título «A structural basis for streptomycin-induced misreading of the genetic code.»El apoyo provino de los Institutos Nacionales de Salud y el Departamento de Energía.
2013-4251/INT | EXT / Sala de prensa