Frontiers in Microbiology
Introducción
La familia Astroviridae contiene virus ARN monocatenario, de sentido positivo y sin envoltura, dentro de dos géneros, Mamastrovirus y Avastrovirus, que infectan a mamíferos y aves, respectivamente. Actualmente, el Comité Internacional de Taxonomía de Virus (Comité Internacional de Taxonomía de Virus , 2018) reconoce 19 especies, a saber, Mamastrovirus-1 a -19, dentro del género Mamastrovirus; sin embargo, hay numerosas cepas en espera de clasificación, algunas de las cuales se consideran provisionalmente nuevas especies (Donato y Vijaykrishna, 2017).
Desde 2010, varios astrovirus se han reconocido cada vez más como neuroinvasivos en varias especies de mamíferos, incluidos los humanos (Quan et al., 2010; Naccache et al., 2015), visón (Blomström et al., 2010), bovino (Li et al., 2013), ovejas (Pfaff et al., 2017), y cerdos (Boros et al., 2017). Después del reconocimiento inicial de encefalitis asociada a astrovirus bovino en ganado de los Estados Unidos (Li et al., 2013), un estudio retrospectivo en casos de encefalitis bovina esporádica de etiología indeterminada en Suiza reveló que este astrovirus neuroinvasivo no se había detectado durante décadas (Selimovic-Hamza et al., 2016). Aunque la epidemiología y las rutas de transmisión de estos astrovirus son desconocidas, se ha sugerido la transmisión entre especies en base al alto nivel de identidad (>98%), compartido entre los astrovirus neuroinvasivos bovinos y ovinos en los niveles de nucleótidos y aminoácidos (Boujon et al., 2017).
Astrovirus bovino (BOASTV), denominado BoAstV-NeuroS1 (Li et al., 2013) y BoAstV-CH13 (Bouzalas et al., 2014), se encontraron inicialmente en el cerebro de ganado bovino con encefalitis no supurativa en los Estados Unidos y Suiza, respectivamente. A pesar de la diferente nomenclatura, ambos virus representan la misma especie genotípica (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a) que aún está a la espera de la clasificación oficial por parte del ICTV. En 2015, se identificó una cepa de BoAstV previamente desconocida, denominada BoAstV-CH15, en el cerebro de vacas con encefalitis en Suiza. La comparación filogenética del genoma completo reveló una relación más estrecha de BoAstV-CH15 con un astrovirus ovino (OvAstV) que con BoAstV-CH13 (Seuberlich et al., 2016). La coinfección con BoAstV-CH13 y BoAstV-CH15 también se documentó en un caso(Seuberlich et al., 2016). El mismo año en Alemania, Schlottau et al. (2016) informaron de un nuevo astrovirus, a saber, el BoAstV-BH89/14, en una vaca con encefalitis, que estaba más estrechamente relacionado con el OvAstV y el BoAstV-CH15. Posteriormente, se identificó BoAstV-CH13/NeuroS1 en 2017 en casos de encefalitis bovina en el este y oeste de Canadá (Spinato et al., 2017; Selimovic-Hamza et al., 2017b). En 2018, se identificó un nuevo BoAstV neuroinvasivo estrechamente relacionado con el BoAstV-NeuroS1/BoAstV-CH13 de América del Norte y Europa en un novillo con encefalomielitis no supurativa en Japón, y se sugirió la aparición de recombinación intragenotípica entre las cepas de América del Norte y Europa (Hirashima et al., 2018).
Si bien se han notificado casos de encefalitis asociada a astrovirus en América del Norte, Europa y Asia, su presencia nunca se ha documentado en el hemisferio sur. Aquí describimos un caso de encefalitis asociada a astrovirus en ganado bovino en Uruguay, que amplía la distribución geográfica y la diversidad genética de astrovirus neuroinvasivos y proporciona evidencia filogeográfica que sugiere que este virus se introdujo en las Américas desde Europa y luego se propagó a Asia.
Materiales y métodos
Historia y Señalización
En junio de 2018, un novillo Holstein de 22 meses de edad en un grupo de 37 novillos en una granja de Colonia 300 hectáreas en Colonia, Uruguay, desarrolló cantos neurológicos progresivos que incluían comportamiento inusual, caminar sin rumbo, dar vueltas en círculos, ataxia, movimientos repetitivos y descoordinados de la lengua y recostarse. El rebaño pastaba en un pasto anual de avena y se complementaba con ensilado de maíz. Un diagnóstico clínico presuntivo de listeriosis cerebral por parte del veterinario provocó un tratamiento con penicilina y estreptomicina, sin embargo, el animal murió espontáneamente después de un curso clínico que duró 3 días.
Examen patológico, Hibridación in situ (ISH) e Inmunohistoquímica (IHQ)
Se retiró la cabeza del novillo de la canal y se presentó al Laboratorio de Diagnóstico Veterinario (Plataforma de Salud Animal) del INIA para realizar un estudio de diagnóstico. La mitad del cerebro, un segmento corto de la médula espinal cervical proximal (C1), el ganglio trigémino y la raíz del nervio trigémino, la glándula salival, el ganglio linfático retrofaríngeo, la orofaringe, el esófago, la lengua y el músculo esquelético, se fijaron por inmersión en formalina neutra tamponada al 10% durante 48-72 h. Los tejidos se procesaron rutinariamente para su histología, se incrustaron en parafina, se seccionaron en microtomas a 4-5 µm y se teñieron con hematoxilina y eosina (H&E) y manchas de gramo.
La ISH cromogénica se realizó manualmente en secciones de 5 µm de tronco cerebral, cerebro y cerebelo fijados con formalina e incrustados en parafina (FFPE) en portaobjetos Superfrost Plus (Thermo Fisher Scientific, Pittsburgh, PA, Estados Unidos) utilizando el kit de ensayo rojo RNAscope 2.5 (Cat #322360, Advanced Cell Diagnostics, Hayward, CA, Estados Unidos) y la sonda BoAstV Cat. #406921. La sonda está compuesta por pares de 20ZZ dirigidos a la región 5232-6180 del virus (GenBank KF233994. 1). Cada sección de tejido de 5 µm se pretrató con calor y proteasa antes de la hibridación por sonda durante 2 h a 40°C y se procesó según las recomendaciones del fabricante. Los controles negativos utilizados para la validación de la señal incluyeron una sonda no relacionada (compatible con el contenido de GC) en secciones en serie y sondeo de tejido de animales no infectados. Los portaobjetos fueron contra-teñidos con hematoxilina y montados con EcoMount (Biocare Medical, Concord, CA, Estados Unidos).
Además, se realizó IHC en secciones de EPFF del tronco encefálico, cerebro y cerebelo, como se describió anteriormente, para la identificación del virus del Nilo Occidental (VNO, Flavivirus) (Palmieri et al., 2011), virus de la rabia (Lyssavirus) (Stein et al., 2010), y Chlamydia spp. (Giannitti et al., 2016) antígenos.
Virología molecular
La extracción de ácido nucleico se realizó a partir de una muestra agrupada de cerebro congelado (-20°C) utilizando el Kit de Aislamiento de ácido nucleico MagMAX® (Thermo Fisher Scientific). Para la detección de astrovirus, se realizó transcripción inversa (RT) con RevertAid Reverse Transcriptase® (Thermo Fisher Scientific) y cebadores hexámeros aleatorios (Qiagen). La PCR se realizó a partir de ADNc utilizando MangoMix® (Biolina) y cebadores que amplifican un fragmento de 432 nucleótidos del gen de la polimerasa de astrovirus (Tse et al., 2011). El producto de PCR se visualizó en gel de agarosa al 2%, purificado con el Kit Combinado de Extracción Rápida de Gel PureLink® y Purificación de PCR (Invitrogen), y secuenciado en Macrogen Inc. (Seúl, Corea del Sur). Para amplificación del genoma completo de astrovirus, Máxima H Menos Transcriptasa Inversa (Thermo Fisher Scientific)y oligo(dT) 18 para la obtención de ADNc, y MangoMix® (Biolina) o Polimerasa de ADN Ranger (Biolina) con cebadores descritos por Hirashima et al., 2018, fueron utilizados. Los productos de PCR se visualizaron en gel de agarosa al 1-2%, purificados y secuenciados como se mencionó anteriormente. El ensamblaje de secuencias se llevó a cabo con SeqMan (Lasergene 8, DNASTAR). Veintiséis secuencias genómicas completas de astrovirus neuroinvasivos de bovinos, ovinos, porcinos, humanos y visones, y astrovirus bovino entérico disponibles en GenBank se descargaron y alinearon utilizando el software Clustal W en MEGA 7 (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) se utilizó para determinar el modelo de evolución de secuencias más adecuado (SYM+I+G4) y construir un árbol filogenético de máxima probabilidad con las secuencias casi completas del BoAstV detectadas en este caso, y las secuencias completas descargadas de GenBank, utilizando bootstrap como método estadístico para evaluar la robustez de los clados. La gráfica de similitud se realizó con el software SimPlot(Lole et al., 1999). Las distancias P a nivel de aminoácidos del ORF2 se estimaron con el software MEGA 7 (Kumar et al., 2016).
Además, se realizó un análisis filogeográfico bayesiano con el paquete BEAST v1.8.4 (Drummond et al., 2012), utilizando: la región de codificación completa del linaje BoAstV CH13 / NeuroS1, ORF1ab (genes no estructurales), ORF2 (gen estructural), ORF1a (proteasa) y una ORF1b parcial (región genómica de la polimerasa, para la que se disponía de cepas canadienses), con todas las secuencias disponibles en GenBank (última adhesión el 18 de abril de 2019), para determinar la tasa evolutiva, las edades/años de los antepasados comunes y la ruta más probable de circulación viral por país (Suiza, Uruguay, Estados Unidos, Canadá y Japón). La falta de recombinación en el conjunto de datos se determinó utilizando el Programa de Detección de Recombinación 4. El modelo de sustitución que mejor se ajustaba a cada alineación se determinó utilizando el software MEGA 7 a través de los valores del criterio de información bayesiano (BIC), y la estructura temporal de cada conjunto de datos se evaluó utilizando TempEst (Rambaut et al., 2016). El reloj molecular relajado lognormal con análisis de horizonte Bayesiano fue seleccionado por el Factor Bayes entre las diferentes combinaciones de relojes moleculares y antecedentes de árboles coalescentes utilizados. Se utilizó como rasgo el país de detección. La longitud de la cadena de Markov Monte Carlo fue de 100 millones de generaciones, asegurando la convergencia del análisis, evaluado en el Trazador v1. 6.0, y la probabilidad posterior se utilizó para evaluar los clados. El árbol de máxima credibilidad de clado (MCCT) se obtuvo utilizando el software TreeAnnotator de BEAST y se visualizó en FigTree v1.4.3.
Por último, el ADN extraído del cerebro congelado fue procesado por PCR para la detección de los virus del herpes bovino 1 y 5 (BHV-1 y -5), como se describió anteriormente (Ashbaugh et al., 1997).
Bacteriología
Se procesaron rutinariamente muestras frescas de cerebro y tronco cerebral para cultivos bacterianos aeróbicos en sangre y agares MacConkey, y cultivos selectivos para Listeria monocytogenes (Al-Zoreky y Sandine, 1990).
Resultados y discusión
Los signos clínicos y los hallazgos epidemiológicos en el caso aquí descrito, aunque no específicos, fueron similares a los descritos en otros casos de encefalitis asociada a astrovirus bovino, que generalmente se describe como esporádica (Selimovic-Hamza et al., 2016), con una variedad de déficits neurológicos (Deiss et al., 2017), con una duración de los signos clínicos que normalmente oscila entre 1 día y 3 semanas (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
El examen macroscópico del cerebro, el segmento C1 de la médula espinal y otros tejidos de la cabeza no reveló lesiones anatómicas macroscópicas significativas. Histológicamente, hubo meningoencefalomielitis linfocítica, histiocítica y plasmacítica de moderada a grave que afectó al telencéfalo (incluido el hemisferio cerebral y el hipocampo), el tronco encefálico y el único segmento examinado de la médula espinal. Las lesiones se distribuyeron predominantemente en la materia gris y en las áreas limitantes de la materia blanca. En las zonas afectadas hubo esposamiento perivascular e inflamación linfoplasmacítica e histiocítica y necrosis neuronal / neuronofagia con gliosis en el neuropilo adyacente. Hubo satellitosis de neuronas necróticas afectadas (Figuras 1A–D). Las lesiones fueron mucho menos frecuentes y graves en el parénquima cerebeloso, aunque hubo leptomeningitis cerebelosa multifocal moderada. No se encontraron bacterias intralesionales con tinciones de H&E y Gram. No se encontraron cambios histológicos significativos en los otros tejidos examinados.FIGURA

Gráfico 1 Lesiones histológicas en el tronco cerebral (A, B) y la corteza cerebral (C,D) y detección de ARN del VAV en la corteza cerebral (E,G). Las imágenes (A–D) son secciones del cerebro manchadas con H& E; las imágenes E y G son secciones de la corteza cerebral que demuestran hibridación utilizando ISH cromogénico utilizando la sonda específica de BoAstV, con hematoxilina; la imagen F es una sección en serie de la sonda DapB de la corteza cerebral (control negativo), con hematoxilina. A) Un espacio perivenular se expande marcadamente por células inflamatorias (en su mayoría linfocitos e histiocitos) que también se infiltran en el neuropilo adyacente. (B) La neurona en el centro tiene perikarión hipereosinofílico y cariorrexis (necrosis) y el cuerpo neuronal está rodeado por un mayor número de células gliales (satellitosis) e inflamatorias. C) La leptomeninge está infiltrada por linfocitos e histiocitos. D) Una región de la corteza cerebral con múltiples neuronas hipereosinofílicas (necróticas) y un vaso grande con manguitos linfocíticos perivasculares. (E) En una sección seriada de la corteza cerebral, la abundante marcación intracitoplasmática de ARN del VV de jactancia se representa mediante una fuerte deposición de cromógeno rojo granular dentro del citoplasma neuronal del soma y las extensiones neuronales (E, G), que no está presente con la hibridación utilizando la sonda de control negativo (F).
Se sospechó una infección viral neuroinvasiva tras el examen histológico del sistema nervioso central. El ganado con encefalitis es motivo de preocupación porque muchos neuropatógenos rumiantes son zoonóticos (Cantile y Youssef, 2016); por lo tanto, un diagnóstico de encefalitis debe requerir pruebas de laboratorio extensas para detectar agentes infecciosos cuando sea posible. En el caso descrito en este documento, IHQ para el VNO, el virus de la rabia y Chlamydia spp., y PCR para BHV-1 y -5 fueron todos negativos, y no se cultivaron bacterias patógenas de tejido cerebral. Debido a que el novillo tenía < 2 años de edad y no se observaron cambios espongiformes en el tronco encefálico, el animal no se sometió a pruebas para detectar la encefalopatía espongiforme bovina (EEB), que es una enfermedad exótica del ganado adulto que nunca se ha notificado en el Uruguay. Además, la EEB no es inflamatoria (Cantile y Youssef, 2016).
Se realizó hibridación in situ utilizando una sonda generada a partir de BoAstV-NeuroS1, y hubo hibridación de sonda abundante en el interior, y limitada al citoplasma de neuronas en el hemisferio cerebral y el hipocampo (Figuras 1E–G). En estas áreas, la hibridación por sonda se colocaba con neuronas necróticas y regiones de gliosis, sin que se detectara hibridación por sonda en las células gliales o en las células inflamatorias de los manguitos perivasculares. El ISH no detectó ácido nucleico viral en el cerebelo, que solo presentaba lesiones inflamatorias mínimas en el parénquima, pero leptomeningitis moderada o en el tronco encefálico, incluidas las secciones con inflamación grave. Esto significa que, topográficamente, la detección de la distribución viral por ISH fue más limitada que la encefalitis en las secciones examinadas, que ocasionalmente se ha descrito en casos de encefalitis asociada a BoAstV-CH13/NeuroS1 en bovinos(Selimovic-Hamza et al., 2017a, b). Una razón para esta falta ocasional de detección de ARN viral en áreas lesionadas del cerebro podría ser el límite de detección del ISH, o la eliminación del virus en áreas inflamadas del cerebro en el momento de la muerte, como se sugirió anteriormente (Selimovic-Hamza et al., 2017b). Como era de esperar, no se detectó hibridación con sonda por ISH en el tejido cerebral utilizado como control negativo.
Se detectó astrovirus en el cerebro mediante RT-PCR. El análisis casi completo de la secuencia del genoma reveló una cepa de Mamastrovirus dentro del clado CH13 / NeuroS1, que llamamos BoAstV-Neuro-Uy, la secuencia se depositó en el banco de Genes con el número de acceso MK386569. El análisis filogenético reveló la proximidad con otros astrovirus neuroinvasivos dentro del clado Virginia/Humano-Visón-Ovino (VA/HMO) (Figura 2), que contiene la mayoría de los astrovirus neuroinvasivos conocidos (Hirashima et al., 2018; Reuter et al., 2018). La secuencia casi completa de BoAstV-Neuro-Uy es de 6427 pb de longitud y tiene una identidad de secuencia del 94% con la cepa KagoshimaSR28-462. BoAstV-Neuro-Uy tiene características similares a otras cepas de linaje CH13 / NeuroS1: una región 5’UTR de 51 nt, ORF1a (proteasa) de 861 aminoácidos (aa), ORF1b de 523 aa (ARN polimerasa dependiente de ARN) y ORF2 de 758 aa (proteína de la cápside). Desafortunadamente, el 3’UTR no se pudo secuenciar, pero se presume que una cola poli(A) estaba presente porque se usó oligo(dT)18 para obtener ADNc. Además, está presente la secuencia heptamérica AAAAAC, una señal de cambio de marco ribosomal. Las distancias P a nivel de aminoácidos de la ORF2 confirmaron la asignación de esta cepa al clado CH13 / NeuroS1. Distancias P < 0.35 entre BoAstV-Neuro-Uy y otros miembros de este clado (Tabla 1) apoyaría una clasificación de estas cepas virales dentro de una misma especie; Mamastrovirus-13 ha sido propuesto recientemente por otros autores (Donato y Vijaykrishna, 2017; Hirashima et al., 2018), aunque la asignación definitiva de especies por parte del ICTV está pendiente. La sonda utilizada para el ISH, generada a partir de BoAstV-NeuroS1, tenía 92,7% de identidad de secuencia con BoAstV-Neuro-Uy.FIGURA
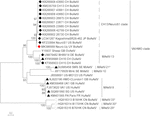
Gráfico 2 Análisis filogenético de secuencias de nucleótidos de longitud completa por el método de máxima verosimilitud. BoAstV-Neuro – Uy está marcado con un rombo rojo. Otras secuencias de astrovirus neuroinvasivos bovinos están marcadas con rombos negros, las secuencias de astrovirus neuroinvasivos no bovinos están marcadas con triángulos negros, y las secuencias de astrovirus bovinos entéricos están marcadas con círculos blancos. Se muestran especies de mamastrovirus; los asteriscos indican especies que aún no han sido reconocidas por el ICTV. Las cepas dentro del clado CH13 / NeuroS1 que aún no han sido asignadas a una especie por el ICTV se identifican con una línea discontinua. Los valores de arranque se indican en nodos. CH, Suiza; JP, Japón; US, Estados Unidos de América; UY, Uruguay, GB, Reino Unido de Gran Bretaña, DE Alemania; SE, Suecia; FR, Francia; CN, China. BoAstV: astrovirus bovino; OvAstV: astrovirus ovino; PoAstV: astrovirus porcino; HuAstV: astrovirus humano; MiAstV: astrovirus de visón.
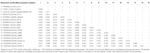
Cuadro 1 Estimates of evolutionary divergence at the amino acid level of the complete ORF2 region between sequences of bovine and ovine Mamastrovirus-13 and strains within the CH13/NeuroS1 clade not yet assigned to a species by the International Committee on Taxonomy of Viruses (ICTV).
Los estudios basados en exámenes neuropatológicos y detección de proteínas y ácidos nucleicos de astrovirus han concluido que existe una relación causal probable entre la infección por astrovirus y las enfermedades y lesiones neurológicas en el ganado bovino (Selimovic-Hamza et al., 2017a; Reuter et al., 2018). Hasta donde sabemos, la encefalitis asociada a astrovirus aún no se ha reproducido experimentalmente. Esto requeriría el aislamiento de los astrovirus neuroinvasivos de los casos clínicos, lo que no se intentó en nuestro caso.
En este caso, no se pudo determinar el origen de la Neuro-Uy de JaCtaV. Sin embargo, se debe considerar el ganado de reserva y la fauna silvestre, ya que el ganado se crió en condiciones extensivas al aire libre. El animal afectado había sido comprado y trasladado a la granja en febrero de 2018 junto con otros 9 novillos. Desafortunadamente, el propietario se negó a realizar más muestreos y pruebas de otros animales en la propiedad, y a realizar una investigación epidemiológica más detallada. Ninguno de los otros animales del grupo había desarrollado enfermedad neurológica en agosto de 2018, la última vez que se contactó con el veterinario. Se ha sugerido una estacionalidad desde el comienzo del invierno hasta el final de la primavera para los casos de encefalitis asociada a astrovirus en Suiza (Selimovic-Hamza et al., 2016). Curiosamente, el caso descrito aquí ocurrió en junio, correspondiente al período de transición otoño-invierno en el hemisferio Sur.
Mientras que los astrovirus neurotrópicos se han identificado en América del Norte(Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) y Asia (Hirashima et al., 2018), su presencia nunca se ha reportado en el hemisferio Sur, por lo que esta comunicación amplía la distribución geográfica de la encefalitis asociada a astrovirus. Para evaluar si la cepa viral detectada en Uruguay podría haberse originado en Europa, América del Norte o Asia, estimamos la tasa evolutiva y realizamos un análisis filogeográfico utilizando secuencias neuroinvasivas de BoAstV disponibles en GenBank. La tasa de evolución estimada utilizando la región de codificación completa fue de 4.27 × 10-4 (95% de densidad de probabilidad más alta-HPD-, 2,19–6,46 × 10-4) sustituciones de nucleótidos/sitio/año, lo que se espera para un virus ARN (Jenkins et al., 2002), pero inferior a la estimada para los astrovirus entéricos humanos (Babkin et al., 2012, 2014). La región ORF1ab mostró una tasa evolutiva similar (4,20 × 10-4, 95% HPD 1,66–6,46 × 10-4 sustituciones/sitio/año) a la región de codificación completa, mientras que la región ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6, 46 × 10-4 sustituciones/sitio/año) y ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 sustituciones / sitio / año) mostraron una tasa evolutiva ligeramente más rápida, y la región genómica de la polimerasa parcial (ORF1b) mostró una tasa evolutiva ligeramente más lenta (5,39 × 10-4, 95% HPD 6,41 × 10-7-1, 10 × 10-3 sustituciones/sitio/año).
Según lo determinado por el análisis filogeográfico con la región codificante completa, y mostrado en el MCTT (Figura 3), hay dos sub-linajes (CH13 y NeuroS1) basados en cepas de referencia, que tienen un ancestro común. El ancestro común más reciente de estos sub-linajes (linaje CH13/NeuroS1) surgió en Europa aproximadamente en 1885 (95% HPD, 1794-1940). A principios de la década de 1900, los dos sub-linajes divergieron, el sub-linaje CH13 permaneció circulando en Europa, mientras que el sub-linaje NeuroS1 se extendió a América y Asia. El escenario más probable es que el sub-linaje NeuroS1 se introdujo en Uruguay desde Europa alrededor del año 1921 (95% de HPD, 1849-1967), presumiblemente a través del comercio de ganado, y luego se extendió a América del Norte y más tarde a Japón (Figura 3). Debido a la limitación en el número de secuencias disponibles en GenBank, que podría haber sesgado el análisis, los resultados obtenidos utilizando la región de codificación completa se compararon con los obtenidos con otras regiones genómicas (ORF1ab, ORF2, ORF1a y ORF1b) disponibles para un mayor número de cepas (es decir, cepas canadienses). En todos los análisis, el escenario más probable es que la introducción del virus en Uruguay se produjera desde Europa (Cifras suplementarias S1A–D). Además, la fecha estimada para esta introducción, obtenida con la región genómica de la polimerasa parcial y ORF1b (Figuras Suplementarias S1A,D), fue similar a la obtenida con la región de codificación completa, mientras que la fecha estimada de introducción obtenida con ORF2 y ORF1a fue anterior pero con un intervalo de HPD del 95% más amplio (Figuras Suplementarias S1B,C). Una introducción del sub-linaje NeuroS1 directamente a Canadá desde Europa, con posterior propagación a Estados Unidos y Japón, también es plausible, como se muestra en la Figura Suplementaria S1D.FIGURA

Gráfico 3 Árbol de máxima credibilidad del clado obtenido mediante el análisis de la región de codificación de longitud completa. El color de las ramas representa el país más probable donde circularon los ancestros, los valores de probabilidad posteriores se muestran en las ramas y los números en cada nodo representan los años de origen para cada clado con el intervalo de HPD del 95%. Los sub-linajes se indican con etiquetas.
Se necesitan más investigaciones para evaluar la distribución geográfica, los mecanismos patogénicos (particularmente los mecanismos de transmisión y entrada), la epidemiología molecular y la posible transmisión entre especies de astrovirus neuroinvasivos.
Disponibilidad de datos
Los conjuntos de datos generados para este estudio se pueden encontrar en GenBank, MK386569.
Las contribuciones de los autores
FG, RDC y MC contribuyeron con la concepción del estudio. FG y RDC realizaron el examen patológico y la toma de muestras. PP realizó la hibridación in situ. FU realizó la inmunohistoquímica. LM, RC y MC realizaron pruebas de virología molecular. MC realizó la secuencia y análisis filogeográficos y figuras asociadas. FG y PP obtuvieron las imágenes histológicas. MF realizó los cultivos bacterianos. FG y MC escribieron el primer borrador del manuscrito. RDC, PP, FU, LM, MF y RC escribieron secciones del manuscrito. Todos los autores contribuyeron a la revisión del manuscrito, leyeron y aprobaron la versión presentada.
Funding
This work was funded by Grants PL-015 N-15156 from INIA and 158 from the «Programa de Iniciación a la Investigación 2017» from «Comisión Sectorial de Investigación Científica» (CSIC). MC and RDC acknowledge support from the «Agencia Nacional de Investigación e Innovación» (ANII) and INIA, respectively, through Ph.D. scholarships. FG acknowledges support from ANII through mobility grant MOV_CA_2018_1_150021.
Declaración de Conflicto de Intereses
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un conflicto de intereses potencial.
El revisor TS declaró una coautoría pasada con uno de los autores PP al editor responsable.
Agradecimientos
Los autores agradecen a Yisell Perdomo y Cecilia Monesiglio del INIA, y a Karen Sverlow y Juliann Beingesser de CAHFS por su asistencia técnica.
Material suplementario
El Material Suplementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
FIGURA S1 / Árboles de credibilidad máxima del clado (MCC) obtenidos mediante el análisis de los ORF1ab de longitud completa (A), ORF2 de longitud completa (B), ORF1a de longitud completa (C) y ORF1b parcial (D). El color de las ramas representa el país más probable donde circularon los ancestros, los valores de probabilidad posteriores se muestran en las ramas, y los números en cada nodo representan los años de origen para cada clado con el intervalo de HPD del 95%. Los sub-linajes se indican con etiquetas.
Notas a pie de página
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., and Sandine, W. E. (1990). Medio altamente selectivo para el aislamiento de Listeria monocytogenes de los alimentos. Appl. Environ. Microbiol. 56, 3154–3157.
Resumen de PubMed | Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chowdhury, S., and Collins, J. K. (1997). Detección específica de desprendimiento y latencia del herpesvirus bovino 1 y 5 mediante una reacción en cadena de la polimerasa anidada. J. Vet. Diagn. Invertir. 9, 387–394. doi: 10.1177/104063879700900408
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., and Tikunova, N. V. (2014). Análisis de recombinación basado en los genomas completos HAstV-2 y HastV-4. Infectar. Genet. Evol. 22, 94–102. doi: 10.1016 / j. meegid.2014.01.010
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V., and Tikunova, N. V. (2012). Alta tasa evolutiva de astrovirus humano. Infectar. Genet. Evol. 12, 435–442. doi: 10.1016 / j. meegid.2012.01.019
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Blomström, A. L., Widén, F., Hammer, A. S., Belák, S., and Berg, M. (2010). Detección de un nuevo astrovirus en tejido cerebral de visón que sufre de síndrome de visón tembloroso mediante el uso de metagenómica viral. J. Clin. Microbiol. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Boros, Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et al. (2017). Brotes de astrovirus neuroinvasivos asociados con encefalomielitis, debilidad y parálisis en cerdos destetados, Hungría. Emerg. Infectar. Dis. 23, 1982–1993. doi: 10.3201 | eid2312.170804
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Boujon, C. L., Koch, M. C., Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Indicación de transmisión entre especies de astrovirus asociada a encefalitis en ovinos y bovinos. Emerg. Infectar. Dis. 23, 1604–1608. doi: 10.3201 / eid2309.170168
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., and Seuberlich, T. (2016). Caracterización molecular basada en el genoma completo de astrovirus bovinos asociados a encefalitis. Infectar. Genet. Evol. 44, 162-168. doi: 10.1016 / J. meegid.06/052/2016
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C., Zurbriggen, A., Vandevelde, M., et al. (2014). Astrovirus neurotrópico en bovinos con encefalitis no supurativa en Europa. J. Clin. Microbiol. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Cantile, C., y Youssef, S. (2016). «Sistema nervioso», en Jubb, Kennedy, and Palmer’s Pathology of Domestic Animals, 6th Edn, ed. M. G. Maxie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T., y Meylan, M. (2017). Signos clínicos neurológicos en bovinos con encefalitis asociada a astrovirus. J. Vet. Pasante. Mediterráneo. 31, 1209–1214. doi: 10.1111 / jvim.14728
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Donato, C., y Vijaykrishna, D. (2017). La amplia gama de huéspedes y la diversidad genética de los astrovirus de mamíferos y aves. Virus 9: E102. doi: 10.3390 / v9050102
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Drummond, A. J., Suchard, M. A., Xie, D., and Rambaut, A. (2012). Filogenética bayesiana con BEAUti y la BESTIA 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev / mss075
Resumen de PubMed / Texto completo de CrossRef | Google Scholar
Giannitti, F., Anderson, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Clamidia pecorum: lesiones fetales y placentarias en abortos caprinos esporádicos. J. Vet. Diagn. Invertir. 28, 184–189. doi: 10.1177/1040638715625729
Resumen de PubMed / Texto Completo de CrossRef | Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Análisis del genoma completo de un nuevo astrovirus bovino neurotrópico detectado en un buey negro japonés con encefalomielitis no supurativa en Japón. Arco. Virol. 163, 2805–2810. doi: 10.1007 / s00705-018-3898-3
Resumen de PubMed / Texto Completo de CrossRef | Google Scholar
Comité Internacional de Taxonomía de Virus (2018). Master Species List (MSL32), Versión 1. Disponible en: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (consultado en marzo de 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G., and Holmes, E. C. (2002). Tasas de evolución molecular en virus de ARN: un análisis filogenético cuantitativo. J. Mol. Evol. 54, 156–165. doi: 10.1007 / s00239-001-0064-3
Resumen de PubMed / Texto Completo de CrossRef | Google Scholar
Kumar, S., Stecher, G., y Tamura, K. (2016). MEGA7: análisis genético evolutivo molecular versión 7.0 para conjuntos de datos más grandes. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093/molbev / msw054
Resumen de PubMed / Texto completo de CrossRef | Google Scholar
Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Astrovirus divergente asociado a enfermedades neurológicas en bovinos. Emerg. Infectar. Dis. 19, 1385–1392. doi: 10.3201 | eid1909.130682
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Genomas de larga duración del virus de inmunodeficiencia humana tipo 1 de seroconvertidores infectados por el subtipo C en la India, con pruebas de recombinación de intersubtipos. J. Virol. 73, 152–160.
Resumen de PubMed | Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et al. (2015). Diagnóstico de infección por astrovirus neuroinvasivo en un adulto inmunocomprometido con encefalitis mediante secuenciación imparcial de próxima generación. Clin. Infectar. Dis. 60, 919–923. doi: 10.1093/cid | ciu912
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Hallazgos patológicos e inmunohistoquímicos de la infección por el virus del Nilo Occidental en psitaciformes. Veterinario. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Un nuevo astrovirus asociado con encefalitis y ganglioneuritis en ovejas domésticas. Transbound. Emerg. Dis. 64, 677–682. doi: 10.1111 / tbed.12623
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Encefalitis por astrovirus en niños con agammaglobulinemia ligada al cromosoma X. Emerg. Infectar. Dis. 16, 918–925. doi: 10.3201 | eid1606.091536
Resumen de PubMed | Texto completo de CrossRef / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L., and Pybus, O. G. (2016). Explorando la estructura temporal de secuencias heterocronas usando TempEst (formely Path-O-Gen). Virus Evol. 2: vew007. doi: 10.1093/ve / vew007
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Reuter, G., Pankovics, P., and Boros, A. (2018). Meningoencefalomielitis no supurativa (aséptica) asociada a infecciones por astrovirulentos por astrovirus en humanos y animales. Clin. Microbiol. Apo. 31, e00040-18. doi: 10.1128 / CMR.00040-18
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Beer, M., et al. (2016). Detección de un nuevo astrovirus bovino en una vaca con encefalitis. Transbound. Emerg. Dis. 63, 253–259. doi: 10.1111 / tbed.12493
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A., and Seuberlich, T. (2017a). Frecuencia y fenotipo patológico de la infección por astrovirus bovino CH13 / NeuroS1 en bovinos con enfermedades neurológicas: hacia la evaluación de la causalidad. Virus 9: E12. doi: 10.3390 / v9010012
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., and Seuberlich, T. (2017b). Infección por astrovirus bovino en ganado de engorde con enfermedad neurológica en el oeste de Canadá. Puede. Veterinario. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., y Seuberlich, T. (2016). Detección de astrovirus en casos históricos de encefalitis bovina esporádica europea, Suiza 1958-1976. Delantero. Veterinario. Sci. 3:91. doi: 10.3389 / fvets.2016.00091
Resumen de PubMed | Texto Completo de CrossRef / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identificación de un segundo astrovirus asociado a encefalitis en bovinos. Emerg. Los Microbios Infectan. 5: e5. doi: 10.1038 / emi.2016.5
Resumen de PubMed / Texto Completo de CrossRef / Google Scholar
Spinato, M. T., Vince, A., Cai, H., and Ojkic, D. (2017). Identificación de astrovirus bovino en casos de encefalitis no supurativa bovina en el este de Canadá. Puede. Veterinario. J. 58, 607-609.
Google Scholar
Stein, L. T., Rech, R. R., Harrison, L., and Brown, C. C. (2010). Estudio inmunohistoquímico del virus de la rabia en el sistema nervioso central de especies domésticas y silvestres. Veterinario. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
Resumen de PubMed / Texto Completo de CrossRef | Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A., and Minh, B. Q. (2016). W-IQ-TREE: una herramienta filogenética en línea rápida para el análisis de máxima verosimilitud. Ácidos nucleicos Res. 44, W232-W235. doi: 10.1093/nar / gkw256
Resumen de PubMed / Texto completo de CrossRef / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Redescubrimiento y caracterización genómica de astrovirus bovinos. J. Gén. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0.030817-0
Resumen de PubMed | Texto completo Cruzado / Google Scholar