Creative Proteomics Blog
aminohappojen sekvenssi proteiinissa tai peptidissä voidaan tunnistaa Edman degradation-menetelmällä, jonka kehitti Pehr Edman. Tällä menetelmällä voidaan merkitä ja pilkkoa peptidi N-terminaalista häiritsemättä muiden aminohappojäämien välisiä peptidisidoksia. Edman-hajoamisreaktion automatisoivat vuonna 1967 Edman ja Beggs. Nykyään käytetään laajalti automatisoitua Edman-hajoamista (proteiinisekvensaattori), ja se voi sekvensoida peptidejä jopa 50 aminohappoon.
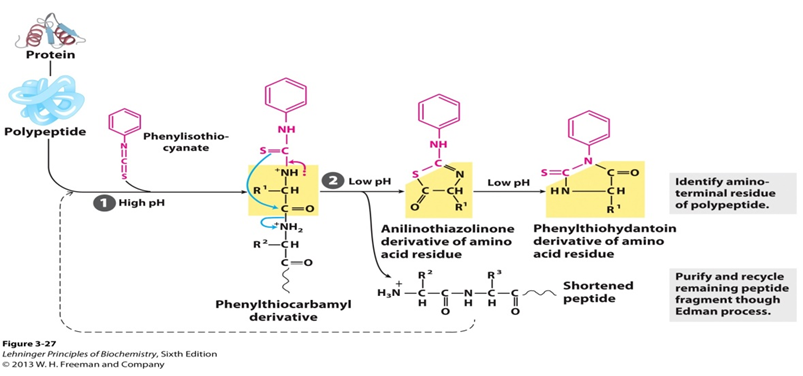
peptidien syklinen hajoaminen, joka perustuu fenyyli-isotiosyanaatin reaktioon n-terminaalisen jäännöksen vapaan aminoryhmän kanssa siten, että aminohapot poistetaan yksi kerrallaan ja tunnistetaan niiden fenyylitiohydantoiinijohdannaisiksi. Spesifisessä prosessissa varaamaton peptidi reagoidaan fenyyli-isotiosyanaatin (PITC) kanssa aminoterminaalissa lievästi emäksisissä olosuhteissa, jolloin saadaan fenyylitiokarbamoyylijohdannainen (PTC-peptidi). Tämän jälkeen johdannaisen tiokarbonyylirikki hyökkää happamissa olosuhteissa N-terminaalisen aminohapon karbonyylihiilen kimppuun. Ensimmäinen aminohappo pilkkoutuu anilinotiatsolinonijohdannaiseksi (ATZ-aminohapoksi) ja loput peptidistä voidaan eristää ja alistua seuraavaan hajoamissykliin. Muodostuttuaan tämä tiatsolonijohdannainen on stabiilimpi kuin fenyylitiokarbamyylijohdannainen. Tämän jälkeen ATZ-aminohappo poistetaan uuttamalla etyyliakteraatilla ja muutetaan fenyylitiohydantoiinijohdannaiseksi (PTH-aminohapoksi). Kromatografian avulla voidaan tunnistaa kussakin syklissä syntyvä PTH-jäännös.
automatisoidussa Edman-hajoamisessa proteiinit voidaan analysoida levittämällä ne In-liuokseen TFA-suodattimeen ja lastaamalla ne sitten Edman-sekvensointilaitteeseen. Seosten proteiinit erotetaan ensin 1D-tai 2D-geeleillä ja pyyhitään sitten PVDF-kalvolle. Proteiinit havaitaan Coomassie blue -, Amido black-tai Poncau s-värjäyksellä ja kiinnostavat proteiinit leikataan pois ja PVDF-kalvopala Ladataan Edman-sekvensseriin.
kun massaspektrometria kehitettiin, Edman-hajoamissekvenssin käyttö alkoi vähentyä. Kuitenkin se stills on edelleen menetelmiä useita erilaisia proteiinin rakenneanalyysin sovelluksia. Sitä voidaan käyttää rekombinanttiproteiinien n-terminaalisen rajan todentamiseen tai proteaasiresistenttien domeenien N-terminaalin määrittämiseen erityisesti silloin, kun proteiini tai domeeni on >40-80 kDa tai sitä ei voida helposti puhdistaa. Sen avulla voidaan myös tunnistaa Uusi N-terminaalinen ja proteolyyttinen pilkkoutumiskohta proteiinikappaleissa. Lisäksi analysoinnissa voidaan käyttää edmanin hajoamista joidenkin uusien proteiinien ja peptidien osalta, joissa sekvenssitietokantoja ei ole saatavilla MS/MS-tietokannan hakuun.
proteiinin N-terminaalinen aminohappo voidaan pilkkoa pois. Näin prosessissa ensimmäinen sykli siis tunnistaa tarkan N-terminaalisen aminohapon. Koska vapautuneet aminohapot tunnistetaan ja kvantifioidaan kromatografialla, voidaan tunnistaa myös molekyylipainoltaan identtiset aminohapot. Esimerkiksi isoleusiinin ja leusiinin massa on 113 Da, mutta niillä on erilainen retentioaika. Lisäksi Edman-sekvensointi voidaan suorittaa PVDF-bloteille 1D-ja 2D-geeleistä, mikä mahdollistaa seoksen proteiinien n-terminaalisen sekvensoinnin. Edman-hajoamissekvenssiä ei kuitenkaan ole saatavilla, kun peptidi, jonka N-terminus on kemiallisesti muunnettu, kuten asetylaatio. Ja koska PITC ei pysty reagoimaan muun kuin α-aminohapon kanssa, sekvensointi lakkaa, jos kohdataan Ei-α-aminohappo kuten isoaspartiihappo. Suurempia proteiineja ei myöskään voida sekvensoida Edman-sekvensoinnilla.
Creative Proteomicsilla ammattimainen proteiinisekvenssialustamme tarjoaa Edman degradation or mass spectrometry (MS) n-terminaalisen sekvenssianalyysin, jolla on toisiaan täydentäviä etuja. Asiantuntijatiimimme, jolla on laaja kokemus, voi auttaa sinua ymmärtämään, mitä yrität tutkia ja täyttämään vaatimuksesi. Lisäksi voimme tarjota muita proteiinin tunnistuspalveluja, kuten:
- Peptidimassa fingerprinting (PMF) service
- peptidien tai proteiinien Sekvenssianalyysi
- de novo protein sequence analysis service
- Shotgun protein identification service
- Membrane proteomics service