Biologie pour les Majors I
Résultats d’apprentissage
- Comprendre la différence entre le pré-ARNm et l’ARNm
Après la transcription, les pré-ARNM eucaryotes doivent subir plusieurs étapes de traitement avant de pouvoir être traduits. Les ARNT et ARNR eucaryotes (et procaryotes) subissent également un traitement avant de pouvoir fonctionner comme composants de la machinerie de synthèse des protéines.
Traitement de l’ARNm
Le pré-ARNm eucaryote subit un traitement intensif avant d’être prêt à être traduit. Les étapes supplémentaires impliquées dans la maturation de l’ARNm eucaryote créent une molécule avec une demi-vie beaucoup plus longue qu’un ARNm procaryote. Les ARNM eucaryotes durent plusieurs heures, alors que l’ARNm typique d’E. coli ne dure pas plus de cinq secondes.
Les ARNM pré-sont d’abord enrobés de protéines stabilisatrices d’ARN; celles-ci protègent les ARNm pré-de la dégradation pendant qu’ils sont traités et exportés hors du noyau. Les trois étapes les plus importantes du traitement pré-ARNm sont l’ajout de facteurs de stabilisation et de signalisation aux extrémités 5′ et 3′ de la molécule, et l’élimination des séquences intermédiaires qui ne spécifient pas les acides aminés appropriés. Dans de rares cas, la transcription de l’ARNm peut être « modifiée » après sa transcription.
Capsulage en 5′
Pendant que le pré-ARNm est encore en cours de synthèse, un capuchon en 7-méthylguanosine est ajouté à l’extrémité 5′ du transcrit de croissance par une liaison phosphate. Cette fraction (groupe fonctionnel) protège l’ARNm naissant de la dégradation. De plus, les facteurs impliqués dans la synthèse des protéines reconnaissent le cap pour aider à initier la traduction par les ribosomes.
Queue Poly-A 3′
Une fois l’élongation terminée, le pré-ARNm est clivé par une endonucléase entre une séquence consensus AAUAAA et une séquence riche en GU, laissant la séquence AAUAAA sur le pré-ARNm. Une enzyme appelée poly-A polymérase ajoute ensuite une chaîne d’environ 200 résidus A, appelée queue poly-A. Cette modification protège en outre le pré-ARNm de la dégradation et signale l’exportation des facteurs cellulaires dont le transcription a besoin vers le cytoplasme.
Épissage de pré-ARNm
Les gènes eucaryotes sont composés d’exons, qui correspondent à des séquences codantes pour les protéines (ex-on signifie qu’elles sont exprimées), et de séquences intermédiaires appelées introns (intron désigne leur rôle intervenant), qui peuvent être impliquées dans la régulation des gènes mais sont retirées du pré-ARNm pendant le traitement. Les séquences intron dans l’ARNm ne codent pas pour des protéines fonctionnelles.
La découverte d’introns a surpris les chercheurs dans les années 1970 qui s’attendaient à ce que les ARNM pré-spécifient des séquences protéiques sans traitement supplémentaire, comme ils l’avaient observé chez les procaryotes. Les gènes des eucaryotes supérieurs contiennent très souvent un ou plusieurs introns. Ces régions peuvent correspondre à des séquences régulatrices; cependant, la signification biologique d’avoir de nombreux introns ou d’avoir de très longs introns dans un gène n’est pas claire. Il est possible que les introns ralentissent l’expression des gènes car il faut plus de temps pour transcrire les pré-ARNM avec beaucoup d’introns. Alternativement, les introns peuvent être des restes de séquence non fonctionnels laissés par la fusion d’anciens gènes tout au long de l’évolution. Ceci est corroboré par le fait que des exons séparés codent souvent des sous-unités ou des domaines protéiques distincts. Pour la plupart, les séquences d’introns peuvent être mutées sans affecter finalement le produit protéique.
Tous les introns d’un pré-ARNm doivent être complètement et précisément éliminés avant la synthèse des protéines. Si le processus se trompe ne serait-ce qu’avec un seul nucléotide, le cadre de lecture des exons rejoints se déplacerait et la protéine résultante serait dysfonctionnelle. Le processus d’élimination des introns et de reconnexion des exons est appelé épissage (figure 1). Les introns sont éliminés et dégradés alors que le pré-ARNm est encore dans le noyau. L’épissage se produit par un mécanisme spécifique à la séquence qui garantit que les introns seront éliminés et que les exons seront rejoints avec la précision et la précision d’un seul nucléotide. L’épissage des ARNM pré-est réalisé par des complexes de protéines et de molécules d’ARN appelés splicéosomes.
Question pratique
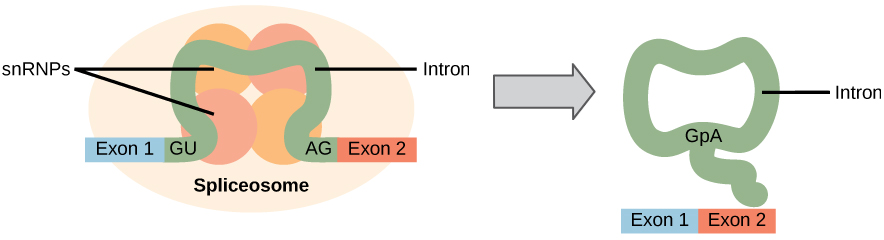
Figure 1. L’épissage pré-ARNm implique l’élimination précise des introns du transcrit d’ARN primaire. Le processus d’épissage est catalysé par des complexes protéiques appelés splicéosomes qui sont composés de protéines et de molécules d’ARN appelées SNRNA. Les splicéosomes reconnaissent des séquences à l’extrémité 5′ et 3′ de l’intron.
Les erreurs d’épissage sont impliquées dans les cancers et autres maladies humaines. Quels types de mutations pourraient conduire à des erreurs d’épissage?
Notez que plus de 70 introns individuels peuvent être présents, et chacun doit subir le processus d’épissage — en plus du capsulage de 5 ‘ et de l’ajout d’une queue poly-A — juste pour générer une seule molécule d’ARNm traduisible.
Modification de l’ARN dans les Trypanosomes
Figure 2. Trypanosoma brucei est l’agent causal de la maladie du sommeil chez l’homme. Les ARNM de cet agent pathogène doivent être modifiés par l’ajout de nucléotides avant que la synthèse des protéines puisse se produire. (crédit: modification de l’œuvre de Torsten Ochsenreiter)
Les trypanosomes sont un groupe de protozoaires qui comprend l’agent pathogène Trypanosoma brucei, qui provoque la maladie du sommeil chez l’homme (figure 2). Les trypanosomes, et pratiquement tous les autres eucaryotes, ont des organites appelés mitochondries qui fournissent à la cellule de l’énergie chimique. Les mitochondries sont des organites qui expriment leur propre ADN et sont considérés comme les restes d’une relation symbiotique entre un eucaryote et un procaryote englouti. L’ADN mitochondrial des trypanosomes présente une exception intéressante au Dogme central: leurs pré-ARNM ne disposent pas des informations correctes pour spécifier une protéine fonctionnelle. Habituellement, c’est parce que l’ARNm manque de plusieurs nucléotides U. La cellule effectue une étape supplémentaire de traitement de l’ARN appelée édition de l’ARN pour y remédier.
D’autres gènes du génome mitochondrial codent des ARN guides de 40 à 80 nucléotides. Une ou plusieurs de ces molécules interagissent par appariement de bases complémentaires avec certains des nucléotides du transcrit pré-ARNm. Cependant, l’ARN guide a plus de nucléotides A que le pré-ARNm n’a de nucléotides U avec lesquels se lier. Dans ces régions, l’ARN guide se boucle. Les extrémités 3′ des ARN guides ont une longue queue poly-U, et ces bases U sont insérées dans des régions du transcrit pré-ARNm au niveau desquelles les ARN guides sont en boucle. Ce processus est entièrement médié par des molécules d’ARN. C’est—à—dire que les ARN guides – plutôt que les protéines – servent de catalyseurs dans l’édition d’ARN.
L’édition d’ARN n’est pas seulement un phénomène de trypanosomes. Dans les mitochondries de certaines plantes, presque tous les ARNM pré-sont modifiés. L’édition d’ARN a également été identifiée chez des mammifères tels que les rats, les lapins et même les humains. Quelle pourrait être la raison évolutive de cette étape supplémentaire dans le traitement pré-ARNm? Une possibilité est que les mitochondries, restes d’anciens procaryotes, possèdent une méthode tout aussi ancienne basée sur l’ARN pour réguler l’expression des gènes. À l’appui de cette hypothèse, les modifications apportées aux ARNM antérieurs diffèrent selon les conditions cellulaires. Bien que spéculatif, le processus d’édition de l’ARN peut être un vestige d’un moment primordial où les molécules d’ARN, au lieu des protéines, étaient responsables de la catalyse des réactions.
Essayez-Le
Contribuez !
Améliorer cette pagen Savoir plus