Biologie pour les Majors II
Résultats d’apprentissage
- Comprendre les systèmes de classification passés et émergents pour les virus
Parce que la plupart des virus ont probablement évolué à partir d’ancêtres différents, les méthodes systématiques utilisées par les scientifiques pour classer les cellules procaryotes et eucaryotes ne sont pas très utiles. Si les virus représentent des « restes » d’organismes différents, même l’analyse génomique ou protéique n’est pas utile. Pourquoi?, Parce que les virus n’ont pas de séquence génomique commune qu’ils partagent tous. Par exemple, la séquence d’ARNr 16S si utile pour construire des phylogénies procaryotes n’est d’aucune utilité pour une créature sans ribosomes ! Les biologistes ont utilisé plusieurs systèmes de classification dans le passé. Les virus étaient initialement regroupés par morphologie partagée. Plus tard, les groupes de virus ont été classés par le type d’acide nucléique qu’ils contenaient, ADN ou ARN, et si leur acide nucléique était simple ou double brin. Cependant, ces méthodes de classification antérieures regroupaient les virus différemment, car elles étaient basées sur différents ensembles de caractères du virus. La méthode de classification la plus couramment utilisée aujourd’hui s’appelle le schéma de classification de Baltimore et est basée sur la façon dont l’ARN messager (ARNm) est généré dans chaque type particulier de virus.
Les systèmes de classification antérieurs
Les virus ne contiennent que quelques éléments permettant de les classer : le génome viral, le type de capside et la structure de l’enveloppe des virus enveloppés. Tous ces éléments ont été utilisés dans le passé pour la classification virale (tableau 1 et figure 1). Les génomes viraux peuvent varier en fonction du type de matériel génétique (ADN ou ARN) et de son organisation (simple ou double brin, linéaire ou circulaire, segmenté ou non segmenté). Chez certains virus, des protéines supplémentaires nécessaires à la réplication sont directement associées au génome ou contenues dans la capside virale.
| Tableau 1. Classification des Virus par Structure Génomique et Noyau | |
|---|---|
| Classifications de base | Exemples |
| ARN | Virus de la rage, rétrovirus |
| ADN | Herpèsvirus, virus de la variole |
| Virus de la rage simple brin | , rétrovirus |
| Double brin | Herpèsvirus, virus de la variole |
| Linear | Virus de la rage, rétrovirus, herpèsvirus, virus de la variole |
| Circulaire | Papillomavirus, nombreux bactériophages |
| Non segmenté: le génome est constitué d’un seul segment de matériel génétique | Virus Parainfluenza |
| Segmenté: le génome est divisé en plusieurs segments | Virus de la grippe |
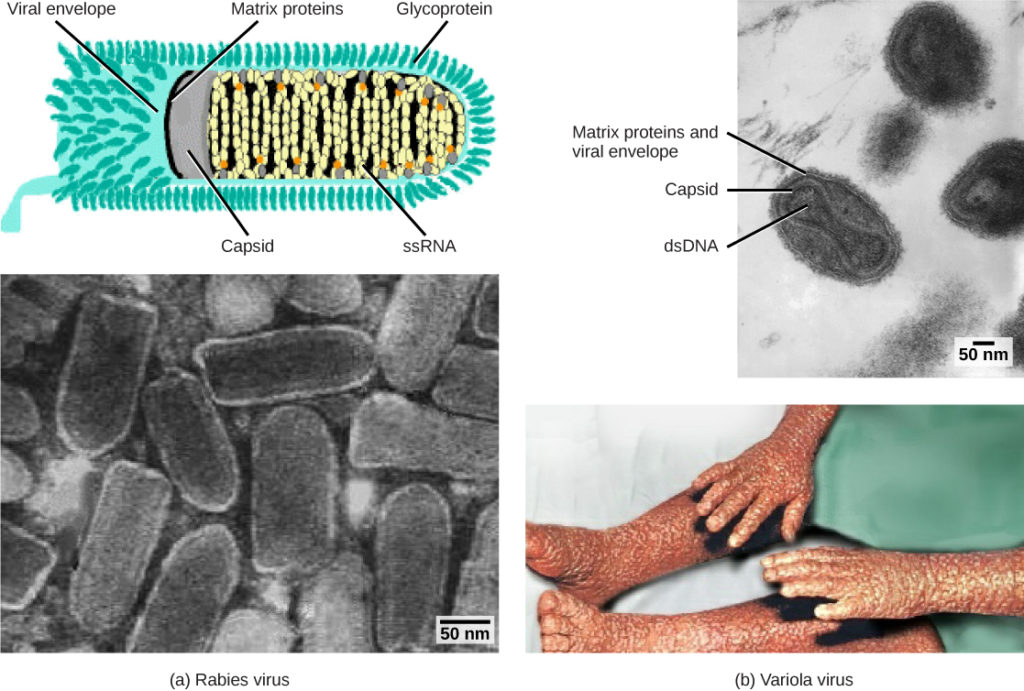
Figure 1. Les virus sont classés en fonction de leur matériel génétique de base et de la conception de leur capside. (a) Le virus de la rage a un noyau d’ARN simple brin (ARNSR) et une capside hélicoïdale enveloppée, tandis que (b) le virus de la variole, l’agent responsable de la variole, a un noyau d’ADN double brin (ADNDSD) et une capside complexe. (crédit « diagramme de la rage »: modification des travaux du CDC; « micrographie de la rage »: modification des travaux du Dr Fred Murphy, CDC; crédit « micrographie de la variole »: modification des travaux du Dr Fred Murphy, Sylvia Whitfield, CDC; crédit « photo de la variole »: modification des travaux du CDC; données de la barre d’échelle de Matt Russell)
Les virus peuvent également être classés selon la conception de leurs capsides (tableau 2 et figure 2). Les capsides sont classés comme icosaédriques nus, icosaédriques enveloppés, hélicoïdaux enveloppés, hélicoïdaux nus et complexes. Le type de matériel génétique (ADN ou ARN) et sa structure (simple ou double brin, linéaire ou circulaire, segmentée ou non segmentée) sont utilisés pour classer les structures du noyau du virus (tableau 2).
| Tableau 2. Classification des Virus par Structure de Capside | |
|---|---|
| Classification des capsides | Exemples |
| Icosaédrique nu | Virus de l’hépatite A, poliovirus |
| Icosaédrique enveloppé | Virus d’Epstein-Barr, virus de l’herpès simplex, virus de la rubéole, virus de la fièvre jaune, VIH-1 |
| Virus de la grippe, virus des oreillons, virus de la rougeole, virus de la rage, virus hélicoïdaux enveloppés | |
| Virus de la mosaïque du tabac en hélice nue | |
| Complexe avec de nombreuses protéines; certains présentent des combinaisons de structures de capside icosaédriques et hélicoïdales | Herpèsvirus, virus de la variole, virus de l’hépatite B, bactériophage T4 |

Figure 2. Les micrographies électroniques de transmission de divers virus montrent leurs structures. La capside du (a) virus de la poliomyélite est icosaédrique nue; (b) la capside du virus d’Epstein-Barr est icosaédrique enveloppée; (c) la capside du virus des oreillons est une hélice enveloppée; (d) la capside du virus de la mosaïque du tabac est hélicoïdale nue; et (e) la capside de l’herpèsvirus est complexe. (crédit a: modification du travail par le Dr Fred Murphy, Sylvia Whitfield; crédit b: modification du travail par Liza Gross; crédit c: modification du travail par le Dr F. A. Murphy, CDC; crédit d: modification du travail par l’USDA ARS; crédit e: modification des travaux de Linda Stannard, Département de microbiologie médicale, Université du Cap, Afrique du Sud, NASA; données sur les barres d’échelle de Matt Russell)
Classification de Baltimore
Le système de classification des virus le plus couramment utilisé a été développé par le biologiste lauréat du prix Nobel David Baltimore au début des années 1970.Outre les différences de morphologie et de génétique mentionnées ci-dessus, le système de classification de Baltimore regroupe les virus en fonction de la production de l’ARNm au cours du cycle réplicatif du virus.
Les virus du groupe I contiennent de l’ADN double brin (ADNc) comme génome. Leur ARNm est produit par transcription de la même manière qu’avec l’ADN cellulaire.
Les virus du groupe II ont comme génome de l’ADN simple brin (adNSS). Ils convertissent leurs génomes monocaténaires en un intermédiaire d’ADND avant que la transcription en ARNm puisse se produire.
Les virus du groupe III utilisent l’ARND comme génome. Les brins se séparent et l’un d’eux sert de matrice pour la génération d’ARNm à l’aide de l’ARN polymérase ARN-dépendante codée par le virus.
Les virus du groupe IV ont un ARNSS comme génome avec une polarité positive. La polarité positive signifie que l’ARN génomique peut servir directement d’ARNm. Les intermédiaires de l’ARND, appelés intermédiaires réplicatifs, sont fabriqués dans le processus de copie de l’ARN génomique. De multiples brins d’ARN pleine longueur de polarité négative (complémentaires à l’ARN génomique brin positif) sont formés à partir de ces intermédiaires, qui peuvent ensuite servir de modèles pour la production d’ARN à polarité positive, y compris à la fois des ARN génomiques pleine longueur et des ARNM viraux plus courts.
Les virus du groupe V contiennent des génomes d’arNSS avec une polarité négative, ce qui signifie que leur séquence est complémentaire de l’ARNm. Comme pour les virus du groupe IV, les intermédiaires de l’ARND sont utilisés pour faire des copies du génome et produire l’ARNm. Dans ce cas, le génome à brin négatif peut être converti directement en ARNm. De plus, des brins d’ARN positifs de pleine longueur sont conçus pour servir de modèles pour la production du génome à brin négatif.
Les virus du groupe VI ont des génomes d’arNSS diploïdes (deux copies) qui doivent être convertis, à l’aide de l’enzyme transcriptase inverse, en ADNc; l’ADNc est ensuite transporté vers le noyau de la cellule hôte et inséré dans le génome hôte. Ensuite, l’ARNm peut être produit par transcription de l’ADN viral qui a été intégré dans le génome de l’hôte.
Les virus du groupe VII ont des génomes d’adNDD partiels et font des intermédiaires d’arNSS qui agissent comme ARNm, mais sont également reconvertis en génomes d’adNDD par transcriptase inverse, nécessaire à la réplication du génome. Les caractéristiques de chaque groupe dans la classification de Baltimore sont résumées dans le tableau 3 avec des exemples de chaque groupe.
| Tableau 3. Classification de Baltimore | |||
|---|---|---|---|
| Groupe | Caractéristiques | Mode de production d’ARNm | Exemple |
| I | ADN double brin | L’ARNm est transcrit directement à partir du modèle d’ADN | Herpès simplex (herpèsvirus) |
| II | ADN simple brin | L’ADN est converti en forme double brin avant que l’ARN ne soit transcrit | Parvovirus canin (parvovirus) |
| III | ARN double brin | L’ARNm est transcrit à partir de le génome de l’ARN | Gastro-entérite infantile (rotavirus) |
| IV | ARN simple brin (+) | Fonctions du génome comme ARNm | Rhume (pircornavirus) |
| V | ARN simple brin (−) | L’ARNm est transcrit à partir du génome de l’ARN | Rage (rhabdovirus) |
| VI | Virus à ARN simple brin avec transcriptase inverse | La transcriptase inverse produit de l’ADN à partir du génome de l’ARN; L’ADN est ensuite incorporé dans le génome de l’hôte; L’ARNm est transcrit à partir de l’ADN incorporé | Virus de l’immunodéficience humaine (VIH) |
| VII | Virus à ADN double brin avec transcriptase inverse | Le génome viral est de l’ADN double brin, mais l’ADN viral est répliqué par un intermédiaire d’ARN ; l’ARN peut servir directement d’ARNm ou de modèle pour fabriquer l’ARNm | Virus de l’hépatite B (hepadnavirus) |