Blog de protéomique créatif
La séquence des acides aminés dans une protéine ou un peptide peut être identifiée par la dégradation d’Edman, qui a été développée par Pehr Edman. Cette méthode peut marquer et cliver le peptide de N-terminal sans perturber les liaisons peptidiques entre d’autres résidus d’acides aminés. La réaction de dégradation d’Edman a été automatisée en 1967 par Edman et Beggs. De nos jours, la dégradation automatisée d’Edman (le séquénateur de protéines) est largement utilisée et peut séquencer des peptides jusqu’à 50 acides aminés.
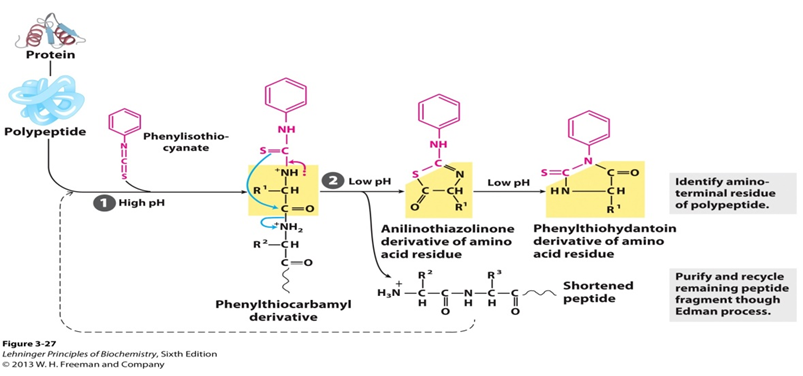
Dégradation cyclique des peptides basée sur la réaction du phénylisothiocyanate avec le groupe amino libre du résidu N-terminal de telle sorte que les acides aminés sont éliminés un à la fois et identifiés comme leurs dérivés de phénylthiohydantoïne. En ce qui concerne le processus spécifique, un peptide non chargé est mis à réagir avec le phénylisothiocyanate (PITC) à l’extrémité aminée dans des conditions légèrement alcalines pour donner un dérivé de phénylthiocarbamoyle (peptide PTC). Ensuite, dans des conditions acides, le soufre thiocarbonyle du dérivé attaque le carbone carbonyle de l’acide aminé N-terminal. Le premier acide aminé est clivé sous forme de dérivé d’anilinothiazolinone (acide aminé ATZ) et le reste du peptide peut être isolé et soumis au cycle de dégradation suivant. Une fois formé, ce dérivé de thiazolone est plus stable que le dérivé de phénylthiocarbamyle. L’acide aminé ATZ est ensuite éliminé par extraction à l’acérate d’éthyle et transformé en un dérivé de phénylthiohydantoïne (acide aminé PTH). Et la chromatographie peut être utilisée pour identifier le résidu de PTH généré par chaque cycle.
En ce qui concerne la dégradation automatisée d’Edman, les protéines peuvent être analysées en les appliquant en solution sur un filtre TFA, puis chargées sur l’instrument de séquençage d’Edman. Les protéines des mélanges sont d’abord séparées par des gels 1D ou 2D, puis épongées sur une membrane PVDF. Les protéines sont détectées par coloration au bleu de Coomassie, au noir d’Amido ou au Poncau S et les protéines d’intérêt sont découpées et la pièce de membrane PVDF chargée sur le séquenceur Edman.
Avec le développement de la spectrométrie de masse, l’utilisation du séquençage de dégradation d’Edman a commencé à diminuer. Cependant, il reste encore les méthodes pour plusieurs types d’applications d’analyse structurale des protéines. Il peut être utilisé pour vérifier la limite N-terminale des protéines recombinantes ou déterminer la N-terminale des domaines résistants à la protéase, en particulier lorsque la protéine ou le domaine est > de 40 à 80 kDa ou ne peut pas être facilement purifié. Il peut également être utilisé pour identifier le nouveau site de clivage N-terminal et protéolytique dans les fragments de protéines. De plus, en ce qui concerne certaines protéines et peptides nouveaux pour lesquels les bases de données de séquences ne sont pas disponibles pour la recherche de bases de données MS/ MS, la dégradation d’Edman peut être utilisée pour l’analyse.
L’acide aminé N-terminal de la protéine peut être clivé. Ainsi, dans le procédé, le premier cycle identifie ainsi l’acide aminé N-terminal exact. De plus, du fait que les acides aminés libérés sont identifiés et quantifiés par chromatographie, les acides aminés de poids moléculaire identique peuvent être identifiés. Par exemple, l’isoleucine et la leucine ont une masse de 113 Da, mais leur temps de rétention est différent. De plus, le séquençage Edman peut être effectué sur des blots PVDF à partir de gels 1D et 2D, ce qui permet le séquençage N-terminal des protéines du mélange. Cependant, le séquençage de dégradation d’Edman ne sera pas disponible lorsque le peptide dont l’extrémité N-terminale a été modifiée chimiquement, telle que l’acétylation. Et comme le PITC ne peut pas réagir avec un acide non α-aminé, le séquençage s’arrêtera si un acide non α-aminé est rencontré comme l’acide isoaspartique. De plus, les protéines plus grosses ne peuvent pas être séquencées par le séquençage d’Edman.
Chez Creative Proteomics, notre plateforme professionnelle de séquençage des protéines fournit une analyse de séquence N-terminale par dégradation d’Edman ou spectrométrie de masse (MS), avec des avantages complémentaires les uns aux autres. Notre équipe d’experts avec une vaste expérience peut vous aider à comprendre ce que vous essayez d’enquêter et à répondre à vos exigences. De plus, nous pouvons fournir d’autres services d’identification des protéines, notamment:
- Service de prise d’empreintes de masse peptidique (PMF)
- Analyse de séquences de peptides ou de protéines
- Service d’analyse de séquences de protéines De novo
- Service d’identification de protéines Shotgun
- Service de protéomique membranaire