Frontiers in Microbiology
Introduction
La famille des Astroviridae contient des virus à ARN simple brin non enveloppés, à sens positif, dans deux genres, le Mamastrovirus et l’Avastrovirus, qui infectent respectivement les mammifères et les oiseaux. Actuellement, le Comité International de Taxonomie des Virus (Comité International de Taxonomie des Virus, 2018) reconnaît 19 espèces, à savoir Mamastrovirus-1 à -19, au sein du genre Mamastrovirus; cependant, il existe de nombreuses souches en attente de classification, dont certaines sont provisoirement considérées comme de nouvelles espèces (Donato et Vijaykrishna, 2017).
Depuis 2010, plusieurs astrovirus sont de plus en plus reconnus comme neuroinvasifs chez diverses espèces de mammifères, y compris les humains (Quan et al., 2010; Naccache et coll., 2015), le vison (Blomström et al., 2010), bovins (Li et al., 2013), des moutons (Pfaff et al., 2017), et les porcs (Boros et al., 2017). Après la reconnaissance initiale de l’encéphalite bovine associée à l’astrovirus chez les bovins américains (Li et al., 2013), une étude rétrospective de cas d’encéphalite bovine sporadique d’étiologie indéterminée en Suisse a révélé que cet astrovirus neuroinvasif était passé inaperçu pendant des décennies (Selimovic-Hamza et al., 2016). Bien que l’épidémiologie et les voies de transmission de ces astrovirus soient inconnues, une transmission inter-espèces a été suggérée en fonction du niveau élevé d’identité (> 98%), partagé entre les astrovirus neuroinvasifs bovins et ovins aux niveaux de nucléotides et d’acides aminés (Boujon et al., 2017).
Astrovirus bovins (BoAstVs), nommés BoAstV-NeuroS1 (Li et al., 2013) et BoAstV-CH13 (Bouzalas et al., 2014), ont été initialement trouvés dans le cerveau de bovins atteints d’encéphalite non suppurée aux États-Unis et en Suisse, respectivement. Malgré la nomenclature différente, les deux virus représentent la même espèce de génotype (Bouzalas et al., 2016; Selimovic-Hamza et coll., 2017a) qui est toujours en attente de classification officielle par l’ICTV. En 2015, une souche BoAstV jusqu’alors inconnue, nommée BoAstV-CH15, a été identifiée dans le cerveau de vaches atteintes d’encéphalite en Suisse. La comparaison phylogénétique du génome complet a révélé une relation plus étroite entre le BoAstV-CH15 et un astrovirus ovin (OvAstV) qu’avec le BoAstV-CH13 (Seuberlich et al., 2016). Une coinfection avec BoAstV-CH13 et BoAstV-CH15 a également été documentée dans un cas (Seuberlich et al., 2016). La même année en Allemagne, Schlottau et al. (2016) ont signalé un nouvel astrovirus, à savoir BoAstV-BH89/14, chez une vache atteinte d’encéphalite, qui était le plus étroitement lié à OvAstV et BoAstV-CH15. Par la suite, BoAstV-CH13/NeuroS1 a été identifié en 2017 dans des cas d’encéphalite bovine dans l’est et l’Ouest du Canada (Spinato et al., 2017; Selimovic-Hamza et coll., 2017b). En 2018, un nouveau BoAstV neuroinvasif étroitement lié au BoAstV-NeuroS1 nord-américain et européen/BoAstV-CH13 a été identifié dans un bouvier atteint d’encéphalomyélite non suppurée au Japon, et la survenue d’une recombinaison intra-génotypique entre les souches nord-américaines et européennes a été suggérée (Hirashima et al., 2018).
Bien que des cas d’encéphalite associée à un astrovirus aient été signalés en Amérique du Nord, en Europe et en Asie, leur présence n’a jamais été documentée dans l’hémisphère sud. Nous décrivons ici un cas d’encéphalite associée à un astrovirus chez des bovins en Uruguay, qui élargit la distribution géographique et la diversité génétique des astrovirus neuroinvasifs et fournit des preuves phylogéographiques qui suggèrent que ce virus a été introduit dans les Amériques depuis l’Europe et s’est ensuite propagé à l’Asie.
Matériaux et méthodes
Histoire et signalisation
En juin 2018, un bouvier Holstein âgé de 22 mois dans un groupe de 37 bouvillons dans une ferme de 300 hectares à Colonia, en Uruguay, a développé des signes neurologiques progressifs comprenant un comportement inhabituel, une marche sans but, des cercles, une ataxie, des mouvements répétitifs et non coordonnés de la langue et une position couchée. Le troupeau broutait sur un pâturage annuel d’avoine et était complété par de l’ensilage de maïs. Un diagnostic clinique présomptif de listériose cérébrale par le vétérinaire a incité un traitement à la pénicilline et à la streptomycine, mais l’animal est décédé spontanément après une évolution clinique de 3 jours.
Examen pathologique, Hybridation in situ (ISH) et Immunohistochimie (IHC)
La tête du bœuf a été retirée de la carcasse et soumise au Laboratoire de Diagnostic Vétérinaire de l’INIA (Plateforme de Santé Animale) pour un travail de diagnostic. La moitié du cerveau, un court segment de la moelle épinière cervicale proximale (C1), le ganglion trijumeau et la racine du nerf trijumeau, la glande salivaire, le ganglion lymphatique rétropharyngé, l’oropharynx, l’œsophage, la langue et le muscle squelettique, ont été fixés par immersion dans du formol tamponné neutre à 10% pendant 48 à 72 h. Les tissus ont été traités de manière routinière pour l’histologie, incorporés dans de la paraffine, coupés en microtomes à 4-5 µm et colorés avec de l’hématoxyline et de l’éosine (H & E) et taches de gramme.
L’ISH chromogène a été réalisée manuellement sur des sections de 5 µm du tronc cérébral, du cerveau et du cervelet fixés au formol et incorporés à la paraffine (FFPE) sur des lames Superfrost Plus (Thermo Fisher Scientific, Pittsburgh, PA, États-Unis) à l’aide du kit de dosage RNAscope 2.5 Red (Cat #322360, Advanced Cell Diagnostics, Hayward, CA, États-Unis) et de la sonde BoAstV Cat. #406921. La sonde est composée de 20 paires ZZ ciblant la région 5232-6180 du virus (GenBank KF233994.1). Chaque section de tissu de 5 µm a été prétraitée avec de la chaleur et de la protéase avant l’hybridation de la sonde pendant 2 h à 40 ° C et traitée conformément aux recommandations du fabricant. Les contrôles négatifs utilisés pour la validation du signal comprenaient une sonde non liée (correspondant au contenu en GC) exécutée sur des sections en série et sondant des tissus d’animaux non infectés. Les lames ont été contre-colorées à l’hématoxyline et montées avec EcoMount (Biocare Medical, Concord, CA, États-Unis).
De plus, l’IHC a été réalisée dans des sections FFPE du tronc cérébral, du cerveau et du cervelet, comme décrit précédemment, pour l’identification du virus du Nil occidental (VNO, Flavivirus) (Palmieri et al., 2011), le virus de la rage (Lyssavirus) (Stein et al., 2010), et Chlamydia spp. (Giannitti et coll., 2016) antigènes.
Virologie moléculaire
L’extraction d’acide nucléique a été réalisée à partir d’un échantillon groupé de cerveau congelé (-20 ° C) à l’aide du kit d’isolation d’acide nucléique MagMAX® (Thermo Fisher Scientific). Pour la détection des astrovirus, la transcription inverse (RT) a été réalisée avec la Transcriptase inverse RevertAid® (Thermo Fisher Scientific) et des amorces hexamères aléatoires (Qiagen). La PCR a été réalisée à partir d’ADNc à l’aide de MangoMix® (Bioline) et d’amorces qui amplifient un fragment de 432 nucléotides du gène de la polymérase de l’astrovirus (Tse et al., 2011). Le produit de PCR a été visualisé dans un gel d’agarose à 2%, purifié à l’aide du Kit combiné d’Extraction rapide de Gel et de purification par PCR PureLink® (Invitrogen), et séquencé chez Macrogen Inc. (Séoul, Corée du Sud). Pour l’amplification du génome entier des astrovirus, Maxima H Moins la Transcriptase inverse (Thermo Fisher Scientific) et oligo (dT) 18 pour l’obtention de l’ADNc, et MangoMix® (Bioline) ou l’ADN Polymérase Ranger (Bioline) avec des amorces décrites par Hirashima et al., 2018, ont été utilisés. Les produits PCR ont été visualisés dans un gel d’agarose à 1-2%, purifiés et séquencés comme mentionné ci-dessus. L’assemblage des séquences a été réalisé avec SeqMan (Lasergene 8, DNASTAR). Vingt-six séquences complètes du génome d’astrovirus neuroinvasif provenant de bovins, de moutons, de porcs, d’humains et de visons et d’astrovirus bovin entérique disponibles dans GenBank ont été téléchargées et alignées à l’aide du logiciel Clustal W dans MEGA 7 (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) a été utilisé pour déterminer le modèle d’évolution de séquence le mieux adapté (SYM + I + G4) et pour construire un arbre phylogénétique à maximum de vraisemblance avec les séquences presque complètes du BoAstV détectées dans ce cas, et ces séquences complètes téléchargées depuis GenBank, en utilisant bootstrap comme méthode statistique pour évaluer la robustesse des clades. Le tracé de similarité a été réalisé avec le logiciel SimPlot (Lole et al., 1999). Les distances P au niveau des acides aminés de l’ORF2 ont été estimées avec le logiciel MEGA 7 (Kumar et al., 2016).
De plus, une analyse phylogéographique bayésienne a été réalisée avec le paquet BEAST v1.8.4 (Drummond et al., 2012), en utilisant: la région codante complète de la lignée BoAstV CH13 / NeuroS1, ORF1ab (gènes non structuraux), ORF2 (gène structural), ORF1a (protéase) et une ORF1b partielle (région génomique de la polymérase, pour laquelle des souches canadiennes étaient disponibles), avec toutes les séquences disponibles dans GenBank (dernière adhésion le 18 avril 2019), afin de déterminer le taux d’évolution, les âges / années des ancêtres communs et la voie de circulation virale la plus probable par pays (Suisse, Uruguay, États-Unis, Canada et Japon ). L’absence de recombinaison dans l’ensemble de données a été déterminée à l’aide du Programme de détection de recombinaison 4. Le modèle de substitution qui correspond le mieux à chaque alignement a été déterminé à l’aide du logiciel MEGA 7 à l’aide de valeurs du critère d’information bayésien (BIC), et la structure temporelle de chaque ensemble de données a été évaluée à l’aide de TempEst (Rambaut et al., 2016). L’horloge moléculaire détendue lognormale avec analyse de l’horizon bayésien a été sélectionnée par le facteur Bayes parmi les différentes combinaisons d’horloges moléculaires et de prieurs d’arbres coalescents utilisées. Le pays de détection a été utilisé comme trait. La longueur Monte-Carlo de la chaîne de Markov était de 100 millions de générations, assurant la convergence de l’analyse, évaluée dans Tracer v1.6.0, et la probabilité postérieure a été utilisée pour évaluer les clades. L’arbre de crédibilité maximale du clade (ECC) a été obtenu à l’aide du logiciel TreeAnnotator de BEAST et visualisé dans FigTree v1.4.3.
Enfin, l’ADN extrait du cerveau congelé a été traité par PCR pour la détection des herpèsvirus bovins 1 et 5 (BHV-1 et-5), comme décrit précédemment (Ashbaugh et al., 1997).
Bactériologie
Des échantillons frais de cerveau et de tronc cérébral ont été systématiquement traités pour des cultures bactériennes aérobies dans des géloses de sang et de MacConkey, et une culture sélective pour Listeria monocytogenes (Al-Zoreky et Sandine, 1990).
Résultats et discussion
Les signes cliniques et les résultats épidémiologiques dans le cas décrit ici, bien que non spécifiques, étaient similaires à ceux décrits dans d’autres cas d’encéphalite associée à un astrovirus bovin, généralement décrite comme sporadique (Selimovic-Hamza et al., 2016), avec une variété de déficits neurologiques (Deiss et al., 2017), avec une durée des signes cliniques qui varie généralement de 1 jour à 3 semaines (Schlottau et al., 2016; Deiss et coll., 2017; Spinato et coll., 2017; Hirashima et coll., 2018).
L’examen macroscopique du cerveau, du segment C1 de la moelle épinière et d’autres tissus de la tête n’a pas révélé de lésions anatomiques grossières significatives. Histologiquement, il y avait une méningo-encéphalomyélite modérée à sévère, lymphocytaire, histiocytaire et plasmocytaire affectant le télencéphale (y compris l’hémisphère cérébral et l’hippocampe), le tronc cérébral et le seul segment de la moelle épinière examiné. Les lésions étaient principalement réparties dans la matière grise et les zones limitantes de la substance blanche. Dans les zones touchées, il y avait des menottes périvasculaires et une inflammation lymphoplasmocytaire et histiocytaire et une nécrose neuronale / neuronophagie avec gliose dans le neuropile adjacent. Il y avait une satellitose de neurones nécrotiques affectés (Figures 1A-D). Les lésions étaient beaucoup moins fréquentes et sévères dans le parenchyme cérébelleux, bien qu’il y ait eu une leptoméningite cérébelleuse modérée multifocale. Aucune bactérie intralésionnelle n’a été trouvée avec des taches H &E et Gram. Aucun changement histologique significatif n’a été constaté dans les autres tissus examinés.

Figure 1. Lésions histologiques du tronc cérébral (A, B) et du cortex cérébral (C, D) et détection de l’ARN BoAstV dans le cortex cérébral (E, G). Les images (A-D) sont des sections de cerveau colorées avec H& E; les images E et G sont des coupes de cortex cérébral démontrant une hybridation par ISH chromogène à l’aide de la sonde spécifique de BoAstV, contre-colorée à l’hématoxyline ; l’image F est une section série de la sonde DapB du cortex cérébral (témoin négatif), contre-colorée à l’hématoxyline. (A) Un espace périvénulaire est nettement élargi par des cellules inflammatoires (principalement des lymphocytes et des histiocytes) qui s’infiltrent également dans le neuropile adjacent. (B) Le neurone au centre présente un périkaryon et une caryorrhexie hyperéosinophiles (nécrose) et le corps neuronal est entouré d’un nombre accru de cellules gliales (satellitose) et inflammatoires. (C) Le leptoméninge est infiltré par les lymphocytes et les histiocytes. (D) Une région du cortex cérébral avec de multiples neurones hyperéosinophiles (nécrotiques) et un gros vaisseau avec des manchettes lymphocytaires périvasculaires. (E) Dans une section sérielle du cortex cérébral, le marquage abondant de l’ARN BoAstV intracytoplasmique est représenté par un fort dépôt granulaire de chromogène rouge dans le cytoplasme neuronal du soma et des extensions neuronales (E, G), qui n’est pas présent lors de l’hybridation à l’aide de la sonde témoin négative (F).
Une infection virale neuroinvasive a été suspectée lors d’un examen histologique du système nerveux central. Les bovins atteints d’encéphalite sont préoccupants car de nombreux neuropathogènes chez les ruminants sont zoonotiques (Cantile et Youssef, 2016); ainsi, un diagnostic d’encéphalite devrait inciter à des tests de laboratoire approfondis pour dépister les agents infectieux lorsque cela est possible. Dans le cas décrit ici, les CSH pour le VNO, le virus de la rage et la Chlamydia spp., et la PCR pour le BHV-1 et -5 étaient tous négatifs, et aucune bactérie pathogène n’a été cultivée à partir du tissu cérébral. Comme le bœuf avait < 2 ans et qu’aucune modification spongiforme n’a été observée dans le tronc cérébral, l’animal n’a pas été testé pour l’encéphalopathie spongiforme bovine (ESB), une maladie exotique des bovins adultes qui n’a jamais été signalée en Uruguay. De plus, l’ESB n’est pas inflammatoire (Cantile et Youssef, 2016).
L’hybridation in situ a été réalisée à l’aide d’une sonde générée à partir de BoAstV-NeuroS1, et il y avait une hybridation de sonde abondante à l’intérieur et limitée au cytoplasme des neurones de l’hémisphère cérébral et de l’hippocampe (Figures 1E–G). Dans ces zones, l’hybridation de la sonde s’est colocalisée avec des neurones nécrotiques et des régions de gliose, sans hybridation de la sonde détectable dans les cellules gliales, ou les cellules inflammatoires des poignets périvasculaires. Aucun acide nucléique viral n’a été détecté par l’ISH dans le cervelet, qui ne présentait que des lésions inflammatoires minimes dans le parenchyme mais une leptoméningite modérée, ou le tronc cérébral, y compris des sections avec une inflammation sévère. Cela signifie que, topographiquement, la détection de la distribution virale par ISH était plus limitée que l’encéphalite dans les sections examinées, ce qui a parfois été décrit dans des cas d’encéphalite associée à BoAstV-CH13 / NeuroS1 chez les bovins (Selimovic-Hamza et al., 2017a, b). Une raison de ce manque occasionnel de détection de l’ARN viral dans les zones lésées du cerveau pourrait être la limite de détection de l’ISH, ou la clairance du virus dans les zones enflammées du cerveau au moment de la mort, comme suggéré précédemment (Selimovic-Hamza et al., 2017b). Comme prévu, aucune hybridation de sonde n’a été détectée par ISH dans le tissu cérébral utilisé comme témoin négatif.
L’astrovirus a été détecté dans le cerveau par RT-PCR. L’analyse de la séquence génomique presque complète a révélé une souche de Mamastrovirus au sein du clade CH13/NeuroS1, que nous avons nommée BoAstV-Neuro-Uy, la séquence a été déposée à GenBank sous le numéro d’accession MK386569. L’analyse phylogénétique a révélé une proximité avec d’autres astrovirus neuroinvasifs au sein du clade Virginie/Homme-Vison-Ovin (VA/HMO) (Figure 2), qui contient la plupart des astrovirus neuroinvasifs connus (Hirashima et al., 2018; Reuter et coll., 2018). La séquence presque complète de BoAstV-Neuro-Uy a une longueur de 6427 pb et une identité de séquence de 94% avec la souche KagoshimaSR28-462. BoAstV-Neuro-Uy présente des caractéristiques similaires à celles d’autres souches de la lignée CH13/NeuroS1: une région 5’UTR de 51 nt, ORF1a (protéase) de 861 acides aminés (aa), ORF1b de 523 aa (ARN polymérase ARN-dépendante) et ORF2 de 758 aa (protéine de capside). Malheureusement, le 3’UTR n’a pas pu être séquencé, mais une queue poly(A) est présumée être présente car oligo(dT) 18 a été utilisé pour obtenir l’ADNc. De plus, la séquence heptamérique AAAAAAC, un signal ribosomique à décalage d’images, est présente. Les distances P au niveau des acides aminés de l’ORF2 ont confirmé l’assignation de cette souche au clade CH13/NeuroS1. P – distances < 0.35 entre le BoAstV-Neuro-Uy et d’autres membres de ce clade (tableau 1) soutiendrait une classification de ces souches virales au sein d’une même espèce; Le Mamastrovirus-13 a été récemment proposé par d’autres auteurs (Donato et Vijaykrishna, 2017; Hirashima et al., 2018), bien que l’attribution définitive d’espèces par l’ICTV soit en attente. La sonde utilisée pour l’ISH, générée à partir de BoAstV-NeuroS1, avait une identité de séquence de 92,7% avec BoAstV-Neuro-Uy.
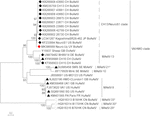
Figure 2. Analyse phylogénétique des séquences nucléotidiques complètes par la méthode du maximum de vraisemblance. BoAstV-Neuro-Uy est marqué d’un losange rouge. D’autres séquences d’astrovirus neuroinvasifs bovins sont marquées de losanges noirs, les séquences d’astrovirus neuroinvasifs non bovins sont marquées de triangles noirs et les séquences d’astrovirus bovins entériques sont marquées de cercles blancs. Les espèces de mamastrovirus sont indiquées; les astérisques indiquent les espèces qui n’ont pas encore été reconnues par l’ICTV. Les souches du clade CH13/NeuroS1 qui n’ont pas encore reçu d’espèce par l’ICTV sont identifiées par une ligne pointillée. Les valeurs d’amorçage sont indiquées dans les nœuds. CH, Suisse; JP, Japon; É.-U., États-Unis d’Amérique; UY, Uruguay; GB, Royaume-Uni de Grande-Bretagne; DE, Allemagne; SE, Suède; FR, France; CN, Chine. BoAstV, astrovirus bovin; OvAstV, astrovirus ovin; PoAstV, astrovirus porcin; HuAstV, astrovirus humain; MiAstV, astrovirus du vison.
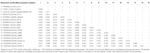
Tableau 1. Estimations de la divergence évolutive au niveau des acides aminés de la région ORF2 complète entre les séquences de Mamastrovirus bovin et ovin-13 et les souches du clade CH13/NeuroS1 non encore attribuées à une espèce par le Comité International de taxonomie des virus (ICTV).
Des études basées sur des examens neuropathologiques et la détection d’acides nucléiques et de protéines d’astrovirus ont conclu à l’existence d’une relation de cause à effet probable entre l’infection à astrovirus et les maladies neurologiques et les lésions chez les bovins (Selimovic-Hamza et al., 2017a; Reuter et coll., 2018). À notre connaissance, l’encéphalite associée à un astrovirus n’a pas encore été reproduite expérimentalement. Cela nécessiterait l’isolement des astrovirus neuroinvasifs des cas cliniques, ce qui n’a pas été tenté dans notre cas.
La source de BoAstV-Neuro-Uy dans ce cas n’a pas pu être déterminée. Cependant, les bovins de réservoir et la faune doivent être pris en compte, car les bovins ont été élevés dans des conditions extérieures étendues. L’animal affecté avait été acheté et déplacé à la ferme en février 2018 avec 9 autres bouvillons. Malheureusement, le propriétaire a refusé un échantillonnage et des tests supplémentaires sur d’autres animaux de la propriété, ainsi qu’une enquête épidémiologique plus détaillée. Aucun des autres animaux du groupe n’avait développé de maladie neurologique en août 2018, la dernière fois que le vétérinaire a été contacté. Une saisonnalité entre le début de l’hiver et la fin du printemps a été suggérée pour les cas d’encéphalite associée à un astrovirus en Suisse (Selimovic-Hamza et al., 2016). Fait intéressant, le cas décrit ici s’est produit en juin, correspondant à la période de transition automne-hiver dans l’hémisphère sud.
Alors que des astrovirus neurotropes ont été identifiés en Amérique du Nord (Li et al., 2013; Spinato et coll., 2017), Europe (Bouzalas et al., 2014) et en Asie (Hirashima et al., 2018), leur présence n’ayant jamais été signalée dans l’hémisphère sud, cette communication élargit donc la répartition géographique de l’encéphalite associée aux astrovirus. Pour évaluer si la souche virale détectée en Uruguay pourrait provenir d’Europe, d’Amérique du Nord ou d’Asie, nous avons estimé le taux d’évolution et effectué une analyse phylogéographique en utilisant des séquences neuroinvasives de BoAstV disponibles dans GenBank. Le taux d’évolution estimé à l’aide de la région de codage complète était de 4.27 × 10-4 (densité de probabilité la plus élevée de 95% – HPD-, 2,19–6,46 × 10-4) substitutions nucléotidiques / site /année, ce qui est attendu pour un virus à ARN (Jenkins et al., 2002), mais inférieure à celle estimée pour les astrovirus humains entériques (Babkin et al., 2012, 2014). La région ORF1ab a montré un taux d’évolution similaire (4,20 × 10-4, 95% HPD 1,66–6,46 × 10-4 substitutions / site/ année) à la région codante complète, tandis que l’ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6,46 × 10-4 substitutions/ site/ année) et ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 substitutions / site / année) ont montré un taux d’évolution légèrement plus rapide, et la région génomique partielle de la polymérase (ORF1b) a montré un taux d’évolution légèrement plus lent (5,39 × 10-4, 95% HPD 6,41 × 10-7-1,10 × 10-3 substitutions / site / année).
Tel que déterminé par l’analyse phylogéographique avec la région codante complète, et montré dans le MCTT (Figure 3), il existe deux sous-lignées (CH13 et NeuroS1) basées sur des souches de référence, qui ont un ancêtre commun. L’ancêtre commun le plus récent de ces sous-lignées (lignée CH13/NeuroS1) est apparu en Europe vers 1885 (95% de HPD, 1794-1940). Au début des années 1900, les deux sous-lignées ont divergé, la sous-lignée CH13 est restée en circulation en Europe, tandis que la sous-lignée NeuroS1 s’est étendue à l’Amérique et à l’Asie. Le scénario le plus probable est que la sous-lignée NeuroS1 a été introduite en Uruguay en provenance d’Europe vers l’année 1921 (95% de HPD, 1849-1967), probablement par le commerce du bétail, puis s’est répandue en Amérique du Nord, puis au Japon (Figure 3). En raison de la limitation du nombre de séquences disponibles dans GenBank, ce qui aurait pu biaiser l’analyse, les résultats obtenus en utilisant la région codante complète ont été comparés à ceux obtenus avec d’autres régions génomiques (ORF1ab, ORF2, ORF1a et ORF1b) disponibles pour un plus grand nombre de souches (c.-à-d. des souches canadiennes). Dans toutes les analyses, le scénario le plus probable est que l’introduction du virus en Uruguay a eu lieu depuis l’Europe (Figures supplémentaires S1A–D). De plus, la date estimée de cette introduction, obtenue avec l’ORF1ab et la région génomique partielle de la polymérase (ORF1b) (Figures supplémentaires S1A, D), était similaire à celle obtenue avec la région codante complète, alors que la date estimée d’introduction obtenue avec ORF2 et ORF1a était antérieure mais avec un intervalle HPD plus large de 95% (Figures supplémentaires S1B, C). Une introduction de la sous-lignée NeuroS1 directement au Canada à partir de l’Europe, suivie d’une propagation aux États-Unis et au Japon, est également plausible, comme le montre la figure supplémentaire S1D.

Figure 3. Arbre de crédibilité maximum du clade obtenu par analyse de la région de codage pleine longueur. La couleur des branches représente le pays le plus probable où les ancêtres ont circulé, les valeurs de probabilité postérieures sont affichées dans les branches et les nombres dans chaque nœud représentent les années d’origine pour chaque clade avec l’intervalle HPD à 95%. Les sous-lignées sont indiquées par des étiquettes.
D’autres études sont nécessaires pour évaluer la répartition géographique, les mécanismes pathogènes (en particulier les mécanismes de transmission et d’entrée), l’épidémiologie moléculaire et la transmission interspécifique potentielle des astrovirus neuroinvasifs.
Disponibilité des données
Les ensembles de données générés pour cette étude se trouvent dans GenBank, MK386569.
Contributions des auteurs
FG, RDC et MC ont contribué à la conception de l’étude. FG et RDC ont effectué l’examen pathologique et l’échantillonnage. PP a effectué l’hybridation in situ. FU a effectué l’immunohistochimie. LM, RC et MC ont effectué des tests de virologie moléculaire. MC a effectué les analyses de séquence et phylogéographiques et les figures associées. FG et PP ont obtenu les images histologiques. MF a effectué les cultures bactériennes. FG et MC ont écrit la première ébauche du manuscrit. RDC, PP, FU, LM, MF et RC ont écrit des sections du manuscrit. Tous les auteurs ont contribué à la révision du manuscrit, ont lu et approuvé la version soumise.
Financement
Ce travail a été financé par les subventions PL-015 N-15156 de l’INIA et 158 du « Programa de Iniciación a la Investigación 2017 » de la « Comisión Sectorial de Investigación Científica » (CSIC). MC et RDC reconnaissent le soutien de l' » Agence Nationale pour la Recherche et l’Innovation » (ANII) et de l’INIA, respectivement, par le biais de bourses de doctorat. FG reconnaît le soutien de l’ANII par le biais de la subvention de mobilité MOV_CA_2018_1_150021.
Déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de relations commerciales ou financières pouvant être interprétées comme un conflit d’intérêts potentiel.
Le réviseur TS a déclaré une co-paternité passée avec l’un des auteurs PP à l’éditeur de gestion.
Remerciements
Les auteurs remercient Yisell Perdomo et Cecilia Monesiglio de l’INIA, ainsi que Karen Sverlow et Juliann Beingesser du CAHFS pour leur assistance technique.
Matériel supplémentaire
Le matériel supplémentaire pour cet article peut être trouvé en ligne à l’adresse suivante:: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
FIGURE S1 / Arbres de crédibilité maximale du clade (ECMC) obtenus par analyse de l’ORF1ab pleine longueur (A), de l’ORF2 pleine longueur (B), de l’ORF1a pleine longueur (C) et de l’ORF1b partielle (D). La couleur des branches représente le pays le plus probable où les ancêtres ont circulé, les valeurs de probabilité postérieures sont affichées dans les branches, et les nombres dans chaque nœud représentent les années d’origine pour chaque clade avec l’intervalle HPD à 95%. Les sous-lignées sont indiquées par des étiquettes.
Notes de bas de page
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., et Sandine, W. E. (1990). Milieu hautement sélectif pour l’isolement de Listeria monocytogenes des aliments. Appl. Environ. Microbiol. 56, 3154–3157.
Résumé PubMed / Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, C. P., Chowdhury, S. et Collins, J. K. (1997). Détection spécifique de l’excrétion et de la latence de l’herpèsvirus bovin 1 et 5 à l’aide d’une réaction en chaîne par polymérase imbriquée. J. Vétérinaire. Diagn. Investir. 9, 387–394. doi: 10.1177/104063879700900408
Résumé PubMed / Texte intégral Croisé / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., et Tikunova, N. V. (2014). Analyse de recombinaison basée sur les génomes complets HAstV-2 et HastV-4. Infecter. Genet. Evol. 22, 94–102. doi: 10.1016 / j.meegid.2014.01.010
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V. et Tikunova, N. V. (2012). Taux d’évolution élevé de l’astrovirus humain. Infecter. Genet. Evol. 12, 435–442. doi: 10.1016 / j.meegid.2012.01.019
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Il s’agit de l’un des plus grands ouvrages de l’histoire de l’art et de l’histoire de l’Art. Détection d’un nouvel astrovirus dans le tissu cérébral d’un vison souffrant du syndrome du vison secouant par métagénomique virale. J. Clin. Microbiol. 48, 4392–4396. doi: 10.1128/ JCM.01040-10
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Boros, Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et coll. (2017). Outbreaks of neuroinvasive astrovirus associated with encephalomyelitis, weakness, and paralysis among sevaned pigs, Hungary. Emerg. Infecter. Dis. 23, 1982–1993. doi: 10.3201/eid2312.170804
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Boujon, C. L., Koch, M. C., Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Indication of cross-species transmission of astrovirus associated with encephalitis in sheep and cattle. Emerg. Infecter. Dis. 23, 1604–1608. doi: 10.3201/ eid2309.170168
PubMed Abstract / CrossRef Full Text / Google Scholar
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., and Seuberlich, T. (2016). Full-génomes based molecular characterization of encéphalite-associated bovine astroviruses. Infect. Genet. Evol. 44, 162-168. doi: 10.1016/j. meegid.052/06/2016
PubMed Abstract / CrossRef Full Text / Google Scholar
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C., Zurbriggen, A., Vandevelde, M., et al. (2014). Astrovirus neurotropique chez les bovins atteints d’encéphalite non suppurative en Europe. J. Clin. Microbiol. 52, 3318–3324. doi: 10.1128/ JCM.01195-14
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Cantile, C., et Youssef, S. (2016). » Nervous System « , dans Jubb, Kennedy et Palmer’s Pathology of Domestic Animals, 6e éd., éd. Il s’agit de la première édition de la série.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T., et Meylan, M. (2017). Signes cliniques neurologiques chez les bovins atteints d’encéphalite associée à un astrovirus. J. Vétérinaire. Stagiaire. Med. 31, 1209–1214. doi: 10.1111/ jvim.14728
Résumé PubMed / CrossRef Texte intégral / Google Scholar
Donato, C., et Vijaykrishna, D. (2017). La vaste gamme d’hôtes et la diversité génétique des astrovirus mammifères et aviaires. Virus 9: E102. doi: 10.3390/v9050102
Résumé PubMed / Texte intégral Croisé / Google Scholar
Drummond, A. J., Suchard, M. A., Xie, D., et Rambaut, A. (2012). Phylogénétique bayésienne avec BEAUti et la BÊTE 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev/mss075
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Giannitti, F., Anderson, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Chlamydia pecorum: lésions fœtales et placentaires lors d’un avortement caprin sporadique. J. Vétérinaire. Diagn. Investir. 28, 184–189. doi: 10.1177/1040638715625729
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Analyse du génome entier d’un nouvel astrovirus bovin neurotrope détecté chez un bœuf noir japonais atteint d’encéphalomyélite non suppurée au Japon. Arch. Virol. 163, 2805–2810. doi: 10.1007/s00705-018-3898-3
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Comité International de Taxonomie des virus (2018). Liste principale des espèces (MSL32), Version 1. Disponible au : https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (consulté en mars 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G. et Holmes, E. C. (2002). Taux d’évolution moléculaire chez les virus à ARN: une analyse phylogénétique quantitative. J. Mol. Evol. 54, 156–165. doi: 10.1007/s00239-001-0064-3
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Kumar, S., Stecher, G. et Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis version 7.0 pour des ensembles de données plus volumineux. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093/molbev/msw054
Résumé PubMed / Texte intégral Croisé / Google Scholar
Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Astrovirus divergent associé à une maladie neurologique chez les bovins. Emerg. Infecter. Dis. 19, 1385–1392. doi: 10.3201/eid1909.130682
Résumé PubMed / Texte intégral Croisé / Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S.s., Novak, N. G., et al. (1999). Génomes complets du virus de l’immunodéficience humaine de type 1 provenant de séroconvertisseurs infectés par le sous-type C en Inde, avec des preuves de recombinaison intersubtype. J. Virol. 73, 152–160.
Résumé PubMed / Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et coll. (2015). Diagnostic d’une infection à astrovirus neuroinvasive chez un adulte immunodéprimé atteint d’encéphalite par séquençage impartial de nouvelle génération. Clin. Infecter. Dis. 60, 919–923. doi: 10.1093/cid/ciu912
Résumé PubMed / Texte intégral Croisé / Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Pathology and immunohistochemical findings of West Nile virus infection in psittaciformes. Vétérinaire. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
Résumé PubMed / Texte intégral Croisé / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Un nouvel astrovirus associé à l’encéphalite et à la ganglionévrite chez les moutons domestiques. Transbound. Emerg. Dis. 64, 677–682. doi: 10.1111/ tbed.12623
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Il s’agit de l’un des principaux facteurs de risque de la maladie. (2010). Encéphalite à astrovirus chez un garçon atteint d’agammaglobulinémie liée à l’X. Emerg. Infecter. Dis. 16, 918–925. doi: 10.3201/eid1606.091536
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L., et Pybus, O. G. (2016). Exploration de la structure temporelle de séquences hétérochrones à l’aide de TempEst (anciennement Path-O-Gen). Virus Evol. 2: vew007. doi: 10.1093/ve/vew007
Résumé publié / Texte intégral croisé / Google Scholar
Reuter, G., Pankovics, P., et Boros, A. (2018). Méningo-encéphalomyélite non suppurative (aseptique) associée à des infections à astrovirus neurovirulentes chez l’homme et l’animal. Clin. Microbiol. Rév.31, e00040-18. doi: 10.1128/CMR.00040-18
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Beer, M., et al. (2016). Détection d’un nouvel astrovirus bovin chez une vache atteinte d’encéphalite. Transbound. Emerg. Dis. 63, 253–259. doi: 10.1111/ tbed.12493
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A. et Seuberlich, T. (2017a). Fréquence et phénotype pathologique de l’infection par l’astrovirus bovin CH13 / NeuroS1 chez les bovins atteints de maladies neurologiques: vers une évaluation de la causalité. Virus 9: E12. doi: 10.3390/v9010012
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., et Seuberlich, T. (2017b). Bovine astrovirus infection in feedlot cattle with neurological disease in Western Canada. Peut. Vétérinaire. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., et Seuberlich, T. (2016). Détection de l’astrovirus dans des cas historiques d’encéphalite bovine sporadique européenne, Suisse 1958-1976. Devant. Vétérinaire. Sci. 3:91. doi: 10.3389/fvets.2016.00091
Résumé PubMed / Texte intégral Croisé / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identification d’un deuxième astrovirus associé à l’encéphalite chez les bovins. Emerg. Les Microbes Infectent. 5:e5. doi: 10.1038/emi.2016.5
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Spinato, M. T., Vince, A., Cai, H. et Ojkic, D. (2017). Identification de l’astrovirus bovin dans les cas d’encéphalite bovine non suppurée dans l’Est du Canada. Peut. Vétérinaire. J. 58, 607-609.
Google Scholar
Stein, L. T., Rech, R. R., Harrison, L. et Brown, C. C. (2010). Étude immunohistochimique du virus de la rage dans le système nerveux central des espèces domestiques et sauvages. Vétérinaire. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
Résumé PubMed / Texte intégral CrossRef / Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A., et Minh, B. Q. (2016). W-IQ-TREE: un outil phylogénétique en ligne rapide pour l’analyse du maximum de vraisemblance. Acides nucléiques Res. 44, W232-W235. doi: 10.1093/nar/gkw256
Résumé publié / Texte intégral croisé / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Redécouverte et caractérisation génomique des astrovirus bovins. J. Gen. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099/vir.0.030817-0
Résumé PubMed / Texte intégral croisé / Google Scholar