La Structure moléculaire Révèle le fonctionnement de l’Antibiotique Streptomycine | Salle de presse BNL
La structure moléculaire Révèle le fonctionnement de l’antibiotique Streptomycine
5 septembre 2013
La streptomycine a été le premier antibiotique développé pour traiter la tuberculose, mais jusqu’à récemment, les scientifiques ne comprenaient pas complètement son fonctionnement au niveau moléculaire. Ils savaient que la streptomycine bloquait un processus critique, la synthèse des protéines par les ribosomes conduisant à la mort cellulaire bactérienne, mais certains détails de l’interaction restaient inconnus. À la source de lumière Synchrotron nationale du Laboratoire national de Brookhaven, les chercheurs ont utilisé la cristallographie aux rayons X pour compléter le tableau.
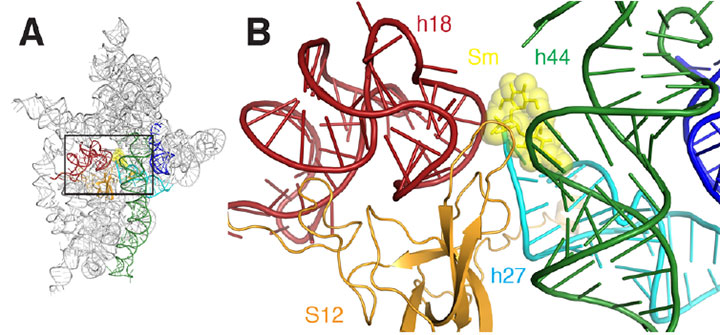
A) Un diagramme en ruban du site de liaison à la streptomycine du ribosome. B) Un gros plan de la zone rectangulaire décrite dans A. La streptomycine est représentée par des sHcks et des sphères jaunes, les hélices sont colorées en rouge, vert foncé, cyan, orange et bleu.
La streptomycine fait partie d’une famille d’antibiotiques qui agissent en interrompant la fonction des ribosomes des cellules bactériennes, les machines moléculaires complexes qui créent des protéines en liant les acides aminés entre eux. Les ribosomes, une cible majeure pour les antibiotiques qui agissent en inhibant la synthèse des protéines, ont deux parties principales ou « sous-unités ». »
La plus grande sous-unité effectue la construction des protéines, guidée par un type d’ARN appelé ARN messager (ARNm), qui s’y lie. La petite sous-unité « lit » l’ARNm et sélectionne la molécule d’ARN de transfert correspondante (ARNt), qui sélectionne et délivre l’acide aminé suivant au ribosome. C’est là que la streptomycine joue un rôle. Il se lie près de la petite sous-unité, ce qui l’amène à mal lire la séquence. Il en résulte la synthèse de protéines aléatoires, qui tue finalement les bactéries. Mais la façon dont cette mauvaise lecture s’est produite est restée un mystère, jusqu’à une récente étude menée par des chercheurs de l’Université Brown et de l’équipe multi-institution Northeastern Collaborative Access du Laboratoire national d’Argonne (géré par l’Université Cornell).
En créant un cristal – une structure ordonnée d’unités identiques – de la petite sous-unité ribosomique liée à l’ARNm en présence de streptomycine, les chercheurs ont généré plusieurs « instantanés » détaillés qui ont révélé des détails clés au niveau moléculaire de l’interaction, montrant finalement comment la streptomycine altère la fonction de la sous-unité. À la ligne de faisceau NSLS X25, ils ont utilisé une technique appelée cristallographie aux rayons X, dans laquelle un faisceau de rayons X est dirigé vers le cristal, interagit avec les molécules et produit un motif de diffraction complexe. À partir du modèle, à l’aide d’un logiciel informatique, le groupe a construit des représentations visuelles du complexe sous-unité – ARNm-streptomycine.
En bref, les chercheurs ont pu « voir » pour la première fois les manières subtiles dont la streptomycine déforme la structure du site de décodage de la sous-unité, ce qui l’amène à lire incorrectement l’ARNm. Par exemple, la liaison à la streptomycine réduit la distance entre deux des nombreuses hélices qui composent la structure moléculaire de la sous-unité. Ceci est particulièrement significatif car ces hélices forment le site de décodage proprement dit, et le décodage n’a lieu correctement que si ces éléments sont orientés exactement à droite par rapport à l’ARNm et à l’ARNt sélectionnés. La liaison à la streptomycine induit également un changement dans la relation entre l’une de ces deux hélices et une troisième hélice, entraînant une rétractation de l’une de l’autre ou un « désengagement ». »
Le résultat final de toutes ces légères altérations est que la streptomycine déstabilise la liaison entre la sous-unité et l’ARNt « correct » tout en stabilisant simultanément la liaison de la sous-unité à l’ARNt « incorrect », éliminant ainsi efficacement la discrimination entre l’ARNt correct et l’ARNt incorrect. Cela provoque des ravages dans la chaîne d’approvisionnement bactérienne pour les nouvelles protéines, perturbant la reproduction et le cycle de vie des bactéries.
« Nos études structurelles ont révélé que la streptomycine induit des distorsions étonnamment importantes dans le ribosome bactérien, ce qui nous aide à comprendre comment cet antibiotique interfère avec la synthèse des protéines chez les bactéries », a déclaré le chercheur principal Gerwald Jogl, professeur agrégé de biologie au département de biologie moléculaire de Brown, Biologie cellulaire & Biochimie. « Dans la continuité de nos découvertes actuelles, nous étudions maintenant comment des mutations dans les ribosomes bactériens peuvent contrecarrer ces réarrangements structurels et permettre aux bactéries de survivre à l’action autrement mortelle de la streptomycine. »
Cette recherche a été publiée dans l’édition du 15 janvier 2013 de Nature Communications, sous le titre « A structural basis for streptomycin-induced misreading of the genetic code. »Le soutien est venu des Instituts nationaux de la Santé et du ministère de l’Énergie.
2013-4251/INT|EXT/Salle de presse