Frontiers in Microbiology
introduktion
Astroviridae-familien indeholder ikke-indhyllede, positive sense, enkeltstrengede RNA-vira inden for to slægter, Mamastrovirus og Avastrovirus, som inficerer henholdsvis pattedyr og fugle. I øjeblikket anerkender Det Internationale Udvalg for taksonomi for vira (Det Internationale Udvalg for taksonomi for vira , 2018) 19 arter, nemlig Mamastrovirus-1 til -19, inden for Mamastrovirus-slægten; der er dog adskillige stammer, der afventer klassificering, hvoraf nogle foreløbigt betragtes som nye arter (Donato og Vijaykrishna, 2017).
siden 2010 er flere astrovira i stigende grad blevet anerkendt som neuroinvasive hos forskellige pattedyrarter, herunder mennesker., 2010; Naccache et al., 2015), mink (Blomstr Larsm et al., 2010), kvæg (Li et al., 2013), får (Pfaff et al., 2017), og grise (Boros et al., 2017). Efter indledende anerkendelse af bovin astrovirus-associeret encephalitis hos amerikanske kvæg (Li et al., 2013), En retrospektiv undersøgelse i tilfælde af sporadisk bovin encephalitis af ubestemt etiologi fra Svejts afslørede, at dette neuroinvasive astrovirus var gået uopdaget i årtier (Selimovic-Hamsa et al., 2016). Selvom epidemiologi og transmissionsveje for disse astrovira er ukendte, er transmission på tværs af arter blevet foreslået baseret på det høje identitetsniveau (>98%), delt mellem kvæg og får neuroinvasive astrovira ved nukleotid-og aminosyreniveauerne (Boujon et al., 2017).
Bovin astrovira (Pralev), navngivet BoAstV-NeuroS1 (Li et al., 2013) og Pralv-CH13 (Bousalas et al., 2014), blev oprindeligt fundet i hjernen hos kvæg med ikke-suppurativ encephalitis i henholdsvis USA og Sverige. På trods af den forskellige nomenklatur repræsenterer begge vira den samme genotypeart (Bousalas et al., 2016; Selimovic et al., 2017a), der stadig afventer Officiel klassificering af ICTV. I 2015 blev en tidligere ukendt boastv-stamme, der hedder BoAstV-CH15, identificeret i hjernen hos køer med encephalitis i Sverige. Fuld genom fylogenetisk sammenligning afslørede et tættere forhold mellem Pralv-CH15 og et får astrovirus (OvAstV) end med Pralv-CH13 (Seuberlich et al., 2016). Coinfektion med BoAstV-CH13 og BoAstV-CH15 blev også dokumenteret i et tilfælde (Seuberlich et al., 2016). Samme år i Tyskland, Schlottau et al. (2016) rapporterede en ny astrovirus, nemlig Pralv-BH89/14, hos en ko med encephalitis, der var mest beslægtet med OvAstV og Pralv-CH15. Derefter blev BoAstV-CH13/NeuroS1 identificeret i 2017 i tilfælde af bovin encephalitis i det østlige og vestlige Canada (Spinato et al., 2017; Selimovic et al., 2017b). I 2018 blev en ny neuroinvasiv Pralv tæt beslægtet med nordamerikansk og europæisk Pralv-NeuroS1/Pralv-CH13 identificeret i en styre med ikke-suppurativ encephalomyelitis i Japan, og forekomsten af intra-genotypisk rekombination mellem de nordamerikanske og europæiske stammer blev foreslået (Hirashima et al., 2018).
mens der er rapporteret tilfælde af astrovirus-associeret encephalitis i Nordamerika, Europa og Asien, er deres tilstedeværelse aldrig blevet dokumenteret på den sydlige halvkugle. Her beskriver vi et tilfælde af astrovirus-associeret encephalitis hos kvæg i Uruguay, som udvider den geografiske fordeling og genetiske mangfoldighed af neuroinvasive astrovirus og giver fylogeografiske beviser, der antyder, at denne virus blev introduceret i Amerika fra Europa og senere spredt til Asien.
materialer og metoder
historie og signalering
i juni 2018 udviklede en 22 måneder gammel Holstein-styring i en gruppe på 37 stude i en 300 hektar stor gård i Colonia, Uruguay, progressive neurologiske synger inklusive usædvanlig opførsel, målløs Vandring, cirkling, ataksi, gentagne og ukoordinerede tungebevægelser og recumbency. Besætningen græssede på en årlig havregræs og blev suppleret med majsensilage. En formodet klinisk diagnose af cerebral listeriose af veterinærlægen førte til behandling med penicillin og streptomycin, men dyret døde spontant efter et klinisk forløb, der varede 3 dage.
patologisk undersøgelse, in situ hybridisering (ISH) og immunhistokemi (IHC)
styrehovedet blev fjernet fra slagtekroppen og sendt til INIAS Veterinary Diagnostic Laboratory (Animal Health Platform) til diagnostisk oparbejdning. Halvdelen af hjernen, et kort segment af proksimal cervikal rygmarv (C1), trigeminal ganglion og roden af trigeminusnerven, spytkirtel, retropharyngeal lymfeknude, oropharyngeal, spiserør, tunge og skeletmuskulatur, blev nedsænkning-fikseret i 10% neutralt bufret formalin i 48-72 timer.
kromogen ISH blev udført manuelt på 5 dele af formalinfast, paraffinindlejret (FFPE) hjernestamme, cerebrum og cerebellum på SuperFrost plus-dias (Thermo Fisher Scientific, Pittsburgh, PA, USA) ved hjælp af rnascope 2.5 Red assay kit (Cat #322360, Advanced Cell Diagnostics, CA, USA) og Pralv probe Cat. #406921. Sonden er sammensat af 20 S par målretning Region 5232-6180 af virus (GenBank KF233994.1). Hver 5 kg sektion af væv blev forbehandlet med varme og protease inden probehybridisering i 2 timer ved 40 kg C og behandlet i henhold til producentens anbefalinger. Negative kontroller, der blev brugt til validering af signal, omfattede en ikke-relateret (GC-indhold matchet) sonde, der kørte på serielle sektioner og sonderende væv fra uinficerede dyr. Slides blev modfarvet med hæmatoksylin og monteret med EcoMount (Biocare Medical, Concord, CA, USA).
derudover blev IHC udført i FFPE-sektioner af hjernestamme, cerebrum og cerebellum, som tidligere beskrevet, til identifikation af Vestnilvirus (VNV, Flavivirus) (Palmieri et al., 2011), rabiesvirus (Lyssavirus) (Stein et al., 2010), og Chlamydia spp. (Giannitti et al., 2016) antigener.
molekylær virologi
nukleinsyreekstraktion blev udført fra en poolet prøve af frosset (-20 liter C) hjerne ved hjælp af Magmaks Nukleinsyreisolationskit Karri (Thermo Fisher Scientific). Til astrovirusdetektion blev revers transkription (RT) udført med RevertAid revers transkriptase (Thermo Fisher Scientific) og tilfældige sekskamerprimere (Chiagen). PCR blev udført fra cDNA ved anvendelse af Mangomiks-kar (Biolin) og primere, der amplificerer et 432-nukleotidfragment af astroviruspolymerasegenet (Tse et al., 2011). PCR-produktet blev visualiseret i 2% agarosegel, oprenset ved hjælp af PureLink-hurtig Gelekstraktion og PCR-Oprensningskombinationssæt (Invitrogen) og sekventeret ved Macrogen Inc. (Seoul, Sydkorea). For astrovirus hele genom amplifikation, Maksima h Minus revers transkriptase(Thermo Fisher Scientific) og oligo (dT)18 til obtention af cDNA og Mangomiks-kar (Biolin) eller Ranger-DNA-Polymerase (Biolin) med primere beskrevet af Hirashima et al., 2018, blev brugt. PCR-produkterne blev visualiseret i 1-2% agarosegel, oprenset og sekventeret som nævnt ovenfor. Sekvenssamling blev udført med Seksman (Lasergene 8, DNASTAR). Seksogtyve komplette genomsekvenser af neuroinvasiv astrovirus fra kvæg, får, svin, mennesker og mink og enterisk bovint astrovirus tilgængelig i GenBank blev hentet og justeret ved hjælp af Clustal V i MEGA 7-programmer (Kumar et al., 2016). 1 (Trifinopoulos et al., 2016) blev brugt til at bestemme den bedst egnede model for sekvensudvikling (SYM+i+G4) og til at konstruere et fylogenetisk træ med maksimal sandsynlighed med de næsten komplette sekvenser af BoAstV detekteret i dette tilfælde og de komplette sekvenser hentet fra GenBank ved hjælp af bootstrap som den statistiske metode til vurdering af clades robusthed. Lighed plot blev udført med SimPlot program (Lole et al., 1999). P-afstande på aminosyreniveau af ORF2 blev estimeret med MEGA 7-programmer (Kumar et al., 2016).
derudover blev der udført en bayesisk fylogeografisk analyse med BEAST v1.8.4-pakken (Drummond et al., 2012), ved hjælp af: den komplette kodningsregion for Pralv CH13 / NeuroS1-afstamning, ORF1ab (ikke-strukturelle gener), ORF2 (strukturelt gen), ORF1a (protease) og en delvis ORF1b (polymerase genomisk region, for hvilken Canadiske stammer var tilgængelige) med alle sekvenser tilgængelige i GenBank (sidste tiltrædelse 18.April 2019) for at bestemme den evolutionære hastighed, de fælles forfædres aldre/år og den mest sandsynlige rute for viral cirkulation efter land (Sverige, Uruguay, USA, Canada og Japan). Manglende rekombination i datasættet blev bestemt ved anvendelse af Rekombinationsdetekteringsprogram 4. Den substitutionsmodel, der bedst passer til hver justering, blev bestemt ved hjælp af MEGA 7-programmer gennem Bayesian information criterion (BIC) værdier, og den tidsmæssige struktur for hvert datasæt blev evalueret ved hjælp af TempEst (Rambaut et al., 2016). Det lognormale afslappede molekylære ur med Bayesian Skyline-analyse blev valgt af Bayes-faktor blandt de forskellige kombinationer af molekylære Ure og anvendte koalescerende træpriorer. Detektionslandet blev brugt som træk. Markov-kæden Monte Carlo-længden var 100 millioner generationer, hvilket sikrede konvergensen af analysen, evalueret i Tracer v1.6.0, og den bageste sandsynlighed blev brugt til at evaluere klader. Den maksimale clade troværdighed træ (MCCT) blev opnået ved hjælp af TreeAnnotator programmel fra BEAST og visualiseret i FigTree v1.4.3.
endelig blev DNA ekstraheret fra frossen hjerne behandlet ved PCR til påvisning af bovine herpesvirus 1 og 5 (BHV-1 og -5), som tidligere beskrevet (Ashbaugh et al., 1997).
bakteriologi
friske prøver af cerebrum og hjernestamme blev rutinemæssigt behandlet til aerobe bakteriekulturer i blod og MacConkey-agarer og selektiv kultur for Listeria monocytogenes (Al-Sandine og Sandine, 1990).
resultater og diskussion
de kliniske tegn og epidemiologiske fund i det her beskrevne tilfælde, omend ikke-specifikke, svarede til dem, der er beskrevet i andre tilfælde af bovin astrovirus-associeret encephalitis, som normalt beskrives som sporadisk (Selimovic-Hamsa et al., 2016), med en række neurologiske underskud (Deiss et al., 2017), med en varighed af kliniske tegn, der typisk spænder fra 1 dag til 3 uger (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
makroskopisk undersøgelse af hjernen, C1-segmentet i rygmarven og andre væv i hovedet afslørede ikke signifikante brutto anatomiske læsioner. Histologisk var der moderat til svær, lymfocytisk, histiocytisk og plasmacytisk meningoencephalomyelitis, der påvirkede telencephalon (inklusive cerebral halvkugle og hippocampus), hjernestamme og det eneste undersøgte segment af rygmarven. Læsioner blev overvejende fordelt i det grå stof og begrænsende områder af hvidt stof. I de berørte områder var der perivaskulær manchettering og lymfoplasmacytisk og histiocytisk inflammation og neuronal nekrose/neuronofagi med gliose i den tilstødende neuropil. Der var satellitose af berørte, nekrotiske neuroner (figur 1a–d). Læsionerne var meget mindre hyppige og alvorlige i cerebellar parenchyma, skønt der var multifokal moderat cerebellar leptomeningitis. Der blev ikke fundet intralesionale bakterier med H&E og Gram pletter. Der blev ikke fundet signifikante histologiske ændringer i de andre undersøgte væv.

Figur 1. Histologiske læsioner i hjernestammen (A,B) og hjernebarken (C,D) og påvisning af Pralv RNA i hjernebarken (E,G). Billeder (A-D) er dele af hjernen farvet med H&E; billeder E og G er sektioner af hjernebarken, der demonstrerer hybridisering ved hjælp af kromogen ISH ved hjælp af den BoAstV-specifikke sonde, modfarvet med hæmatoksylin; billede F er en seriel sektion af hjernebarken DapB probe (negativ kontrol), modfarvet med hæmatoksylin. (A) et perivenulært rum udvides markant af inflammatoriske celler (for det meste lymfocytter og histiocytter), der også infiltrerer den tilstødende neuropil. Den neuronale krop er omgivet af et øget antal gliale (satellitose) og inflammatoriske celler. C) leptomeninge infiltreres af lymfocytter og histiocytter. (D) en region i hjernebarken med flere hypereosinofile (nekrotiske) neuroner og et stort kar med perivaskulære lymfocytiske manchetter. (E) i en seriel sektion af hjernebarken er den rigelige intracytoplasmatiske Pralv-RNA-mærkning afbildet ved stærk, granulær rød kromogenaflejring inden for neuronal cytoplasma af soma og neuronale udvidelser (E, G), der ikke er til stede ved hybridisering ved hjælp af negativ kontrolprobe (F).
en neuroinvasiv virusinfektion blev mistænkt ved histologisk undersøgelse af centralnervesystemet. Kvæg med encephalitis er bekymrende, fordi mange drøvtyggere neuropatogener er dyrepatogener (Cantile and Youssef, 2016); således bør en diagnose af encephalitis bede om omfattende laboratorietest for at screene for infektiøse agenser, når det er muligt. I det tilfælde, der er beskrevet heri, IHC ‘ er for VNV, rabiesvirus og Chlamydia spp. PCR for BHV – 1 og -5 var alle negative, og ingen patogene bakterier blev dyrket fra hjernevæv. Da styret var < 2 år gammelt, og der ikke blev observeret nogen spongiforme ændringer i hjernestammen, blev dyret ikke testet for bovin spongiform encephalopati (BSE), som er en eksotisk sygdom hos voksne kvæg, der aldrig er rapporteret i Uruguay. Desuden er BSE ikke inflammatorisk (Cantile and Youssef, 2016).
in situ hybridisering blev udført under anvendelse af en sonde genereret fra Pralv-NeuroS1, og der var probehybridisering rigelig inden for og begrænset til cytoplasma af neuroner i den cerebrale halvkugle og hippocampus (figur 1e–G). I disse områder sondehybridiseringen kolokaliseret med nekrotiske neuroner og regioner af gliose uden sondehybridisering detekterbar i gliacellerne eller inflammatoriske celler i de perivaskulære manchetter. Ingen viral nukleinsyre blev påvist af ISH i lillehjernen, som kun havde minimale inflammatoriske læsioner i parenchymen, men moderat leptomeningitis eller hjernestammen, inklusive sektioner med svær betændelse. Dette betyder, at topografisk påvisning af viral distribution med ISH var mere begrænset end encephalitis i de undersøgte sektioner, som lejlighedsvis er blevet beskrevet i tilfælde af Pralv-CH13/NeuroS1-associeret encephalitis hos kvæg (Selimovic-Hamsa et al., 2017a, b). En årsag til denne lejlighedsvise mangel på viral RNA-detektion i læsionerede områder af hjernen kan være detektionsgrænsen for ISH eller clearance af virussen i betændte områder af hjernen på dødstidspunktet, som tidligere foreslået., 2017b). Som forventet blev der ikke påvist nogen probehybridisering af ISH i hjernevævet anvendt som negativ kontrol.
Astrovirus blev påvist i hjernen ved RT-PCR. Næsten komplet genomsekvensanalyse afslørede en Mamastrovirusstamme inden for CH13/NeuroS1-kladen, vi kaldte BoAstV-Neuro-Uy, sekvensen blev deponeret i GenBank under tiltrædelsesnummer MK386569. Den fylogenetiske analyse afslørede nærhed med andre neuroinvasive astrovira inden for Virginia/Human-mink-får (VA/HMO) clade (figur 2), som indeholder mest kendte neuroinvasive astrovira (Hirashima et al., 2018; Reuter et al., 2018). Den næsten komplette sekvens af Pralv-Neuro-Uy er 6427 bp i længden og har en sekvensidentitet på 94% med KagoshimaSR28-462 stamme. Pralv-Neuro-Uy har lignende træk som andre stammer af afstamning CH13/NeuroS1: en 5 ‘ UTR-region på 51 nt, ORF1a (protease) på 861 aminosyrer (aa), ORF1b på 523 aa (RNA-afhængig RNA-polymerase) og ORF2 på 758 aa (kapsidprotein). Desværre kunne 3 ‘ UTR ikke sekventeres, men en poly(A) hale antages at være til stede, fordi oligo(dT)18 blev brugt til at opnå cDNA. Derudover er den heptameriske aaaaaac-sekvens, et ribosomalt frameshift-signal, til stede. P-afstande på aminosyreniveauet af ORF2 bekræftede tildelingen af denne stamme til CH13/NeuroS1-kladen. P-afstande < 0.35 mellem BoAstV-Neuro-Uy og andre medlemmer af denne clade (tabel 1) ville understøtte en klassificering af disse virale stammer inden for en samme art; Mamastrovirus-13 er for nylig blevet foreslået af andre forfattere (Donato og Vijaykrishna, 2017; Hirashima et al., 2018), selvom bestemt artstildeling af ICTV afventer. Sonden anvendt til ISH, genereret fra Pralv-NeuroS1, havde 92,7% sekvensidentitet med Pralv-Neuro-uy.
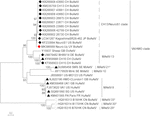
figur 2. Fylogenetisk analyse af nukleotidsekvenser i fuld længde ved maksimal sandsynlighedsmetode. BoAstV-Neuro-Uy er markeret med en rød rhombus. Andre sekvenser af bovine neuroinvasive astrovira er markeret med sort rhombi, sekvenser fra ikke-bovine neuroinvasive astrovira er markeret med sorte trekanter, og sekvenser fra enteriske bovine astrovira er markeret med hvide cirkler. Mamastrovirus arter er vist; stjerner angiver arter, der endnu ikke er anerkendt af ICTV. Stammer inden for CH13 / NeuroS1-kladen, der endnu ikke er tildelt en art af ICTV, identificeres med en stiplet linje. Bootstrap-værdier er angivet i noder. CH, Sverige; JP, Japan; USA, Amerikas Forenede Stater; UY, Uruguay; GB, Det Forenede Kongerige Storbritannien; De, Tyskland; Se, Sverige; FR, Frankrig; CN, Kina. Praleri, bovint astrovirus; OvAstV, får astrovirus; PoAstV, svin astrovirus; HuAstV, humant astrovirus; MiAstV, mink astrovirus.
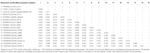
tabel 1. Estimater af evolutionær divergens ved aminosyreniveauet i den komplette ORF2-region mellem sekvenser af kvæg og får Mamastrovirus-13 og stammer inden for CH13/NeuroS1-kladen, der endnu ikke er tildelt en art af International Committee on Taksonomy of virus (ICTV).
undersøgelser baseret på neuropatologiske undersøgelser og astrovirus-nukleinsyre-og proteindetektion har konkluderet, at der er en sandsynlig årsagssammenhæng mellem astrovirusinfektion og neurologisk sygdom og læsioner hos kvæg., 2017a; Reuter et al., 2018). Efter vores bedste viden er astrovirus-associeret encephalitis endnu ikke gengivet eksperimentelt. Dette ville kræve isolering af neuroinvasive astrovira fra kliniske tilfælde, hvilket ikke blev forsøgt i vores tilfælde.
kilden til Pralev-Neuro-Uy i dette tilfælde kunne ikke bestemmes. Reservoirkvæg og dyreliv bør dog overvejes, da kvæget blev opdrættet under omfattende udendørs forhold. Det berørte dyr var købt og flyttet til gården i februar 2018 sammen med andre 9 stude. Desværre nægtede ejeren yderligere prøveudtagning og testning af andre dyr i ejendommen og en mere detaljeret epidemiologisk undersøgelse. Ingen af de andre dyr i gruppen havde udviklet neurologisk sygdom fra August 2018, sidste gang dyrlægen blev kontaktet. En sæsonbestemt periode fra begyndelsen af vinteren til slutningen af foråret er blevet foreslået for tilfælde af astrovirusassocieret encephalitis i Sverige (Selimovic-Hamsa et al., 2016). Interessant nok forekom den her beskrevne sag i Juni svarende til overgangsperioden efterår-vinter på den sydlige halvkugle.
mens neurotrope astrovira er blevet identificeret i Nordamerika (Li et al., 2013; Spinato et al., 2017), Europa (Bousalas et al., 2014) og Asien (Hirashima et al., 2018), er deres tilstedeværelse aldrig blevet rapporteret på den sydlige halvkugle, så denne meddelelse udvider den geografiske fordeling af astrovirus-associeret encephalitis. For at vurdere, om den virale stamme, der blev påvist i Uruguay, kunne have sin oprindelse i Europa, Nordamerika, eller Asien, vi estimerede den evolutionære hastighed og udførte en fylogeografisk analyse ved hjælp af neuroinvasive BoAstV-sekvenser tilgængelige i GenBank. Den estimerede evolutionshastighed ved hjælp af den komplette kodningsregion var 4.27 10-4 (95% højeste sandsynlighedstæthed-HPD -, 2,19-6,46 10-4) nukleotidsubstitutioner/sted/år, hvilket forventes for en RNA-virus (Jenkins et al., 2002), men lavere end det, der anslås for enteriske humane astrovira (Babkin et al., 2012, 2014). Den ORF1ab region viste en lignende evolutionære sats (4.20 × 10-4, 95% HPD 1.66–6.46 × 10-4 udskiftninger/site/år) som komplet kodning af regionen, mens den ORF1a (2.92 × 10-4, 95% HPD 1.19 × 10-6-6.46 × 10-4 udskiftninger/site/år) og ORF2 (2.86 × 10-4, 95% HPD 4.13 × 10-6-5.79 10-4 substitutioner/sted / år) viste en lidt hurtigere evolutionær hastighed, og den partielle polymerasegenomiske region (ORF1b) viste en lidt langsommere evolutionær hastighed (5,39 10-4, 95% HPD 6,41 10-7-1, 10 10-3 substitutioner/sted/år).
som bestemt af den fylogeografiske analyse med den komplette kodningsregion og vist i MCTT (figur 3) er der to underlinier (CH13 og NeuroS1) baseret på referencestammer, der har en fælles forfader. Den seneste fælles forfader til disse underlinjer (afstamning CH13/NeuroS1) opstod i Europa cirka i 1885 (95% HPD, 1794-1940). I begyndelsen af 1900 ‘ erne divergerede de to underlinjer, CH13-underlinjen forblev cirkulerende i Europa, mens NeuroS1-underlinjen spredte sig til Amerika og Asien. Det mest sandsynlige scenario er, at NeuroS1-underlinjen blev introduceret i Uruguay fra Europa omkring år 1921 (95% HPD, 1849-1967), formodentlig gennem husdyrhandel, derefter spredt til Nordamerika og senere til Japan (figur 3). På grund af begrænsningen i antallet af sekvenser, der er tilgængelige i GenBank, som kunne have forudindtaget analysen, blev de opnåede resultater ved anvendelse af det komplette kodningsregion sammenlignet med dem, der blev opnået med andre genomiske regioner (ORF1ab, ORF2, ORF1a og ORF1b), der var tilgængelige for et større antal stammer (dvs.Canadiske stammer). I alle analyserne er det mest sandsynlige scenario, at introduktionen af virussen til Uruguay fandt sted fra Europa (supplerende tal S1A–d). Derudover svarede den estimerede dato for denne introduktion, opnået med ORF1ab og partiel polymerase genomisk region (ORF1b) (supplerende tal S1A,D), til den, der blev opnået med den komplette kodningsregion,mens den estimerede introduktionsdato opnået med ORF2 og ORF1a var tidligere, men med bredere 95% HPD-interval (supplerende tal S1B, C). En introduktion af sub-lineage NeuroS1 direkte til Canada fra Europa, med efterfølgende spredning til USA og Japan, er også plausibel, s vist i supplerende figur S1D.

figur 3. Maksimal clade troværdighed træ opnået ved analyse af kodningsregionen i fuld længde. Farven på grenene repræsenterer det mest sandsynlige land, hvor forfædrene cirkulerede, posterior sandsynlighedsværdier vises i grenene, og tallene i hver knude repræsenterer oprindelsesårene for hver klade med 95% HPD-intervallet. Underlinier er angivet med etiketter.
yderligere undersøgelser er nødvendige for at vurdere den geografiske fordeling, patogene mekanismer (især mekanismer til transmission og indrejse), molekylær epidemiologi og potentiel interspecies transmission af neuroinvasive astrovira.
datatilgængelighed
datasættene genereret til denne undersøgelse findes i GenBank, MK386569.
Forfatterbidrag
FG, RDC og MC bidrog med opfattelsen af undersøgelsen. FG og RDC udførte den patologiske undersøgelse og prøveudtagning. PP udførte in situ hybridisering. FU udførte immunhistokemi. LM, RC og MC udførte molekylær virologi test. MC udførte sekvens-og fylogeografiske analyser og tilhørende figurer. FG og PP opnåede de histologiske billeder. MF udførte bakteriekulturerne. FG og MC skrev det første udkast til manuskriptet. RDC, PP, FU, LM, MF og RC skrev dele af manuskriptet. Alle forfattere bidrog til manuskriptrevision, læste og godkendte den indsendte version.
Funding
dette arbejde blev finansieret af tilskud PL-015 N-15156 fra INIA og 158 fra “Programa de Iniciaci Pristn a la Investigaci Pristn 2017 “fra” Comisi Pristern Sektorial de Investigaci prisT cient Pritfica ” (CSIC). MC og RDC anerkender støtte fra henholdsvis” National Agency for Research and Innovation ” (ANII) og INIA gennem ph.d. – stipendier. FG anerkender støtte fra ANII gennem mobilitetstilskud MOV_CA_2018_1_150021.
interessekonflikt Erklæring
forfatterne erklærer, at forskningen blev udført i mangel af kommercielle eller økonomiske forhold, der kunne fortolkes som en potentiel interessekonflikt.
anmelderen TS erklærede et tidligere medforfatterskab med en af forfatterne PP til håndteringsredaktøren.
anerkendelser
forfatterne takker Yisell Perdomo og Cecilia Monesiglio fra INIA, og Karen Sverlav og Juliann Beingesser fra cahfs for teknisk assistance.
supplerende materiale
det supplerende materiale til denne artikel kan findes online på: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
figur S1 / maksimal clade troværdighed træer (MCCTs) opnået ved analyse af fuld længde ORF1ab (a), Fuld længde ORF2 (B), Fuld længde ORF1a (C) og delvis ORF1b (D). Farven på grenene repræsenterer det mest sandsynlige land, hvor forfædrene cirkulerede, posterior sandsynlighedsværdier vises i grenene, og tallene i hver knude repræsenterer oprindelsesårene for hver klade med 95% HPD-intervallet. Underlinier er angivet med etiketter.
fodnoter
- ^http://iqtree.cibiv.univie.ac.at
Sandine (1990). Meget selektivt medium til isolering af Listeria monocytogenes fra fødevarer. Appl. Environ. Mikrobiol. 56, 3154–3157.
PubMed abstrakt | Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chovdhury, S. og Collins, J. K. (1997). Specifik påvisning af udskillelse og latens af bovint herpesvirus 1 og 5 ved anvendelse af en indlejret polymerasekædereaktion. J. Vet. Diagn. Investere. 9, 387–394. doi: 10.1177/104063879700900408
PubMed abstrakt / CrossRef fuldtekst / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Girakovskaia, E. V. og Tikunova, N. V. (2014). Rekombinationsanalyse baseret på HAstV-2 og HastV-4 komplette genomer. Inficere. Genet. Evol. 22, 94–102. doi: 10.1016 / j. meegid.2014.01.010
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Babkin, I. V., Tikunov, A. Y., Girakovskaia, E. V., Netesov, S. V. og Tikunova, N. V. (2012). Høj evolutionær hastighed af humant astrovirus. Inficere. Genet. Evol. 12, 435–442. doi: 10.1016 / j. meegid.2012.01.019
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Blomstrl, A. L., bred L., F., Hammer, A. S., Bel L., S. og Berg, M. (2010). Påvisning af et nyt astrovirus i hjernevæv af mink, der lider af rystende minksyndrom ved brug af viral metagenomics. J. Clin. Mikrobiol. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Boros, Kurt, Albert, M., Pankovics, P., B Kurt, H., Pesavento, P. A., Phan, T. G., et al. (2017). Udbrud af neuroinvasiv astrovirus forbundet med encephalomyelitis, svaghed og lammelse blandt fravænnede grise, Ungarn. Emerg. Inficere. Dis. 23, 1982–1993. doi: 10.3201 / eid2312. 170804
PubMed abstrakt | CrossRef Fuld tekst | Google Scholar
Bojon, C. L., Koch, M. C. (2017). Indikation af overførsel på tværs af arter af astrovirus forbundet med encephalitis hos får og kvæg. Emerg. Inficere. Dis. 23, 1604–1608. doi: 10.3201 / eid2309.170168
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
I. G., J. L., J. L., J. L., S., Dr. L. L. L., C., Bruggmann, R. og Seuberlich, T. (2016). Fuldgenombaseret molekylær karakterisering af encephalitis-associerede bovine astrovira. Inficere. Genet. Evol. 44, 162-168. doi: 10.1016 / J. meegid.06/052/2016
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
I. G., I. G., I. G., I. G., I. G., I. G., I. M., M., et al. (2014). Neurotrop astrovirus hos kvæg med nonsuppurativ encephalitis i Europa. J. Clin. Mikrobiol. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Cantile, C. og Youssef, S. (2016). “Nervesystemet”, i Jubb, Kennedy og Palmer ‘ s Pathology of Domestic Animals, 6.Edn, Red. M. G. Maksie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamsa, S., Seuberlich, T. og Meylan, M. (2017). Neurologiske kliniske tegn hos kvæg med astrovirus-associeret encephalitis. J. Vet. Praktikant. Middelhavs. 31, 1209–1214. doi: 10.1111 / jvim.14728
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Donato, C. og Vijaykrishna, D. (2017). Det brede værtsområde og den genetiske mangfoldighed af astrovira fra pattedyr og fugle. Virus 9: E102. doi: 10.3390 / v9050102
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Drummond, A. J., Suchard, M. A., D. og Rambaut, A. (2012). Bayesian fylogenetik med BEAUti og Udyret 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093 / molbev / mss075
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
det er en af de mest almindelige måder at gøre det på. (2016). Chlamydia pecorum: føtale og placentale læsioner ved sporadisk abort af geder. J. Vet. Diagn. Investere. 28, 184–189. doi: 10.1177/1040638715625729
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Hele genomanalyse af et nyt neurotropisk bovint astrovirus påvist i en japansk sort styre med ikke-suppurativ encephalomyelitis i Japan. Bue. Virol. 163, 2805–2810. doi: 10.1007 / s00705-018-3898-3
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Det Internationale Udvalg for taksonomi af vira (2018). Master artsliste (MSL32), Version 1. Tilgængelig på: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (adgang til marts, 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G. og Holmes, E. C. (2002). Satser for molekylær evolution i RNA-vira: en kvantitativ fylogenetisk analyse. J. Mol. Evol. 54, 156–165. doi: 10.1007 / s00239-001-0064-3
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Kumar, S., Stecher, G. og Tamura, K. (2016). MEGA7: molekylær evolutionær genetik analyse version 7.0 for større datasæt. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093 / molbev / mv054
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
li, L., Diab, S., Mcgrav, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Divergerende astrovirus forbundet med neurologisk sygdom hos kvæg. Emerg. Inficere. Dis. 19, 1385–1392. doi: 10.3201 / eid1909. 130682
PubMed abstrakt | CrossRef Fuld tekst | Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Fuld længde human immundefektvirus type 1 genomer fra subtype C-inficerede serokonvertere i Indien, med tegn på intersubtype rekombination. J. Virol. 73, 152–160.
PubMed Abstrakt | Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et al. (2015). Diagnose af neuroinvasiv astrovirusinfektion hos en immunkompromitteret voksen med encephalitis ved upartisk næste generations sekventering. Clin. Inficere. Dis. 60, 919–923. doi: 10.1093 / cid / ciu912
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Palmieri, C., Franca, M., Usal, F., Anderson, M., Barr, B., skoven, L., et al. (2011). Patologi og immunhistokemiske fund af vestnilvirusinfektion hos psittaciformes. Dyrlæge. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., h Prisper, D., et al. (2017). En ny astrovirus forbundet med encephalitis og ganglioneuritis hos tamfår. På tværs. Emerg. Dis. 64, 677–682. doi: 10.1111 / tbed.12623
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Astrovirus encephalitis hos dreng med Røntgenforbundet agammaglobulinæmi. Emerg. Inficere. Dis. 16, 918–925. doi: 10.3201 / eid1606. 091536
PubMed abstrakt | CrossRef Fuld tekst / Google Scholar
Rambaut, A., Lam, T. T., maks Carvalho, L. og Pybus, O. G. (2016). Udforskning af den tidsmæssige struktur af heterochronous sekvenser ved hjælp af TempEst (formely Path-O-Gen). Virus Evol. 2: vev007. doi: 10.1093 / ve / ve007
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Reuter, G., Pankovics, P. og Boros, A. (2018). Nonsuppurativ (aseptisk) meningoencephalomyelitis forbundet med neurovirulente astrovirusinfektioner hos mennesker og dyr. Clin. Mikrobiol. Rev. 31, e00040-18. doi: 10.1128 / CMR.00040-18
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Schlottau, K., Schulse, C., Bilk, S., Hanke, D., H Primper, D., Beer, M., et al. (2016). Påvisning af et nyt bovint astrovirus hos en ko med encephalitis. På tværs. Emerg. Dis. 63, 253–259. doi: 10.1111 / tbed.12493
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
S., Boujon, C. L., Hilbe, M., Oevermann, A. og Seuberlich, T. (2017a). Hyppighed og patologisk fænotype af bovint astrovirus CH13/NeuroS1-infektion hos neurologisk sygdomskvæg: mod vurdering af årsagssammenhæng. Virus 9: E12. doi: 10.3390 / v9010012
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
S., Philibert, H., Clark, E. G. og Seuberlich, T. (2017b). Bovin astrovirus infektion i feedlot kvæg med neurologisk sygdom i det vestlige Canada. Kan. Dyrlæge. J. 58, 601-603.
Google Scholar
I. G., Vandevelde, M., Oevermann, A. og Seuberlich, T. (2016). Påvisning af astrovirus i historiske tilfælde af europæisk sporadisk bovin encephalitis, Sverige 1958-1976. Front. Dyrlæge. Sci. 3:91. doi: 10.3389 / fvets.2016.00091
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar
Seuberlich, T., D., Selimovic-Hamsa, S., Dr., Oevermann, A., Bruggmann, R., et al. (2016). Identifikation af et andet encephalitis-associeret astrovirus hos kvæg. Emerg. Mikrober Inficerer. 5: e5. doi: 10.1038 / emi.2016.5
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Spinato, M. T., Vince, A., Cai, H. og Ojkic, D. (2017). Identifikation af bovint astrovirus i tilfælde af bovin ikke-suppurativ encephalitis i det østlige Canada. Kan. Dyrlæge. J. 58, 607-609.
Google Scholar
Stein, L. T., Rech, R. R., Harrison, L. og brun, C. C. (2010). Immunhistokemisk undersøgelse af rabiesvirus inden for centralnervesystemet hos husdyr-og dyrearter. Dyrlæge. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A. Og Minh, B. K. (2016). Et hurtigt online fylogenetisk værktøj til maksimal sandsynlighedsanalyse. Nukleinsyrer Res. 44, B232–B235. doi: 10.1093 / nar / gk256
PubMed abstrakt / CrossRef Fuld tekst / Google Scholar
Tse, H., Chan, H. M., Tsoi, H. V., Fan, R. Y., Lau, C. C., Lau, S. K., Et Al. (2011). Genopdagelse og genomisk karakterisering af bovine astrovira. J. Gen. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0.030817-0
PubMed Abstrakt / CrossRef Fuld Tekst / Google Scholar