Biologia per le Major II
Risultati di Apprendimento
- conoscere il passato ed emergenti sistemi di classificazione dei virus
Perché la maggior parte dei virus si sviluppò probabilmente da diversi antenati, i metodi sistematici che gli scienziati hanno usato per classificare le cellule procariote ed eucariote non sono molto utili. Se i virus rappresentano “resti” di diversi organismi, anche l’analisi genomica o proteica non è utile. Perché?, Perché i virus non hanno sequenza genomica comune che loro tutti condividono. Ad esempio, la sequenza 16S rRNA così utile per la costruzione di filogenie procariote è di alcuna utilità per una creatura senza ribosomi! I biologi hanno utilizzato diversi sistemi di classificazione in passato. I virus sono stati inizialmente raggruppati per morfologia condivisa. Successivamente, i gruppi di virus sono stati classificati in base al tipo di acido nucleico che contenevano, DNA o RNA e se il loro acido nucleico era a singolo o doppio filamento. Tuttavia, questi metodi di classificazione precedenti raggruppavano i virus in modo diverso, perché erano basati su diversi set di caratteri del virus. Il metodo di classificazione più comunemente usato oggi è chiamato schema di classificazione di Baltimora e si basa su come l’RNA messaggero (mRNA) viene generato in ogni particolare tipo di virus.
Sistemi di classificazione passati
I virus contengono solo pochi elementi con cui possono essere classificati: il genoma virale, il tipo di capside e la struttura dell’involucro per i virus avvolti. Tutti questi elementi sono stati utilizzati in passato per la classificazione virale (Tabella 1 e Figura 1). I genomi virali possono variare nel tipo di materiale genetico (DNA o RNA) e nella sua organizzazione (singolo o doppio filamento, lineare o circolare e segmentato o non segmentato). In alcuni virus, ulteriori proteine necessarie per la replicazione sono associate direttamente al genoma o contenute all’interno del capside virale.
| Tabella 1. Classificazione dei Virus dalla Struttura del Genoma e Core | |
|---|---|
| Core Classificazioni | Esempi |
| RNA | virus della Rabbia, retrovirus |
| DNA | Herpesvirus, virus del vaiolo |
| Single-stranded | virus della Rabbia, retrovirus |
| Double-stranded | Herpesvirus, virus del vaiolo |
| Lineare | virus della Rabbia, retrovirus, herpesvirus, virus del vaiolo |
| Circolare | Papillomavirus, molti batteriofagi |
| Non segmentato: genoma è costituito da un unico segmento di materiale genetico | Parainfluenza virus |
| Segmentato: genoma è diviso in più segmenti | i virus dell’Influenza |
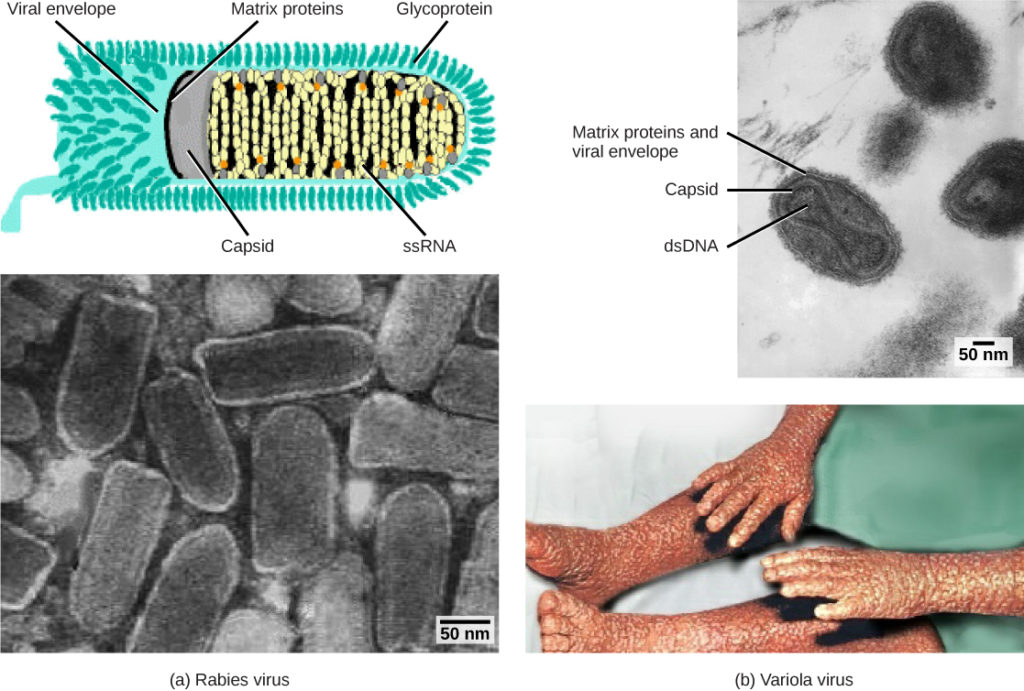
Figura 1. I virus sono classificati in base al loro materiale genetico principale e al design del capside. (a) Il virus della rabbia ha un nucleo di RNA a singolo filamento (ssRNA) e un capside elicoidale avvolto, mentre (b) il virus variola, l’agente causale del vaiolo, ha un nucleo di DNA a doppio filamento (dsDNA) e un capside complesso. (credito “diagramma rabbia”: modifica del lavoro da CDC; “rabbia micrograph”: modifica del lavoro da Dr. Fred Murphy, CDC; credito “piccolo vaiolo micrograph”: modifica del lavoro da Dr. Fred Murphy, Sylvia Whitfield, CDC; credito “vaiolo foto”: modifica del lavoro da CDC; scala-bar dati da Matt Russell)
I virus possono anche essere classificati in base alla progettazione dei loro capsidi (Tabella 2 e Figura 2). I capsidi sono classificati come icosaedrici nudi, icosaedrici avvolti, elicoidali avvolti, elicoidali nudi e complessi. Il tipo di materiale genetico (DNA o RNA) e la sua struttura (a singolo o doppio filamento, lineare o circolare e segmentato o non segmentato) sono utilizzati per classificare le strutture del nucleo del virus (Tabella 2).
| Tabella 2. Classificazione dei Virus con Capside Struttura | |
|---|---|
| Capside Classificazione | Esempi |
| Nudo icosaedrica | il virus dell’Epatite A, poliovirus |
| Avvolto icosaedrica | il virus di Epstein-Barr virus, l’herpes simplex virus, virus della rosolia, virus della febbre gialla, l’HIV-1 |
| Avvolto elicoidale | virus Influenzali, virus della parotite virus, virus del morbillo, virus della rabbia |
| Nudo elicoidale | virus del mosaico del Tabacco |
| Complesso con molte proteine; alcune combinazioni di icosaedrica e strutture del capside elicoidale | Herpesvirus, virus del vaiolo l’epatite B virus batteriofago T4 |

Figura 2. Micrografie di elettroni di trasmissione di vari virus mostrano le loro strutture. Il capside del (a) virus della poliomielite è nudo icosaedrico; (b) il capside del virus di Epstein-Barr è avvolto icosaedrico; (c) il capside del virus della parotite è un’elica avvolta; (d) il capside del virus del mosaico del tabacco è nudo elicoidale; e (e) il capside dell’herpesvirus è complesso. (credito a: modifica del lavoro di Dr. Fred Murphy, Sylvia Whitfield; credito b: modifica del lavoro di Liza Gross; credito c: modifica del lavoro di Dr. F. A. Murphy, CDC; credito d: modifica del lavoro di USDA ARS; credito e: la modifica, il lavoro di Linda Stannard, del Dipartimento di Microbiologia Medica, Università di Cape Town, in Sud Africa, la NASA; la barra della scala dati da Matt Russell)
Baltimore Classificazione
Il più comunemente utilizzato il sistema di classificazione dei virus è stato sviluppato dal Premio Nobel per biologo David Baltimore nei primi anni 1970. Oltre alle differenze nella morfologia e genetica di cui sopra, il Baltimore schema di classificazione dei gruppi di virus secondo il quale l’mRNA viene prodotta durante il ciclo replicativo del virus.
I virus del Gruppo I contengono DNA a doppio filamento (dsDNA) come genoma. Il loro mRNA è prodotto dalla trascrizione più o meno allo stesso modo del DNA cellulare.
I virus del gruppo II hanno DNA a filamento singolo (ssDNA) come genoma. Convertono i loro genomi a filamento singolo in un intermedio dsDNA prima che possa verificarsi la trascrizione in mRNA.
I virus del gruppo III usano il dsRNA come genoma. I fili si separano e uno di essi viene utilizzato come modello per la generazione di mRNA utilizzando la RNA polimerasi RNA-dipendente codificata dal virus.
I virus del Gruppo IV hanno ssRNA come genoma con polarità positiva. Polarità positiva significa che l’RNA genomico può servire direttamente come mRNA. Intermedi di dsRNA, chiamati intermedi replicativi, sono realizzati nel processo di copia dell’RNA genomico. Da questi intermedi si formano molteplici filamenti di RNA a tutta lunghezza di polarità negativa (complementari all’RNA genomico a filamento positivo), che possono quindi servire da modelli per la produzione di RNA a polarità positiva, inclusi sia RNA genomico a tutta lunghezza che MRNA virali più brevi.
I virus del gruppo V contengono genomi ssRNA con polarità negativa, il che significa che la loro sequenza è complementare all’mRNA. Come con i virus del gruppo IV, gli intermedi dsRNA sono usati per fare copie del genoma e produrre mRNA. In questo caso, il genoma negativo-incagliato può essere convertito direttamente in mRNA. Inoltre, i filamenti di RNA positivi a lunghezza intera sono fatti per servire da modelli per la produzione del genoma a filamento negativo.
I virus del gruppo VI hanno genomi SSRNA diploidi (due copie) che devono essere convertiti, utilizzando l’enzima trascrittasi inversa, in dsDNA; il dsDNA viene quindi trasportato al nucleo della cellula ospite e inserito nel genoma ospite. Quindi, l’mRNA può essere prodotto dalla trascrizione del DNA virale che è stato integrato nel genoma ospite.
I virus del gruppo VII hanno genomi dsDNA parziali e producono intermedi ssRNA che fungono da mRNA, ma vengono anche riconvertiti in genomi dsDNA mediante trascrittasi inversa, necessaria per la replicazione del genoma. Le caratteristiche di ciascun gruppo nella classificazione di Baltimora sono riassunte nella Tabella 3 con esempi di ciascun gruppo.
| Tabella 3. Classificazione di Baltimore | |||
|---|---|---|---|
| Gruppo | Caratteristiche | Modalità di Produzione di mRNA | Esempio |
| I | Double-stranded DNA | mRNA trascritto direttamente dal DNA template | Herpes simplex (herpesvirus) |
| II | Single-stranded DNA | DNA viene convertito in double-stranded forma prima di RNA trascritto | parvovirus Canino (parvovirus) |
| III | Double-stranded RNA | mRNA trascritto da il genoma RNA | Infanzia gastroenterite (rotavirus) |
| IV | Singolo filamento di RNA (+) | Genoma funzioni come mRNA | raffreddore Comune (pircornavirus) |
| V | Singolo filamento di RNA (−) | mRNA trascritto dalla RNA del genoma | la Rabbia (rhabdovirus) |
| VI | Singolo filamento di RNA virus con la trascrittasi inversa | trascrittasi Inversa rende di DNA dal genoma RNA; il DNA viene poi incorporato nel genoma ospite; mRNA è la trascrizione di una incorporated DNA | il virus dell’immunodeficienza Umana (HIV) |
| VII | Doppio filamento virus a DNA con reverse transcriptase | Il genoma virale è la doppia elica del DNA, ma il DNA virale viene replicato attraverso un intermedio di RNA; l’RNA può servire direttamente come mRNA o come un modello per fare mRNA | il virus dell’Epatite B (hepadnavirus) |