Biologia per Majors I
Risultati di apprendimento
- Comprendere la differenza tra pre-mRNA e mRNA
Dopo la trascrizione, i pre-MRNA eucariotici devono essere sottoposti a diverse fasi di lavorazione prima di poter essere tradotti. Anche i TRNA e gli RRNA eucariotici (e procariotici) vengono sottoposti a lavorazione prima che possano funzionare come componenti nel macchinario di sintesi proteica.
Elaborazione dell’mRNA
Il pre-mRNA eucariotico subisce un’ampia elaborazione prima di essere pronto per essere tradotto. I passaggi aggiuntivi coinvolti nella maturazione dell’mRNA eucariotico creano una molecola con un’emivita molto più lunga rispetto a un mRNA procariotico. Gli MRNA eucariotici durano diverse ore, mentre il tipico mRNA di E. coli dura non più di cinque secondi.
I pre-mRNA sono dapprima rivestiti in proteine stabilizzanti l’RNA; queste proteggono il pre-mRNA dalla degradazione mentre viene elaborato ed esportato fuori dal nucleo. I tre passaggi più importanti dell’elaborazione pre-mRNA sono l’aggiunta di fattori stabilizzanti e di segnalazione alle estremità 5 ‘e 3’ della molecola e la rimozione di sequenze intermedie che non specificano gli amminoacidi appropriati. In rari casi, la trascrizione dell’mRNA può essere “modificata” dopo essere stata trascritta.
5′ Capping
Mentre il pre-mRNA è ancora in fase di sintesi, un cappuccio di 7-metilguanosina viene aggiunto all’estremità 5 ‘ della trascrizione in crescita da un legame fosfato. Questa frazione (gruppo funzionale) protegge il nascente mRNA dalla degradazione. Inoltre, i fattori coinvolti nella sintesi proteica riconoscono il cappuccio per aiutare a iniziare la traduzione dai ribosomi.
3′ Poly-A Tail
Una volta completato l’allungamento, il pre-mRNA viene scisso da un’endonucleasi tra una sequenza di consenso AAUAAA e una sequenza ricca di GU, lasciando la sequenza AAUAAA sul pre-mRNA. Un enzima chiamato polimerasi poli-A aggiunge quindi una stringa di circa 200 residui A, chiamata coda poli-A. Questa modifica protegge ulteriormente il pre-mRNA dalla degradazione e segnala l’esportazione dei fattori cellulari di cui la trascrizione ha bisogno nel citoplasma.
Pre-mRNA Splicing
geni Eucariotici sono composti di esoni, che corrispondono a sequenze codificanti proteine (ex-on significa che essi non sono espressi), e intervenendo sequenze chiamate introni (introne indica loro di intervenire ruolo), che possono essere coinvolti nella regolazione genica, ma vengono rimossi dal pre-mRNA durante la lavorazione. Le sequenze di introni nell’mRNA non codificano le proteine funzionali.
La scoperta degli introni è stata una sorpresa per i ricercatori negli 1970 che si aspettavano che i pre-MRNA specificassero sequenze proteiche senza ulteriori elaborazioni, come avevano osservato nei procarioti. I geni degli eucarioti superiori contengono molto spesso uno o più introni. Queste regioni possono corrispondere a sequenze regolatorie; tuttavia, il significato biologico di avere molti introni o di avere introni molto lunghi in un gene non è chiaro. È possibile che gli introni rallentino l’espressione genica perché ci vuole più tempo per trascrivere i pre-MRNA con molti introni. In alternativa, gli introni possono essere residui di sequenza non funzionali lasciati dalla fusione di geni antichi durante l’evoluzione. Ciò è supportato dal fatto che esoni separati spesso codificano subunità o domini proteici separati. Per la maggior parte, le sequenze di introni possono essere mutate senza influenzare in ultima analisi il prodotto proteico.
Tutti gli introni di un pre-mRNA devono essere completamente e precisamente rimossi prima della sintesi proteica. Se il processo sbaglia anche da un singolo nucleotide, il telaio di lettura degli esoni ricongiunti si sposterebbe e la proteina risultante sarebbe disfunzionale. Il processo di rimozione degli introni e riconnessione degli esoni è chiamato splicing (Figura 1). Gli introni vengono rimossi e degradati mentre il pre-mRNA è ancora nel nucleo. Lo splicing avviene mediante un meccanismo specifico della sequenza che garantisce la rimozione degli introni e la ricongiunzione degli esoni con l’accuratezza e la precisione di un singolo nucleotide. Lo splicing dei pre-mRNA è condotto da complessi di proteine e molecole di RNA chiamati spliceosomi.
Domanda pratica
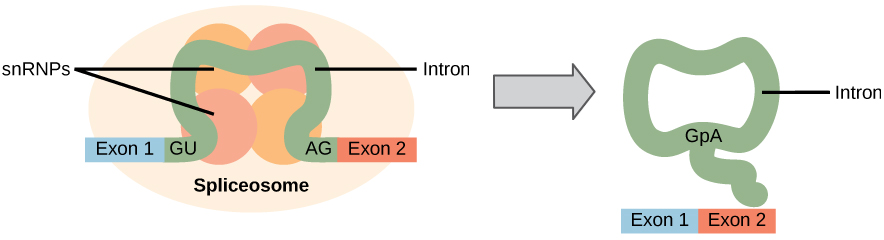
Figura 1. Lo splicing pre-mRNA comporta la rimozione precisa degli introni dalla trascrizione primaria dell’RNA. Il processo di splicing è catalizzato da complessi proteici chiamati spliceosomi che sono composti da proteine e molecole di RNA chiamate SNRNA. Gli spliceosomi riconoscono le sequenze all’estremità 5′ e 3 ‘ dell’introne.
Gli errori nello splicing sono implicati nei tumori e in altre malattie umane. Quali tipi di mutazioni potrebbero portare a errori di splicing?
Si noti che più di 70 introni individuali possono essere presenti, e ciascuno deve subire il processo di splicing—oltre a 5′ tappatura e l’aggiunta di una coda poli-A—solo per generare una singola molecola di mRNA traducibile.
Modifica dell’RNA nei tripanosomi
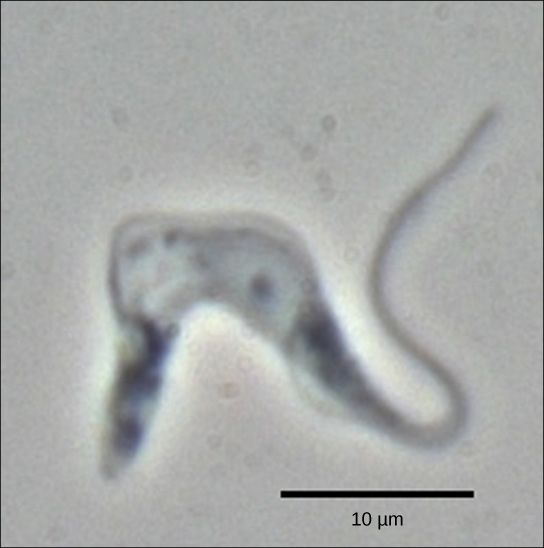
Figura 2. Trypanosoma brucei è l’agente eziologico della malattia del sonno negli esseri umani. Gli MRNA di questo agente patogeno devono essere modificati mediante l’aggiunta di nucleotidi prima che possa verificarsi la sintesi proteica. (credito: modifica del lavoro di Torsten Ochsenreiter)
I tripanosomi sono un gruppo di protozoi che includono il patogeno Trypanosoma brucei, che causa la malattia del sonno negli esseri umani (Figura 2). I tripanosomi, e praticamente tutti gli altri eucarioti, hanno organelli chiamati mitocondri che forniscono alla cellula energia chimica. I mitocondri sono organelli che esprimono il proprio DNA e si ritiene che siano i resti di una relazione simbiotica tra un eucariote e un procariote inghiottito. Il DNA mitocondriale dei tripanosomi presenta un’interessante eccezione al Dogma centrale: i loro pre-mRNA non hanno le informazioni corrette per specificare una proteina funzionale. Di solito, questo è perché l’mRNA manca diversi nucleotidi U. La cellula esegue un ulteriore passo di elaborazione dell’RNA chiamato RNA editing per rimediare a questo.
Altri geni nel genoma mitocondriale codificano 40-a 80-nucleotide RNA guida. Una o più di queste molecole interagisce per accoppiamento di base complementare con alcuni dei nucleotidi nella trascrizione pre-mRNA. Tuttavia, l’RNA guida ha più nucleotidi A che il pre-mRNA ha nucleotidi U con cui legarsi. In queste regioni, l’RNA guida si interrompe. Le 3 ‘ estremità degli RNA guida hanno una lunga coda poli-U e queste basi U sono inserite nelle regioni della trascrizione pre-mRNA in cui gli RNA guida sono in loop. Questo processo è interamente mediato da molecole di RNA. Cioè, gli RNA guida—piuttosto che le proteine-servono come catalizzatori nell’editing dell’RNA.
L’editing dell’RNA non è solo un fenomeno di tripanosomi. Nei mitocondri di alcune piante, quasi tutti i pre-MRNA sono modificati. L’editing di RNA è stato identificato anche in mammiferi come ratti, conigli e persino umani. Quale potrebbe essere la ragione evolutiva di questo ulteriore passaggio nell’elaborazione pre-mRNA? Una possibilità è che i mitocondri, essendo resti di antichi procarioti, abbiano un metodo altrettanto antico basato sull’RNA per regolare l’espressione genica. A sostegno di questa ipotesi, le modifiche apportate ai pre-MRNA differiscono a seconda delle condizioni cellulari. Anche se speculativo, il processo di editing di RNA può essere un retaggio da un tempo primordiale in cui le molecole di RNA, invece di proteine, erano responsabili per catalizzare le reazioni.
Provalo
Contribuisci!
Migliora questa paginaimpara di più