Creative Proteomics Blog
La sequenza di aminoacidi in una proteina o peptide può essere identificata dalla degradazione di Edman, che è stata sviluppata da Pehr Edman. Questo metodo può etichettare e fendere il peptide da N-terminale senza interrompere i legami peptidici tra altri residui di aminoacidi. La reazione di degradazione di Edman è stata automatizzata nel 1967 da Edman e Beggs. Al giorno d’oggi, la degradazione automatizzata di Edman (il sequenator della proteina) è usata ampiamente e può sequenziare i peptidi fino a 50 amminoacidi.
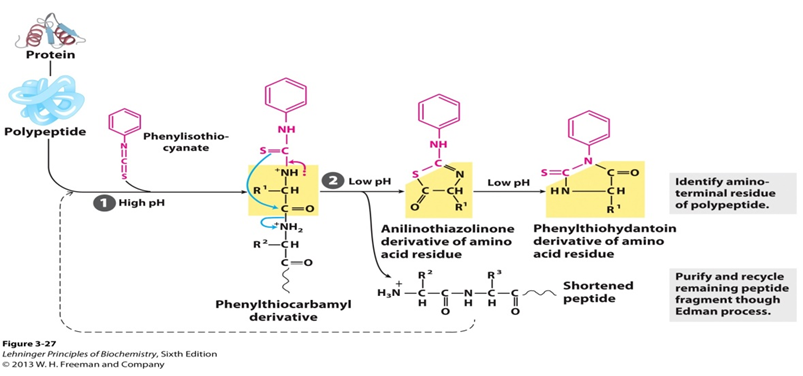
Degradazione ciclica dei peptidi basata sulla reazione del fenilisotiocianato con il gruppo amminico libero del residuo N-terminale in modo tale che gli amminoacidi vengano rimossi uno alla volta e identificati come derivati della feniltioidantoina. Parlando al processo specifico, un peptide non caricato viene fatto reagire con fenilisotiocianato (PITC) al terminale amminico in condizioni leggermente alcaline per dare un derivato feniltiocarbamoil (PTC-peptide). Quindi, in condizioni acide, lo zolfo tiocarbonilico del derivato attacca il carbonio carbonilico dell’amminoacido N-terminale. Il primo amminoacido viene scisso come derivato dell’anilinotiazolinone (ATZ-aminoacido) e il resto del peptide può essere isolato e sottoposto al successivo ciclo di degradazione. Una volta formato, questo derivato tiazolone è più stabile del derivato feniltiocarbamilico. L’aminoacido ATZ viene quindi rimosso mediante estrazione con acerato etilico e convertito in un derivato della feniltioidantoina (PTH-aminoacido). E la cromatografia può essere utilizzata per identificare il residuo di PTH generato da ciascun ciclo.
Per quanto riguarda la degradazione automatica di Edman, le proteine possono essere analizzate applicandole in soluzione su un filtro TFA e quindi caricate sullo strumento di sequenziamento di Edman. Le proteine nelle miscele vengono prima separate da gel 1D o 2D e quindi macchiate su una membrana PVDF. Le proteine vengono rilevate dalla colorazione Coomassie blue, Amido black o Poncau S e le proteine di interesse vengono tagliate e il pezzo di membrana PVDF caricato sul sequencer Edman.
Con la spettrometria di massa è stata sviluppata, l’uso del sequenziamento della degradazione di Edman ha iniziato a diminuire. Tuttavia, rimane ancora i metodi per diversi tipi di applicazioni di analisi strutturale delle proteine. Può essere utilizzato per verificare il limite N-terminale delle proteine ricombinanti o determinare l’N-terminale dei domini resistenti alla proteasi, in particolare quando la proteina o il dominio è >40-80 kDa o non può essere facilmente purificato. Può anche essere utilizzato per identificare il nuovo sito di scissione N-terminale e proteolitico nei frammenti proteici. Inoltre, per quanto riguarda alcune nuove proteine e peptidi in cui i database di sequenza non sono disponibili per la ricerca di database MS/MS, la degradazione di Edman può essere utilizzata per l’analisi.
L’amminoacido N-terminale della proteina può essere scisso. Pertanto, nel processo, il primo ciclo identifica quindi l’aminoacido N-terminale esatto. Inoltre, poiché gli amminoacidi rilasciati sono identificati e quantificati mediante cromatografia, è possibile identificare gli amminoacidi con peso molecolare identico. Ad esempio, isoleucina e leucina hanno una massa di 113 Da, ma hanno tempi di ritenzione diversi. Inoltre, il sequenziamento di Edman può essere eseguito su macchie PVDF da gel 1D e 2D, che consente il sequenziamento N-terminale delle proteine nella miscela. Tuttavia, il sequenziamento della degradazione di Edman non sarà disponibile quando il peptide il cui N-terminale è stato modificato chimicamente, come l’acetilazione. E poiché il PITC non può reagire con acido non-α-amminoacido, il sequenziamento si fermerà se si incontra un acido non-α-amminoacido come l’acido isoaspartico. Inoltre, le proteine più grandi non possono essere sequenziate dal sequenziamento di Edman.
A Creative Proteomics, la nostra piattaforma professionale di sequenziamento delle proteine fornisce l’analisi della sequenza N-terminale mediante degradazione di Edman o spettrometria di massa (MS), con vantaggi complementari tra loro. Il nostro team di esperti con una vasta esperienza può aiutarti a capire cosa stai cercando di indagare e soddisfare le tue esigenze. Inoltre, possiamo fornire altri servizi di identificazione delle proteine, tra cui:
- Peptide mass fingerprinting (PMF) service
- Sequence analysis of peptides or proteins
- De novo protein sequence analysis service
- Shotgun protein identification service
- Membrane proteomics service