Frontiere della Microbiologia
Introduzione
Il Astroviridae famiglia contiene non avvolto, positivo senso, single-stranded RNA virus all’interno di due generi, Mamastrovirus e Avastrovirus, che infettano gli uccelli e i mammiferi, rispettivamente. Attualmente, il Comitato internazionale sulla tassonomia dei virus (International Committee on Taxonomy of Viruses , 2018) riconosce 19 specie, vale a dire Mamastrovirus-1 attraverso -19, all’interno del genere Mamastrovirus; tuttavia, ci sono numerosi ceppi in attesa di classificazione, alcuni dei quali sono provvisoriamente considerati nuove specie (Donato e Vijaykrishna, 2017).
Dal 2010, diversi astrovirus sono stati sempre più riconosciuti come neuroinvasivi in varie specie di mammiferi, inclusi gli esseri umani (Quan et al., 2010; Naccache et al., 2015), visone (Blomström et al., 2010), bovini (Li et al., 2013), pecore (Pfaff et al., 2017), e maiali (Boros et al., 2017). Dopo il riconoscimento iniziale di encefalite bovina associata all’astrovirus nei bovini degli Stati Uniti (Li et al., 2013), uno studio retrospettivo in casi di encefalite bovina sporadica di eziologia indeterminata dalla Svizzera ha rivelato che questo astrovirus neuroinvasivo era passato inosservato per decenni (Selimovic-Hamza et al., 2016). Sebbene l’epidemiologia e le vie di trasmissione di questi astrovirus siano sconosciute, la trasmissione incrociata tra specie è stata suggerita in base all’elevato livello di identità (>98%), condiviso tra astrovirus neuroinvasivi bovini e ovini a livello di nucleotidi e amminoacidi (Boujon et al., 2017).
Astrovirus bovini (BoAstVs), denominati BoAstV-NeuroS1 (Li et al., 2013) e BoAstV-CH13 (Bouzalas et al., 2014), sono stati inizialmente trovati nel cervello di bovini con encefalite non suppurativa negli Stati Uniti e in Svizzera, rispettivamente. Nonostante la diversa nomenclatura, entrambi i virus rappresentano la stessa specie di genotipo (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a) che è ancora in attesa di classificazione ufficiale da parte dell’ICTV. Nel 2015, un ceppo di BoAstV precedentemente sconosciuto, denominato BoAstV-CH15, è stato identificato nel cervello di mucche con encefalite in Svizzera. Il confronto filogenetico completo del genoma ha rivelato una relazione più stretta di BoAstV-CH15 con un astrovirus ovino (OvAstV) che con BoAstV-CH13 (Seuberlich et al., 2016). La coinfezione con BoAstV-CH13 e BoAstV-CH15 è stata anche documentata in un caso (Seuberlich et al., 2016). Lo stesso anno in Germania, Schlottau et al. (2016) ha riportato un nuovo astrovirus, vale a dire BoAstV-BH89/14, in una mucca con encefalite, che era più strettamente correlata a OvAstV e BoAstV-CH15. Successivamente, BoAstV-CH13 / NeuroS1 è stato identificato nel 2017 in casi di encefalite bovina nel Canada orientale e occidentale (Spinato et al., 2017; Selimovic-Hamza et al., 2017b). In 2018, un romanzo neuroinvasivo BoAstV strettamente correlato con il nordamericano e l’europeo BoAstV-NeuroS1/BoAstV-CH13, è stato identificato in un manzo con encefalomielite non suppurativa in Giappone, e l’insorgenza di ricombinazione intra-genotipica tra i ceppi nordamericani ed europei è stata suggerita (Hirashima et al., 2018).
Mentre casi di encefalite associata all’astrovirus sono stati segnalati in Nord America, Europa e Asia, la loro presenza non è mai stata documentata nell’emisfero australe. Qui descriviamo un caso di encefalite associata all’astrovirus nei bovini in Uruguay, che amplia la distribuzione geografica e la diversità genetica degli astrovirus neuroinvasivi e fornisce prove filogeografiche che suggeriscono che questo virus è stato introdotto nelle Americhe dall’Europa e successivamente diffuso in Asia.
Materiali e metodi
Storia e segnalazione
Nel giugno 2018, un manzo Holstein di 22 mesi in un gruppo di 37 manzi in una fattoria di hectare 300 ettari a Colonia, in Uruguay, ha sviluppato sings neurologici progressivi tra cui comportamento insolito, camminata senza scopo, cerchio, atassia, movimenti della lingua ripetitivi e non coordinati e recumbency. La mandria pascolava su un pascolo annuale di avena e veniva integrata con insilato di mais. Una diagnosi clinica presuntiva di listeriosi cerebrale da parte del veterinario ha richiesto il trattamento con penicillina e streptomicina, tuttavia l’animale è morto spontaneamente dopo un decorso clinico della durata di 3 giorni.
Esame patologico, ibridazione in situ (ISH) e immunoistochimica (IHC)
La testa del manzo è stata rimossa dalla carcassa e sottoposta al Laboratorio diagnostico veterinario dell’INIA (Animal Health Platform) per il lavoro diagnostico. Metà del cervello, un breve segmento del midollo spinale cervicale prossimale (C1), del ganglio trigeminale e della radice del nervo trigemino, della ghiandola salivare, del linfonodo retrofaringeo, dell’orofaringe, dell’esofago, della lingua e del muscolo scheletrico, sono stati fissati per immersione in formalina tamponata neutra al 10% per 48-72 h. I tessuti sono stati trattati di routine per l’istologia, incorporati in paraffina, sezionati Macchie di grammo.
L’ISH cromogenico è stato eseguito manualmente su sezioni da 5 µm di tronco cerebrale, cervello e cervelletto (FFPE) fissati in formalina, incorporati in paraffina su vetrini Superfrost Plus (Thermo Fisher Scientific, Pittsburgh, PA, Stati Uniti) utilizzando il kit di analisi rosso RNAscope 2.5 (Cat # 322360, Advanced Cell Diagnostics, Hayward, CA, Stati Uniti) e la sonda BoAstV Cat. #406921. La sonda è composta da 20ZZ coppie targeting regione 5232-6180 del virus (GenBank KF233994. 1). Ogni sezione di tessuto da 5 µm è stata pretrattata con calore e proteasi prima dell’ibridazione della sonda per 2 ore a 40°C ed elaborata secondo le raccomandazioni dei produttori. I controlli negativi utilizzati per la convalida del segnale includevano una sonda non correlata (GC-content matched) eseguita su sezioni seriali e sondando il tessuto da animali non infetti. I vetrini sono stati contrastati con ematossilina e montati con EcoMount (Biocare Medical, Concord, CA, Stati Uniti).
Inoltre, IHC è stato eseguito in sezioni FFPE di tronco cerebrale, cervello e cervelletto, come precedentemente descritto, per l’identificazione del virus del Nilo occidentale (WNV, Flavivirus) (Palmieri et al., 2011), virus della rabbia (Lyssavirus) (Stein et al., 2010), e Chlamydia spp. (Giannitti et al., 2016) antigeni.
Virologia molecolare
L’estrazione dell’acido nucleico è stata effettuata da un campione aggregato di cervello congelato (-20°C) utilizzando MagMAX Nucleic Acid Isolation Kit® (Thermo Fisher Scientific). Per il rilevamento di astrovirus, la trascrizione inversa (RT) è stata eseguita con RevertAid Reverse Transcriptase® (Thermo Fisher Scientific) e random hexamer primer (Qiagen). La PCR è stata eseguita da cDNA utilizzando MangoMix® (Bioline) e primer che amplificano un frammento di 432 nucleotidi del gene della polimerasi astrovirus (Tse et al., 2011). Il prodotto PCR è stato visualizzato in gel di agarosio al 2%, purificato utilizzando PureLink® Quick Gel Extraction e PCR Purification Combo Kit (Invitrogen) e sequenziato presso Macrogen Inc. (Seoul, Corea del Sud). Per astrovirus intero genoma amplificazione, Maxima H Meno trascrittasi inversa (Thermo Fisher Scientific)e oligo(dT) 18 per obtention di cDNA, e MangoMix® (Bioline) o Ranger DNA Polimerasi (Bioline) con primer descritto da Hirashima et al., 2018, sono stati utilizzati. I prodotti PCR sono stati visualizzati in gel di agarosio 1-2%, purificati e sequenziati come menzionato sopra. Assemblaggio sequenza è stata condotta con SeqMan (Lasergene 8, DNASTAR). Ventisei sequenze genomiche complete di astrovirus neuroinvasivo da bovini, ovini, suini, umani e visoni e astrovirus bovino enterico disponibili in GenBank sono stati scaricati e allineati utilizzando Clustal W nel software MEGA 7 (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) è stato usato per determinare il modello più adatto dell’evoluzione della sequenza (SYM + I + G4) e per costruire un albero filogenetico di massima verosimiglianza con le sequenze quasi complete del BoAstV rilevate in questo caso e quelle sequenze complete scaricate da GenBank, usando bootstrap come metodo statistico per valutare la robustezza dei cladi. La trama di somiglianza è stata eseguita con il software SimPlot (Lole et al., 1999). Le distanze P a livello di aminoacidi dell’ORF2 sono state stimate con il software MEGA 7 (Kumar et al., 2016).
Inoltre, è stata eseguita un’analisi filogeografica bayesiana con il pacchetto BEAST v1.8. 4 (Drummond et al., 2012), usando: il codice completo regione di BoAstV CH13/NeuroS1 lignaggio, ORF1ab (non di geni strutturali), ORF2 (gene strutturale), ORF1a (proteasi) e un parziale ORF1b (polimerasi regione genomica, per il quale Canadese ceppi erano disponibili), con tutte le sequenze disponibili in GenBank (ultimo di adesione è il 18 aprile, 2019), per determinare il tasso evolutivo, l’età/anni di antenati comuni, e il percorso più probabile di circolazione virale da paese a paese (la Svizzera, Uruguay, Stati Uniti, Canada e Giappone). La mancanza di ricombinazione nel set di dati è stata determinata utilizzando il programma di rilevamento della ricombinazione 4. Il modello di sostituzione che si adatta meglio a ciascun allineamento è stato determinato utilizzando il software MEGA 7 attraverso i valori Bayesian Information criterion (BIC) e la struttura temporale di ciascun set di dati è stata valutata utilizzando TempEst (Rambaut et al., 2016). L’orologio molecolare lognormal relaxed con analisi bayesiana dello Skyline è stato selezionato da Bayes Factor tra le diverse combinazioni di orologi molecolari e priori di alberi coalescenti utilizzati. Il paese di rilevamento è stato utilizzato come tratto. La lunghezza della catena Markov Monte Carlo era di 100 milioni di generazioni, garantendo la convergenza dell’analisi, valutata in Tracer v1. 6.0, e la probabilità posteriore è stata utilizzata per valutare i cladi. Il maximum clade Credibility tree (MCCT) è stato ottenuto utilizzando il software TreeAnnotator di BEAST e visualizzato in FigTree v1.4.3.
Infine, il DNA estratto dal cervello congelato è stato elaborato mediante PCR per la rilevazione di herpesvirus bovini 1 e 5 (BHV-1 e -5), come precedentemente descritto (Ashbaugh et al., 1997).
Batteriologia
Campioni freschi di cervello e tronco cerebrale sono stati regolarmente elaborati per colture batteriche aerobiche in sangue e agar MacConkey e colture selettive per Listeria monocytogenes (Al-Zoreky e Sandine, 1990).
Risultati e discussione
I segni clinici e i risultati epidemiologici nel caso qui descritto, anche se non specifici, erano simili a quelli descritti in altri casi di encefalite bovina associata all’astrovirus, che di solito è descritta come sporadica (Selimovic-Hamza et al., 2016), con una varietà di deficit neurologici (Deiss et al., 2017), con una durata dei segni clinici che varia tipicamente da 1 giorno a 3 settimane (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
L’esame macroscopico del cervello, del segmento C1 del midollo spinale e di altri tessuti della testa non ha rivelato lesioni anatomiche grossolane significative. Istologicamente, c’era meningoencefalomielite da moderata a grave, linfocitica, istiocitica e plasmacitica che interessava il telencefalo (compreso l’emisfero cerebrale e l’ippocampo), il tronco cerebrale e l’unico segmento esaminato del midollo spinale. Le lesioni erano prevalentemente distribuite nella materia grigia e limitando le aree della sostanza bianca. Nelle aree affette si sono verificate cuffing perivascolare e infiammazione linfoplasmacitica e istiocitica e necrosi neuronale / neuronofagia con gliosi nel neuropile adiacente. C’era una satellitosi di neuroni necrotici affetti (Figure 1A–D). Le lesioni erano molto meno frequenti e gravi nel parenchima cerebellare, sebbene vi fosse una leptomeningite cerebellare moderata multifocale. Non sono stati trovati batteri intralesionali con macchie H&E e Gram. Non sono state riscontrate alterazioni istologiche significative negli altri tessuti esaminati.

Figura 1. Lesioni istologiche nel tronco cerebrale (A,B) e corteccia cerebrale (C,D) e rilevazione di RNA di BoAstV nella corteccia cerebrale (E, G). Le immagini (A-D) sono sezioni del cervello macchiate con H & E; le immagini E e G sono sezioni di corteccia cerebrale che dimostrano l’ibridazione usando l’ISH cromogenico usando la sonda specifica di BoAstV, controstinta con ematossilina; l’immagine F è una sezione seriale della sonda DAPB della corteccia cerebrale (controllo negativo), controstinta con ematossilina. (A) Uno spazio perivenulare è notevolmente ampliato dalle cellule infiammatorie (principalmente linfociti e istiociti) che infiltrano anche il neuropile adiacente. (B) Il neurone al centro ha perikaryon ipereosinofilo e karyorrhexis (necrosi) e il corpo neuronale è circondato da un numero maggiore di cellule gliali (satellitosi) e infiammatorie. (C) La leptomeninge è infiltrata dai linfociti e dagli istiociti. (D) Una regione della corteccia cerebrale con neuroni ipereosinofili multipli (necrotici) e un grande vaso con polsini linfocitari perivascolari. (E) In una sezione seriale della corteccia cerebrale, l’abbondante etichettatura del RNA di BOASTV intracitoplasmatico è rappresentata da una forte deposizione granulare di cromogeno rosso all’interno del citoplasma neuronale del soma e delle estensioni neuronali (E, G), che non è presente con l’ibridazione utilizzando la sonda di controllo negativa (F).
Una infezione virale neuroinvasiva è stata sospettata dopo l’esame istologico del sistema nervoso centrale. I bovini con encefalite sono preoccupanti perché molti neuropatogeni dei ruminanti sono zoonotici (Cantile e Youssef, 2016); pertanto, una diagnosi di encefalite dovrebbe richiedere ampi test di laboratorio per lo screening di agenti infettivi quando possibile. Nel caso qui descritto, IHCs per WNV, virus della rabbia e Chlamydia spp., e PCR per BHV-1 e -5 furono tutti negativi, e nessun batterio patogeno fu coltivato da tessuto cerebrale. Poiché il manzo aveva < 2 anni e non sono stati osservati cambiamenti spongiformi nel tronco cerebrale, l’animale non è stato testato per l’encefalopatia spongiforme bovina (BSE), che è una malattia esotica dei bovini adulti che non è mai stata segnalata in Uruguay. Inoltre, la BSE non è infiammatoria (Cantile e Youssef, 2016).
L’ibridazione in situ è stata eseguita utilizzando una sonda generata da BoAstV-NeuroS1, e c’era un’ibridazione della sonda abbondante all’interno e limitata al citoplasma dei neuroni nell’emisfero cerebrale e nell’ippocampo (Figure 1E–G). In queste aree l’ibridazione sonda colocalizzata con neuroni necrotici e regioni di gliosi, senza ibridazione sonda rilevabile nelle cellule gliali, o cellule infiammatorie dei polsini perivascolari. Nessun acido nucleico virale è stato rilevato da ISH nel cervelletto, che aveva solo lesioni infiammatorie minime nel parenchima ma leptomeningite moderata, o il tronco cerebrale, comprese le sezioni con grave infiammazione. Ciò significa che, topograficamente, la rilevazione della distribuzione virale da parte di ISH è stata più limitata dell’encefalite nelle sezioni esaminate, che è stata occasionalmente descritta nei casi di encefalite associata a vanagloria V-CH13/NeuroS1 nei bovini (Selimovic-Hamza et al., 2017a, b). Una ragione di questa occasionale mancanza di rilevamento dell’RNA virale nelle aree lesionate del cervello potrebbe essere il limite di rilevamento dell’ISH, o la clearance del virus nelle aree infiammate del cervello al momento della morte, come precedentemente suggerito (Selimovic-Hamza et al., 2017b). Come previsto, nessuna ibridazione sonda è stata rilevata da ISH nel tessuto cerebrale utilizzato come controllo negativo.
L’astrovirus è stato rilevato nel cervello da RT-PCR. L’analisi quasi completa della sequenza del genoma ha rivelato un ceppo Mamastrovirus all’interno del clade CH13/NeuroS1, che abbiamo chiamato BoAstV-Neuro-Uy, la sequenza è stata depositata in GenBank con il numero di adesione MK386569. L’analisi filogenetica ha rivelato la vicinanza con altri astrovirus neuroinvasivi all’interno del clade Virginia/Human-Mink-Ovine (VA/HMO) (Figura 2), che contiene gli astrovirus neuroinvasivi più noti (Hirashima et al., 2018; Reuter et al., 2018). La sequenza quasi completa di BoAstV-Neuro-Uy è 6427 bp in lunghezza e ha un’identità di sequenza del 94% con ceppo KagoshimaSR28-462. BoAstV-Neuro-Uy ha caratteristiche simili ad altri ceppi di lignaggio CH13 / NeuroS1: una regione 5’UTR di 51 nt, ORF1a (proteasi) di 861 aminoacidi (aa), ORF1b di 523 aa (RNA polimerasi RNA-dipendente) e ORF2 di 758 aa (proteina capside). Sfortunatamente, il 3 ‘ UTR non poteva essere sequenziato, ma si presume che una coda poli(A) sia presente perché oligo(dT)18 è stato usato per ottenere cDNA. Inoltre, è presente la sequenza eptamericana AAAAAAC, un segnale di frameshift ribosomiale. Le distanze P a livello aminoacidico dell’ORF2 hanno confermato l’assegnazione di questo ceppo al clade CH13/NeuroS1. P-distanze < 0.35 tra BoAstV-Neuro-Uy e altri membri di questo clade (Tabella 1) supporterebbe una classificazione di questi ceppi virali all’interno di una stessa specie; Mamastrovirus-13 è stato recentemente proposto da altri autori (Donato e Vijaykrishna, 2017; Hirashima et al., 2018), anche se l’assegnazione di specie definita da parte dell’ICTV è in sospeso. La sonda utilizzata per l’ISH, generata da BoAstV-NeuroS1, aveva un’identità di sequenza del 92,7% con BoAstV-Neuro-Uy.
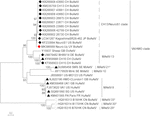
Figura 2. Analisi filogenetica di sequenze nucleotidiche a lunghezza intera con metodo di massima verosimiglianza. BoAstV-Neuro-Uy è contrassegnato da un rombo rosso. Altre sequenze di astrovirus neuroinvasivi bovini sono contrassegnate con rombi neri, le sequenze di astrovirus neuroinvasivi non bovini sono contrassegnate con triangoli neri e le sequenze di astrovirus bovini enterici sono contrassegnate con cerchi bianchi. Le specie di Mamastrovirus sono mostrate; gli asterischi indicano specie che devono ancora essere riconosciute dall’ICTV. I ceppi all’interno del clade CH13/NeuroS1 non ancora assegnati a una specie dall’ICTV sono identificati con una linea tratteggiata. I valori di Bootstrap sono indicati nei nodi. CH, Svizzera; JP, Giappone; US, Stati Uniti d’America; UY, Uruguay; GB, Regno Unito di Gran Bretagna; DE, Germania; SE, Svezia; FR, Francia; CN, Cina. Vanto, astrovirus bovino; OvAstV, astrovirus ovino; PoAstV, astrovirus porcino; HuAstV, astrovirus umano; MiAstV, astrovirus visone.
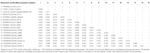
Tabella 1. Stime della divergenza evolutiva a livello di aminoacidi della regione ORF2 completa tra sequenze di Mamastrovirus-13 bovino e ovino e ceppi all’interno del clade CH13/NeuroS1 non ancora assegnati a una specie dal Comitato internazionale sulla tassonomia dei virus (ICTV).
Studi basati su esami neuropatologici e sulla rilevazione di acido nucleico e proteine dell’astrovirus hanno concluso che esiste una probabile relazione causale tra infezione da astrovirus e malattia neurologica e lesioni nei bovini (Selimovic-Hamza et al., 2017a; Reuter et al., 2018). Per quanto ne sappiamo, l’encefalite associata all’astrovirus non è stata ancora riprodotta sperimentalmente. Ciò richiederebbe l’isolamento degli astrovirus neuroinvasivi dai casi clinici, che non è stato tentato nel nostro caso.
La fonte di vanagloria in questo caso non è stata determinata. Tuttavia, il bestiame e la fauna selvatica del bacino idrico dovrebbero essere considerati, poichè i bovini sono stati allevati nelle vaste circostanze all’aperto. L’animale colpito era stato acquistato e trasferito nella fattoria a febbraio 2018 insieme ad altri manzi 9. Sfortunatamente, il proprietario ha rifiutato ulteriori campionamenti e test di altri animali nella proprietà e un’indagine epidemiologica più dettagliata. Nessuno degli altri animali del gruppo aveva sviluppato una malattia neurologica ad agosto 2018, l’ultima volta che il veterinario è stato contattato. Una stagionalità dall’inizio dell’inverno fino alla fine della primavera è stata suggerita per i casi di encefalite associata all’astrovirus in Svizzera (Selimovic-Hamza et al., 2016). È interessante notare che il caso qui descritto si è verificato a giugno, corrispondente al periodo di transizione autunno-inverno nell’emisfero australe.
Mentre gli astrovirus neurotropi sono stati identificati in Nord America (Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) e Asia (Hirashima et al., 2018), la loro presenza non è mai stata segnalata nell’emisfero australe, quindi questa comunicazione amplia la distribuzione geografica dell’encefalite associata all’astrovirus. Per valutare se il ceppo virale rilevato in Uruguay potrebbe aver avuto origine in Europa, Nord America o Asia, abbiamo stimato il tasso evolutivo ed eseguito un’analisi filogeografica utilizzando sequenze neuroinvasive di BoAstV disponibili in GenBank. Il tasso evolutivo stimato utilizzando la regione di codifica completa era 4.27 × 10-4 (95% più alta densità di probabilità-HPD-, 2,19–6,46 × 10-4) sostituzioni nucleotidiche / sito / anno, che è previsto per un virus a RNA (Jenkins et al., 2002), ma inferiore a quello stimato per gli astrovirus umani enterici (Babkin et al., 2012, 2014). La regione ORF1ab ha mostrato un tasso evolutivo simile (4,20 × 10-4, 95% HPD 1,66–6,46 × 10-4 sostituzioni/sito/anno) come regione codificante completa, mentre ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6, 46 × 10-4 sostituzioni/sito/anno) e ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 sostituzioni / sito / anno) ha mostrato un tasso evolutivo leggermente più veloce e la regione genomica della polimerasi parziale (ORF1b) ha mostrato un tasso evolutivo leggermente più lento (5,39 × 10-4, 95% HPD 6,41 × 10-7-1, 10 × 10-3 sostituzioni/sito/anno).
Come determinato dall’analisi filogeografica con la regione codificante completa, e mostrato nel MCTT (Figura 3), ci sono due sottolinee (CH13 e NeuroS1) basate su ceppi di riferimento, che hanno un antenato comune. Il più recente antenato comune di queste sottolinee (lignaggio CH13/NeuroS1) sorse in Europa approssimativamente nel 1885 (95% HPD, 1794-1940). All’inizio del 1900, i due sotto-lignaggi divergevano, il sotto-lignaggio CH13 rimase in circolazione in Europa, mentre il sotto-lignaggio NeuroS1 si diffuse in America e in Asia. Lo scenario più probabile è che il sottolinea NeuroS1 sia stato introdotto in Uruguay dall’Europa intorno all’anno 1921 (95% HPD, 1849-1967), presumibilmente attraverso il commercio di bestiame, poi si è diffuso in Nord America e in seguito in Giappone (Figura 3). A causa della limitazione del numero di sequenze disponibili in GenBank, che potrebbe aver distorto l’analisi, i risultati ottenuti utilizzando il codice completo regione sono stati confrontati con quelli ottenuti con altre regioni genomiche (ORF1ab, ORF2, ORF1a, e ORF1b) disponibile per un maggior numero di ceppi (cioè, Canadese ceppi). In tutte le analisi lo scenario più probabile è che l’introduzione del virus in Uruguay sia avvenuta dall’Europa (Cifre supplementari S1A–D). Inoltre, la data stimata per questa introduzione, ottenuta con l’ORF1ab e la regione genomica della polimerasi parziale (ORF1b) (Cifre supplementari S1A,D), era simile a quella ottenuta con la regione di codifica completa, mentre la data stimata di introduzione ottenuta con ORF2 e ORF1a era precedente ma con un intervallo HPD più ampio del 95% (Cifre supplementari S1B,C). Un’introduzione del sottolinea NeuroS1 direttamente in Canada dall’Europa, con successiva diffusione negli Stati Uniti e in Giappone, è anche plausibile, s mostrato nella figura supplementare S1D.

Figura 3. Albero di massima credibilità del clade ottenuto dall’analisi della regione di codifica a lunghezza intera. Il colore dei rami rappresenta il paese più probabile in cui gli antenati circolavano, i valori di probabilità posteriori sono mostrati nei rami e i numeri in ogni nodo rappresentano gli anni di origine per ogni clade con l’intervallo HPD 95%. Le sottolinee sono indicate con etichette.
Sono necessarie ulteriori indagini per valutare la distribuzione geografica, i meccanismi patogeni (in particolare i meccanismi di trasmissione e ingresso), l’epidemiologia molecolare e la potenziale trasmissione interspecie di astrovirus neuroinvasivi.
Disponibilità dei dati
I set di dati generati per questo studio possono essere trovati in GenBank, MK386569.
Contributi dell’autore
FG, RDC e MC hanno contribuito alla concezione dello studio. FG e RDC hanno eseguito l’esame patologico e il campionamento. PP ha eseguito l’ibridazione in situ. FU ha eseguito l’immunoistochimica. LM, RC e MC hanno eseguito test di virologia molecolare. MC ha eseguito la sequenza e le analisi filogeografiche e le figure associate. FG e PP hanno ottenuto le immagini istologiche. MF ha eseguito le colture batteriche. FG e MC hanno scritto la prima bozza del manoscritto. RDC, PP, FU, LM, MF e RC hanno scritto sezioni del manoscritto. Tutti gli autori hanno contribuito alla revisione del manoscritto, hanno letto e approvato la versione presentata.
Finanziamento
Questo lavoro è stato finanziato da Sovvenzioni PL-015 N-15156 da INIA e 158 dal “Programa de Iniciación a la Investigación 2017 “da” Comisión Sectorial de Investigación Científica ” (CSIC). MC e RDC riconoscono il sostegno dell ‘”Agenzia Nazionale per la Ricerca e l’Innovazione” (ANII) e dell’INIA, rispettivamente, attraverso borse di dottorato. FG riconosce il sostegno di ANII attraverso la concessione di mobilità MOV_CA_2018_1_150021.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Il revisore TS ha dichiarato una passata co-paternità con uno degli autori PP all’editor di gestione.
Ringraziamenti
Gli autori ringraziano Yisell Perdomo e Cecilia Monesiglio di INIA, e Karen Sverlow e Juliann Beingesser di CAHFS per l’assistenza tecnica.
Materiale supplementare
Il materiale supplementare per questo articolo può essere trovato online all’indirizzo: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
FIGURA S1 / Maximum clade credibility trees (MCCTs) ottenuti dall’analisi dell’ORF1ab a lunghezza intera (A), ORF2 a lunghezza intera (B), ORF1a a lunghezza intera (C) e ORF1b parziale (D). Il colore dei rami rappresenta il paese più probabile in cui gli antenati circolavano, i valori di probabilità posteriori sono mostrati nei rami e i numeri in ciascun nodo rappresentano gli anni di origine per ciascun clade con l’intervallo HPD del 95%. Le sottolinee sono indicate con etichette.
Note in calce
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., e Sandine, W. E. (1990). Mezzo altamente selettivo per l’isolamento di Listeria monocytogenes dal cibo. Appl. Ambiente. Microbiolo. 56, 3154–3157.
PubMed Abstract / Google Scholar
Nel 1997, dopo la morte del padre, si trasferì a Parigi, dove si trasferì a Parigi, dove rimase fino al 1997. Rilevazione specifica dello spargimento e della latenza dell’herpesvirus bovino 1 e 5 mediante una reazione a catena della polimerasi nidificata. J. Vet. Diagram. Investire. 9, 387–394. doi: 10.1177/104063879700900408
PubMed Abstract / CrossRef Full Text / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., e Tikunova, N. V. (2014). Analisi di ricombinazione basata sui genomi completi HAstV-2 e HastV-4. Infettare. Genet. Evol. 22, 94–102. doi: 10.1016 / j. meegid.2014.01.010
PubMed Abstract / CrossRef Testo completo / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V., e Tikunova, N. V. (2012). Alto tasso evolutivo di astrovirus umano. Infettare. Genet. Evol. 12, 435–442. doi: 10.1016 / j. meegid.2012.01.019
PubMed Abstract / CrossRef Testo completo / Google Scholar
Blomström, A. L., Widén, F., Hammer, A. S., Belák, S., e Berg, M. (2010). Rilevamento di un nuovo astrovirus nel tessuto cerebrale di visone affetto da sindrome di visone tremante mediante l’uso di metagenomica virale. J. Clin. Microbiolo. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
PubMed Abstract | CrossRef Testo completo / Google Scholar
Boros, Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et al. (2017). Focolai di astrovirus neuroinvasivo associati a encefalomielite, debolezza e paralisi tra suini svezzati, Ungheria. Emerg. Infettare. Dis. 23, 1982–1993. doi: 10.3201 | eid2312. 170804
PubMed Abstract / CrossRef Full Text / Google Scholar
Boujon, C. L., Koch, M. C., Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Indicazione della trasmissione incrociata di astrovirus associata a encefalite negli ovini e nei bovini. Emerg. Infettare. Dis. 23, 1604–1608. doi: 10.3201/eid2309.170168
PubMed Abstract / CrossRef Testo completo / Google Scholar
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., e Seuberlich, T. (2016). Caratterizzazione molecolare basata su genoma completo degli astrovirus bovini associati all’encefalite. Infettare. Genet. Evol. 44, 162-168. doi: 10.1016 / J. meegid.06/052/2016
PubMed Abstract / CrossRef Full Text / Google Scholar
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C.,Zurbriggen, A., Vandevelde, M., et al. (2014). Astrovirus neurotropico nei bovini con encefalite non suppurativa in Europa. J. Clin. Microbiolo. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
PubMed Abstract / CrossRef Testo completo / Google Scholar
Cantile, C., e Youssef, S. (2016). “Sistema nervoso”, in Jubb, Kennedy e Palmer’s Pathology of Domestic Animals, 6th Edn, ed. M. G. Maxie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Nel 2017 è stato nominato presidente del Consiglio dei Ministri. Segni clinici neurologici in bovini con encefalite associata ad astrovirus. J. Vet. Stagista. Med. 31, 1209–1214. doi: 10.1111 / jvim.14728
PubMed Abstract / CrossRef Testo completo / Google Scholar
Donato, C., e Vijaykrishna, D. (2017). L’ampia gamma di ospiti e la diversità genetica degli astrovirus di mammiferi e aviari. Virus 9:E102. doi: 10.3390 / v9050102
PubMed Abstract / CrossRef Full Text / Google Scholar
Nel 2012 è stato nominato membro del Consiglio di amministrazione. Phylogenetics bayesiano con BEAUti e la BESTIA 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev / mss075
PubMed Abstract / CrossRef Full Text / Google Scholar
Giannitti, F., Anderson, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Chlamydia pecorum: lesioni fetali e placentari nell’aborto caprino sporadico. J. Vet. Diagram. Investire. 28, 184–189. doi: 10.1177/1040638715625729
PubMed Abstract / CrossRef Testo completo / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Analisi del genoma intero di un nuovo astrovirus bovino neurotropico rilevato in un manzo nero giapponese con encefalomielite non suppurativa in Giappone. Arco. Virol. 163, 2805–2810. doi: 10.1007 / s00705-018-3898-3
PubMed Abstract / CrossRef Testo completo / Google Scholar
Comitato Internazionale sulla tassonomia dei virus (2018). Master Species List (MSL32), Versione 1. Disponibile all’indirizzo: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (accesso a marzo, 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G., e Holmes, E. C. (2002). Tassi di evoluzione molecolare nei virus a RNA: un’analisi filogenetica quantitativa. J. Mol. Evol. 54, 156–165. doi: 10.1007 / s00239-001-0064-3
PubMed Abstract / CrossRef Testo completo / Google Scholar
Nel 2016 si trasferisce a New York. MEGA7: molecular evolutionary genetics analysis versione 7.0 per dataset più grandi. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093/molbev / msw054
PubMed Abstract / CrossRef Full Text / Google Scholar
Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Astrovirus divergente associato a malattia neurologica nei bovini. Emerg. Infettare. Dis. 19, 1385–1392. doi: 10.3201/eid1909.130682
PubMed Astratto | CrossRef Testo Completo | Google Scholar
Lole, K. S., di Bollinger, R. C., Pikkio, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Virus dell’immunodeficienza umana di tipo 1 genomi da sieroconvertitori infettati dal sottotipo C in India, con evidenza di ricombinazione intersubtype. J. Virol. 73, 152–160.
PubMed Abstract / Google Scholar
Naccache, S. N., Peggs, KS, Mattes, FM, Phadke, R., Garson, JA, Grant, P., et al. (2015). Diagnosi dell’infezione neuroinvasiva da astrovirus in un adulto immunocompromesso con encefalite mediante sequenziamento imparziale di nuova generazione. Clin. Infettare. Dis. 60, 919–923. doi: 10.1093 / cid / ciu912
PubMed Abstract / CrossRef Full Text / Google Scholar
Il suo nome deriva dal greco antico, che significa “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”. (2011). Patologia e risultati immunoistochimici dell’infezione da virus del Nilo occidentale in psittaciformes. Veterinario. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
PubMed Abstract / CrossRef Full Text / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Un nuovo astrovirus associato a encefalite e ganglioneurite nelle pecore domestiche. In arrivo. Emerg. Dis. 64, 677–682. doi: 10.1111 / tbed.12623
PubMed Astratto | CrossRef Testo Completo | Google Scholar
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Encefalite da astrovirus in ragazzo con agammaglobulinemia legata all’X. Emerg. Infettare. Dis. 16, 918–925. doi: 10.3201 | eid1606. 091536
PubMed Abstract / CrossRef Full Text / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L., e Pybus, OG (2016). Esplorare la struttura temporale di sequenze eterocrone utilizzando TempEst (formely Path-O-Gen). Virus Evol. 2: vew007. doi: 10.1093/ve / vew007
PubMed Abstract / CrossRef Full Text / Google Scholar
Reuter, G., Pankovics, P., e Boros, A. (2018). Meningoencefalomielite non suppurativa (asettica) associata a infezioni da astrovirus neurovirulenti nell’uomo e negli animali. Clin. Microbiolo. Apoc 31, e00040-18. doi: 10.1128 / CMR.00040-18
PubMed Abstract / CrossRef Testo completo / Google Scholar
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Beer, M., et al. (2016). Rilevamento di un nuovo astrovirus bovino in una mucca con encefalite. In arrivo. Emerg. Dis. 63, 253–259. doi: 10.1111 / tbed.12493
PubMed Abstract / CrossRef Testo completo / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A., e Seuberlich, T. (2017a). Frequenza e fenotipo patologico dell’infezione da astrovirus bovino CH13 / NeuroS1 nei bovini con malattie neurologiche: verso la valutazione della causalità. Virus 9:E12. doi: 10.3390 / v9010012
PubMed Abstract / CrossRef Full Text / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., e Seuberlich, T. (2017b). Infezione da astrovirus bovino nei bovini da foraggio con malattia neurologica nel Canada occidentale. Può. Veterinario. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., e Seuberlich, T. (2016). Rilevazione di astrovirus in casi storici di encefalite bovina sporadica europea, Svizzera 1958-1976. Anteriore. Veterinario. Sic. 3:91. doi: 10.3389 / fvet.2016.00091
PubMed Abstract / CrossRef Testo completo / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identificazione di un secondo astrovirus associato all’encefalite nei bovini. Emerg. I microbi infettano. 5: e5. doi: 10.1038 / emi.2016.5
PubMed Abstract / CrossRef Testo completo / Google Scholar
Spinato, M. T., Vince, A., Cai, H., e Ojkic, D. (2017). Identificazione dell’astrovirus bovino nei casi di encefalite bovina non suppurativa nel Canada orientale. Può. Veterinario. J. 58, 607-609.
Google Scholar
Stein, LT, Rech, RR, Harrison, L., e Brown, CC (2010). Studio immunoistochimico del virus della rabbia all’interno del sistema nervoso centrale delle specie domestiche e della fauna selvatica. Veterinario. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
PubMed Abstract / CrossRef Testo completo / Google Scholar
Trifinopoulos, J., Nguyen, LT, von Haeseler, A., e Minh, B. Q. (2016). W-IQ-TREE: uno strumento filogenetico online veloce per l’analisi della massima verosimiglianza. Acidi nucleici Res. 44, W232–W235. doi: 10.1093 / nar / gkw256
PubMed Abstract / CrossRef Full Text / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Riscoperta e caratterizzazione genomica degli astrovirus bovini. J. Gen. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0.030817-0
PubMed Abstract / CrossRef Full Text / Google Scholar