生物学専攻のためのI
学習成果
- プレmRNAとmRNAの違いを理解する
転写後、真核生物のpre-mrnaは、翻訳される前にいくつかの処理ステップを経なければならない。 真核生物(および原核生物)tRNAおよびrRNAもまた、タンパク質合成機械の構成要素として機能する前に処理を受ける。
mRNA処理
真核生物のpre-mRNAは、翻訳の準備が整う前に広範な処理を受けます。 真核生物のmRNA成熟に関与する追加のステップは、原核生物のmRNAよりもはるかに長い半減期を有する分子を生成する。 真核生物のmRNAは数時間持続するが、典型的なE.coli mRNAは5秒以下持続する。
プレmrnaはまずRNA安定化タンパク質でコーティングされており、プレmRNAが処理されて核から輸出される間に分解から保護されています。 プレmRNA処理の三つの最も重要なステップは、分子の5’と3’末端に安定化およびシグナル伝達因子の添加、および適切なアミノ酸を指定しない介在配列の除去である。 まれに、mRNA転写物は、転写後に「編集」され得る。
5’キャッピング
プレmRNAがまだ合成されている間、7-メチルグアノシンキャップリン酸結合によって成長する転写物の5’末端に追加されます。 この部分(官能基)は、初期のmRNAを分解から保護する。 さらに、蛋白質の統合にかかわる要因はリボソームによって翻訳を始めるのを助けるために帽子を確認します。
3’Poly-A Tail
伸長が完了すると、プレmRNAはAAUAAAコンセンサス配列とGUリッチ配列の間のエンドヌクレアーゼによって切断され、プレmRNA上にAAUAAA配列を残す。 ポリ-aポリメラーゼと呼ばれる酵素は、ポリ-aテールと呼ばれる約200a残基の文字列を追加します。 この修飾は、pre-mRNAを分解からさらに保護し、転写物が細胞質に必要とする細胞因子の輸出を示す。
Pre-mRNAスプライシング
真核生物の遺伝子は、タンパク質コード配列に対応するエクソン(ex-onは発現していることを意味する)と、遺伝子調節に関与している可能性があるが、処理中にプレmRNAから除去されるイントロン(intronは介在する役割を意味する)と呼ばれる介在配列で構成されている。 MRNA中のイントロン配列は機能性タンパク質をコードしない。
イントロンの発見は、原核生物で観察されていたように、プレmrnaがそれ以上の処理なしにタンパク質配列を特定することを期待していた1970年代の研究者に驚きとしてもたらされた。 高等真核生物の遺伝子は、非常に多くの場合、一つ以上のイントロンを含んでいます。 これらの領域は調節配列に対応しているかもしれないが、多くのイントロンを有するか、または非常に長いイントロンを遺伝子に有することの生物学的意義は不明である。 多くのイントロンでプレmrnaを転写するのに時間がかかるため、イントロンが遺伝子発現を遅くする可能性があります。 あるいは、イントロンは、進化を通して古代の遺伝子の融合から残された非機能的な配列残党である可能性があります。 これは、独立したエクソンは、多くの場合、別々のタンパク質サブユニットまたはドメインをエンコードするという事実によってサポートされています。 ほとんどの場合、イントロンの配列は、最終的にタンパク質産物に影響を与えることなく変異させることができる。
プレmRNAのイントロンはすべて、タンパク質合成の前に完全かつ正確に除去する必要があります。 プロセスが一塩基でも誤ると、再結合されたエクソンの読み取りフレームがシフトし、結果として得られるタンパク質は機能不全になる。 イントロンを除去してエクソンを再接続するプロセスをスプライシングと呼びます(図1)。 プレmRNAが核にまだある間、イントロンは取除かれ、低下します。 スプライシングは、イントロンが除去され、エクソンが一塩基の精度と精度で再び結合されることを保証する配列特異的機構によって起こる。 プレmrnaのスプライシングは、スプライソソームと呼ばれるタンパク質とRNA分子の複合体によって行われる。
練習問題
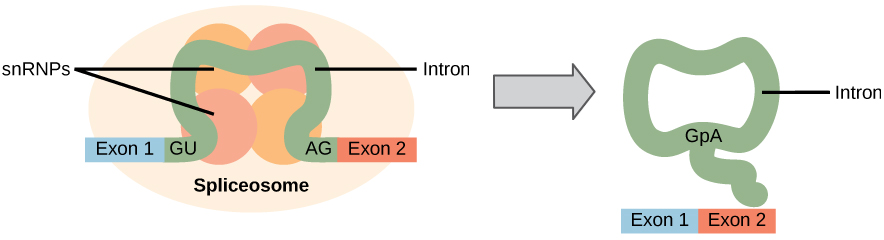
図1. プレmRNAスプライシングは、一次RNA転写産物からイントロンを正確に除去することを含む。 スプライシング過程は、snrnaと呼ばれるタンパク質とRNA分子で構成されるスプライソソームと呼ばれるタンパク質複合体によって触媒される。 スプライソソームは、イントロンの5’および3’末端の配列を認識する。
スプライシングの誤りは、癌や他のヒト疾患に関係している。 どのような種類の突然変異がスプライシングエラーにつながるのでしょうか?
70以上の個々のイントロンが存在することができ、それぞれがスプライシングのプロセスを経る必要があることに注意してください—5’キャッピングとポリ-aテールの添加に加えて—単一の、翻訳可能なmRNA分子を生成するだけです。
トリパノソームにおけるRNA編集
図2。 Trypanosoma bruceiは人間の睡眠病の原因物質です。 この病原体のmrnaは、タンパク質合成が起こる前にヌクレオチドの添加によって修飾されなければならない。 (クレジット:Torsten Ochsenreiterによる作業の変更)
トリパノソームは、ヒトに睡眠病を引き起こす病原体Trypanosoma bruceiを含む原生動物のグループです(図2)。 トリパノソーム、および実質的にすべての他の真核生物は、化学エネルギーを細胞に供給するミトコンドリアと呼ばれるオルガネラを有する。 ミトコンドリアは、自身のDNAを発現する細胞小器官であり、真核生物と包まれた原核生物との共生関係の名残であると考えられている。 トリパノソームのミトコンドリアDNAは、中央の教義に興味深い例外を示す:それらのプレmrnaは、機能的なタンパク質を指定するための正しい情報を持っ 通常、これは、mRNAがいくつかのUヌクレオチドを欠いているためである。 細胞は、これを改善するために、RNA編集と呼ばれる追加のRNA処理ステップを実行する。
ミトコンドリアゲノム中の他の遺伝子は、40-80ヌクレオチドガイドRnaをコードしている。 これらの分子の1つまたは複数は、プレm RNA転写物中のヌクレオチドのいくつかとの相補的塩基対形成によって相互作用する。 しかし、ガイドRNAは、プレmRNAが結合するUヌクレオチドを有するよりも多くのaヌクレオチドを有する。 これらの領域では、ガイドRNAはループする。 ガイドRnaの3’末端は、長いポリ−U尾部を有し、これらのU塩基は、ガイドRnaがループされるプレ−mRNA転写物の領域に挿入される。 このプロセスは、RNA分子によって完全に媒介される。 すなわち、ガイドRnaは、タンパク質ではなく、RNA編集の触媒として機能する。
RNA編集は単なるトリパノソームの現象ではありません。 いくつかの植物のミトコンドリアでは、ほとんどすべてのプレmrnaが編集されています。 RNA編集はまたラット、ウサギおよび人間のようなほ乳類で識別されました。 Pre-mRNA処理におけるこの追加ステップの進化的理由は何でしょうか? 一つの可能性は、ミトコンドリアは、古代の原核生物の残党であることは、遺伝子発現を調節するための同じように古代のRNAベースの方法を持ってい この仮説を支持するために、プレmrnaに行われた編集は、細胞の状態に応じて異なります。 推測的ではあるが、RNA編集のプロセスは、タンパク質の代わりにRNA分子が反応を触媒する役割を果たしていた原始的な時代からのホールドオーバーである可能性がある。
それを試してみてください
貢献します!
このページをもっと見る