Creative Proteomics Blog
sekvensen av aminosyrer i et protein eller peptid kan identifiseres Ved Edman degradering, som ble utviklet Av Pehr Edman. Denne metoden kan merke og spalte peptidet Fra N-terminal uten å forstyrre peptidbindingene mellom andre aminosyrerester. Edman degraderingsreaksjonen ble automatisert i 1967 av Edman Og Beggs. I dag brukes den automatiserte Edman-nedbrytningen (proteinsekvenatoren) mye, og den kan sekvensere peptider opp til 50 aminosyrer.
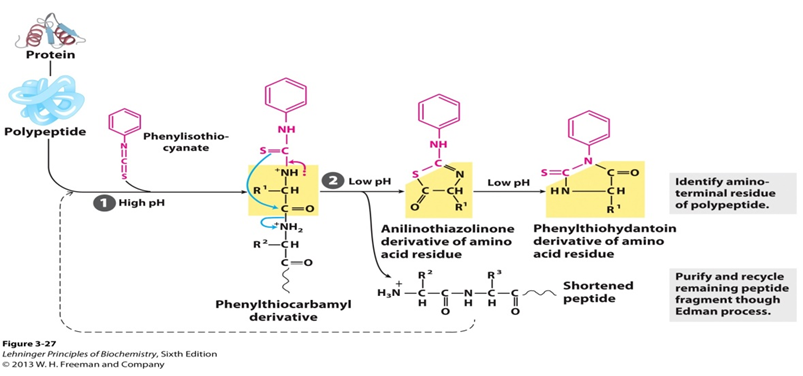
Syklisk nedbrytning av peptider basert på reaksjonen av fenylisotiocyanat med den frie aminogruppen Av Den n-terminale rest, slik at aminosyrer fjernes en om gangen og identifiseres som deres fenyltiohydantoinderivater. Når det gjelder den spesifikke prosessen, reageres et uladet peptid med fenylisotiocyanat (PITC) ved aminoterminalen under mildt alkaliske forhold for å gi et fenyltiokarbamoylderivat (PTC-peptid). Deretter, under sure forhold, angriper tiokarbonylsvovelet av derivatet karbonylkarbonet I Den N-terminale aminosyren. Den første aminosyren spaltes som anilinotiazolinonderivat (ATZ-aminosyre) og resten av peptidet kan isoleres og underkastes neste nedbrytningssyklus. Når det er dannet, er dette tiazolonderivatet mer stabilt enn fenyltiokarbamylderivat. Atz-aminosyren fjernes deretter ved ekstraksjon med etylacerat og omdannes til et fenyltiohydantoinderivat (pth-aminosyre). Og kromatografi kan brukes til å identifisere pth rester generert av hver syklus.
når det gjelder Den automatiserte Edman-nedbrytningen, kan proteiner analyseres ved å bruke dem i oppløsning på ET tfa-filter og deretter lastes på Edman-sekvenseringsinstrumentet. Proteiner i blandinger blir først separert AV 1d eller 2d geler og deretter blottet på EN PVDF-membran. Proteinene er oppdaget Av Coomassie blå, amido svart eller Poncau s flekker og proteiner av interesse kuttet ut OG PVDF membran stykke lastet på Edman sequencer.
med massespektrometri ble utviklet, begynte Bruken Av Edman-nedbrytningssekvensering å synke. Imidlertid forblir det stillbilder metodene for flere typer proteinstrukturelle analyseapplikasjoner. Det kan brukes til å verifisere den n-terminale grensen til rekombinante proteiner eller bestemme N-terminusen til proteaseresistente domener, spesielt når proteinet eller domenet er > 40 til 80 kDa eller ikke lett kan renses. Det kan også brukes til å identifisere det nye N-terminale og proteolytiske spaltningsstedet i proteinfragmentene. I tillegg til noen nye proteiner og peptider der sekvensdatabaser ikke er tilgjengelige FOR MS/MS-databasesøk, Kan Edman-nedbrytning brukes til analyse.
den n-terminale aminosyren i proteinet kan spaltes av. I prosessen identifiserer den første syklusen således den nøyaktige N-terminale aminosyren. I tillegg, fordi de frigjorte aminosyrene identifiseres og kvantifiseres ved kromatografi, kan aminosyrene med identisk molekylvekt identifiseres. For eksempel har isoleucin og leucin en masse på 113 Da, men de har forskjellig retensjonstid. Videre Kan Edman-sekvensering utføres PÅ PVDF-blotter FRA 1D – og 2D-geler, noe som muliggjør N-terminal sekvensering av proteiner i blandingen. Edman degraderingssekvensering vil imidlertid ikke være tilgjengelig når peptidet Hvis N-terminus er kjemisk modifisert, slik som acetylering. OG SIDEN PITC ikke kan reagere med ikke-α-aminosyre, vil Sekvenseringen stoppe hvis det oppstår en ikke-α-aminosyre som isoaspartsyre. Videre kan større proteiner ikke sekvenseres Av Edman-sekvenseringen.
På Creative Proteomics gir vår profesjonelle proteinsekvenseringsplattform N-terminal sekvensanalyse ved Edman-nedbrytning eller massespektrometri (MS), med komplementære fordeler til hverandre. Vårt team av eksperter med lang erfaring kan hjelpe deg å forstå hva du prøver å undersøke og møte dine behov. I tillegg kan vi tilby andre proteinidentifikasjonstjenester, inkludert:
- Peptid masse fingerprinting (PMF) service
- Sekvens analyse av peptider eller proteiner
- de novo protein sekvens analyse service
- Shotgun protein identifikasjon service
- Membran proteomikk service