Biologie voor Majors II
leerresultaten
- begrijp vroegere en nieuwe classificatiesystemen voor virussen
omdat de meeste virussen waarschijnlijk van verschillende voorouders evolueerden, zijn de systematische methodes die wetenschappers hebben gebruikt om prokaryotic en eukaryotic cellen te classificeren niet zeer nuttig. Als virussen “restanten” van verschillende organismen vertegenwoordigen, dan is zelfs genomische of eiwitanalyse niet nuttig. Waarom? Omdat virussen geen gemeenschappelijke genomische sequentie hebben die ze allemaal delen. Bijvoorbeeld, is de 16S rRNA opeenvolging zo nuttig voor het construeren van prokaryote fylogenies van geen nut voor een schepsel zonder ribosomen! Biologen hebben in het verleden verschillende classificatiesystemen gebruikt. Virussen werden aanvankelijk gegroepeerd op basis van gedeelde morfologie. Later, werden de groepen virussen geclassificeerd door het type van nucleic zuur zij bevatten, DNA of RNA, en of hun nucleic zuur enkel-of dubbel-strandde was. Echter, deze eerdere classificatiemethoden gegroepeerd virussen anders, omdat ze waren gebaseerd op verschillende sets van tekens van het virus. De meest meestal gebruikte classificatiemethode vandaag wordt genoemd het Baltimore classificatieschema, en is gebaseerd op hoe boodschappersRNA (mRNA) in elk bepaald type van virus wordt geproduceerd.
oude classificatiesystemen
virussen bevatten slechts enkele elementen waarmee ze kunnen worden geclassificeerd: het virale genoom, het type capside en de omhullingsstructuur van de omhulde virussen. Al deze elementen zijn in het verleden gebruikt voor virale classificatie (Tabel 1 en Figuur 1). De virale genomen kunnen in het type van genetisch materiaal (DNA of RNA) en zijn organisatie (single – of double-stranded, lineair of cirkelvormig, en gesegmenteerd of niet-gesegmenteerd) variëren. In sommige virussen, worden de extra die proteã nen voor replicatie worden vereist direct geassocieerd met het genoom of binnen de virale capside bevatten.
| Tabel 1. Virus Classificatie door het Genoom van de Structuur en de Kern | |
|---|---|
| Kern Classificaties | Voorbeelden |
| RNA | Rabiës virus, retroviruses |
| DNA | Herpesviruses, pokken virus |
| Single-stranded | Rabiës virus, retroviruses |
| Double-stranded | Herpesviruses, pokken virus |
| Lineaire | Rabiës virus, retroviruses, herpesviruses, pokken virus |
| Circulaire | |
| niet-gesegmenteerd: genoom bestaat uit een enkel segment van genetisch materiaal | Parainfluenzavirussen |
| gesegmenteerd: genoom is onderverdeeld in meerdere segmenten | influenzavirussen |
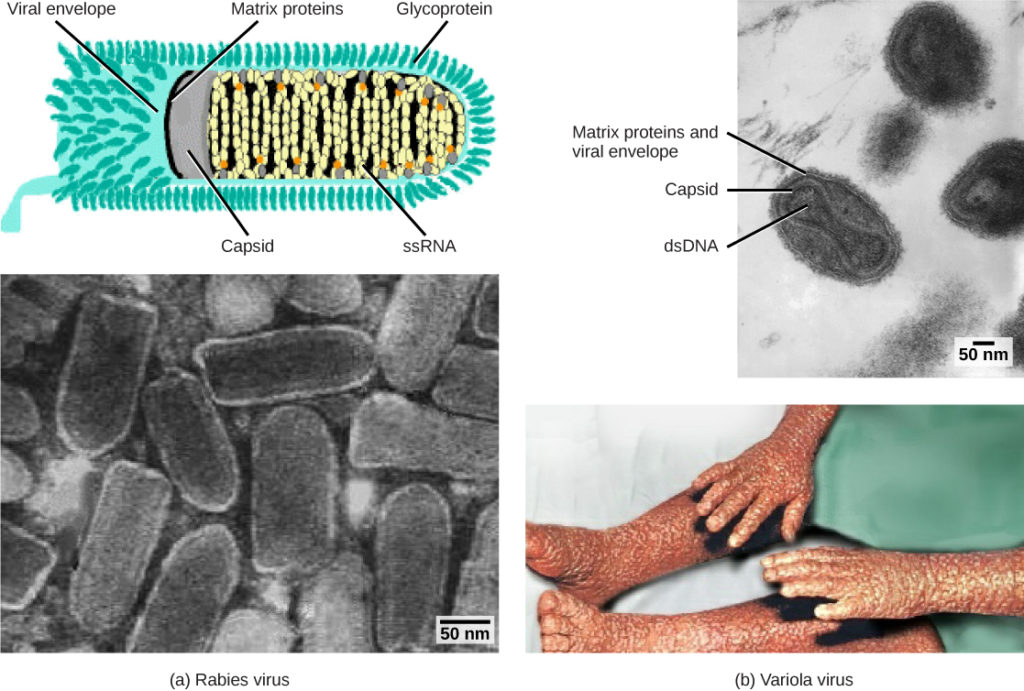
figuur 1. Virussen worden geclassificeerd op basis van hun kern genetisch materiaal en capside ontwerp. a) rabiësvirus heeft een enkelvoudige RNA-kern (ssRNA) en een omhulde spiraalvormige capside, terwijl B) variolavirus, de veroorzaker van pokken, een dubbelstrengs DNA-kern (dsDNA) en een complexe capside heeft. (credit “rabies diagram”: wijziging van het werk door CDC; “rabies micrograph”: wijziging van het werk door Dr. Fred Murphy, CDC; credit “small pox micrograph”: wijziging van het werk door Dr. Fred Murphy, Sylvia Whitfield, CDC; credit “pokken foto”: wijziging van het werk door CDC; schaalgegevens van Matt Russell)
virussen kunnen ook worden ingedeeld op basis van het ontwerp van hun kapsels (Tabel 2 en Figuur 2). Kapsels worden geclassificeerd als naakte icosaëdrale, omhulde icosaëdrale, omhulde spiraalvormige, naakte spiraalvormige en complexe. Het type genetisch materiaal (DNA of RNA) en de structuur ervan (single – of double-stranded, lineair of cirkelvormig, en gesegmenteerd of niet-gesegmenteerd) worden gebruikt om de viruskernstructuren te classificeren (Tabel 2).
| Tabel 2. Virus Classificatie door Capside-Structuur | |
|---|---|
| Capside-Indeling | Voorbeelden |
| Naakt icosahedrale | Hepatitis A virus, polioviruses |
| Gehuld icosahedrale | Epstein-Barr-virus, herpes simplex virus, rubella virus, gele koorts virus, HIV-1 |
| Gehuld spiraalvormige | Influenza virussen, bof-virus, mazelen virus, rabies-virus |
| Naakt spiraalvormige | Tabak mozaïek virus |
| Complex met veel eiwitten; sommige combinaties van icosahedrale en spiraalvormige capside structuren | Herpesviruses, de pokken-virus, hepatitis-B-virus, T4 bacteriofaag |

Figuur 2. Transmissie elektronenmicrografen van verschillende virussen tonen hun structuren. De capside van het (A) poliovirus is naked icosahedral; (b) The Epstein-Barr virus capside is enveloped icosahedral; (c) The bof virus capside is an enveloped helix; (d) the tobacco mosaic virus capside is naked spiraalical; en (e) the herpesvirus capside is complex. (credit a: modification of work by Dr. Fred Murphy, Sylvia Whitfield; credit b: modification of work by Liza Gross; credit c: modification of work by dr. F. A. Murphy, CDC; credit d: modification of work by USDA ARS; credit e: wijziging van het werk van Linda Stannard, afdeling Medische Microbiologie, Universiteit van Kaapstad, Zuid-Afrika, NASA; scale-bar gegevens van Matt Russell)
Baltimore Classification
het meest gebruikte systeem voor virusclassificatie werd ontwikkeld door Nobelprijswinnaar David Baltimore in het begin van de jaren zeventig. naast de hierboven genoemde verschillen in morfologie en genetica, groepeert het Baltimore classification scheme virussen volgens de manier waarop het mRNA wordt geproduceerd tijdens de replicatieve cyclus van het virus.
Groep I-virussen bevatten dubbelstrengs DNA (dsDNA) als hun genoom. Hun mRNA wordt geproduceerd door transcriptie op vrijwel dezelfde manier als met cellulair DNA.
virussen van groep II hebben single-stranded DNA (ssDNA) als hun genoom. Zij zetten hun single-stranded genomen in een dsDNA tussenpersoon om alvorens de transcriptie aan mRNA kan voorkomen.
Groep III-virussen gebruiken dsRNA als hun genoom. De afzonderlijke bundels, en één van hen wordt gebruikt als malplaatje voor de generatie van mRNA gebruikend de RNA-afhankelijke polymerase van RNA die door het virus wordt gecodeerd.
virussen van groep IV hebben ssRNA als genoom met een positieve polariteit. Positieve polariteit betekent dat genomic RNA direct als mRNA kan dienen. De tussenpersonen van dsRNA, genoemd replicatieve tussenpersonen, worden gemaakt in het proces van het kopiëren van genomic RNA. De veelvoudige, bundels van RNA van volledige lengte van negatieve polariteit (vleiend aan positief-vastgelopen genomic RNA) worden gevormd van deze tussenpersonen, die dan als malplaatjes voor de productie van RNA met positieve polariteit, met inbegrip van zowel genomic RNA van volledige lengte als kortere virale mRNAs kunnen dienen.
Groep V-virussen bevatten ssrna-genomen met een negatieve polariteit, wat betekent dat hun sequentie complementair is aan het mRNA. Zoals met groep IV virussen, worden de tussenpersonen dsRNA gebruikt om exemplaren van het genoom te maken en mRNA te produceren. In dit geval, kan het negatief-strandde genoom direct in mRNA worden omgezet. Bovendien, worden de volledige bundels van RNA gemaakt om als malplaatjes voor de productie van het negatief-strandde genoom te dienen.
Groep VI-virussen hebben diploïde (twee kopieën) ssrna-genomen die met behulp van het enzym reverse transcriptase moeten worden omgezet in dsDNA; dsDNA wordt dan getransporteerd naar de kern van de gastheercel en in het gastheergenoom ingevoegd. Dan, kan mRNA door transcriptie van virale DNA worden veroorzaakt dat in het gastheergenoom werd geïntegreerd.
Groep VII-virussen hebben partiële dsDNA-genomen en maken ssRNA-tussenproducten die als mRNA fungeren, maar worden ook weer omgezet in dsDNA-genomen door reverse transcriptase, noodzakelijk voor genoomreplicatie. De kenmerken van elke groep in de Baltimore classificatie zijn samengevat in Tabel 3 met voorbeelden van elke groep.
| Tabel 3. Baltimore Indeling | |||
|---|---|---|---|
| Groep | Kenmerken | Modus van mRNA Productie | Voorbeeld |
| I | Double-stranded DNA | mRNA wordt bewerkt rechtstreeks van de DNA-template | Herpes simplex (herpes-virus) |
| II | Single-stranded DNA | DNA wordt geconverteerd naar double-stranded formulier voor RNA is getranscribeerd | parvo (parvovirus) |
| III | Double-stranded RNA | mRNA transcriptie van het RNA-genoom | Jeugd gastro-enteritis (rotavirus) |
| IV | enkelstrengs RNA (+) | Genoom functies als mRNA | verkoudheid (pircornavirus) |
| V | enkelstrengs RNA (−) | mRNA transcriptie van het genoom van RNA | Hondsdolheid (rhabdovirus) |
| VI | enkelstrengs RNA-virussen met reverse transcriptase | Reverse transcriptase maakt DNA van het genoom van RNA; DNA wordt vervolgens ingebouwd in het gastheer genoom; mRNA wordt getranscribeerd van het geïncorporeerde DNA | humaan immunodeficiëntievirus (HIV).) |
| VII | dubbelstrengs DNA-virussen met reverse transcriptase | het virale genoom is dubbelstrengs DNA, maar viraal DNA wordt gerepliceerd via een RNA-intermediair; het RNA kan direct dienen als mRNA of als sjabloon om mRNA | Hepatitis B-virus (hepadnavirus) te maken) |