Biology for Majors I
leerresultaten
- begrijp het verschil tussen pre-mRNA en mRNA
na transcriptie, moet eukaryotic pre-mRNAs verscheidene verwerkingsstappen ondergaan alvorens zij kunnen worden vertaald. Eukaryotic (en prokaryotic) tRNAs en rRNAs ondergaan ook verwerking alvorens zij als componenten in de machines van de eiwitsynthese kunnen functioneren.
mRNA-verwerking
het eukaryotische pre-mRNA ondergaat een uitgebreide verwerking voordat het klaar is om te worden vertaald. De extra stappen betrokken bij eukaryotic mRNA rijping leiden tot een molecule met een veel langere halfwaardetijd dan prokaryotic mRNA. Eukaryotic mRNAs duren enkele uren, terwijl de typische E. coli mRNA duurt niet meer dan vijf seconden.
Pre-mRNA ‘ s worden eerst gecoat in RNA-stabiliserende eiwitten; deze beschermen het pre-mRNA tegen afbraak terwijl het wordt verwerkt en geëxporteerd uit de kern. De drie belangrijkste stappen van pre-mRNA verwerking zijn de toevoeging van het stabiliseren en signalerende factoren bij de einden 5′ en 3’ van de molecule, en de verwijdering van tussenliggende opeenvolgingen die niet de aangewezen aminozuren specificeren. In zeldzame gevallen kan het mRNA-transcript worden “bewerkt” nadat het is getranscribeerd.
5 ‘- Cap
terwijl het pre-mRNA nog steeds wordt gesynthetiseerd, wordt een 7-methylguanosine-cap toegevoegd aan het 5′ – uiteinde van het groeiende transcript door een fosfaatverbinding. Deze groep (functionele groep) beschermt de ontluikende mRNA tegen degradatie. Bovendien erkennen de factoren betrokken bij eiwitsynthese GLB om te helpen vertaling door ribosomen in werking te stellen.
3 ‘ Poly-a-staart
zodra de elongatie is voltooid, wordt de pre-mRNA gesplitst door een endonuclease tussen een aauaaa-consensusreeks en een Gu-rijke sequentie, waarbij de AAUAAA-sequentie op de pre-mRNA blijft. Een enzym genaamd poly – a polymerase voegt dan een string van ongeveer 200 A residuen, genaamd de poly-a staart. Deze wijziging beschermt verder pre-mRNA tegen degradatie en signaleert de uitvoer van de cellulaire factoren die het transcript aan het cytoplasma nodig heeft.
Pre-mRNA Splicing
eukaryotische genen zijn samengesteld uit exonen, die overeenkomen met eiwitcoderende sequenties (ex-on betekent dat ze tot expressie komen), en tussenliggende sequenties genaamd introns (intron geeft hun tussenliggende rol aan), die betrokken kunnen zijn bij de genregulatie, maar tijdens de verwerking uit het pre-mRNA worden verwijderd. De opeenvolgingen van Intron in mRNA coderen geen functionele proteã nen.
de ontdekking van introns kwam als een verrassing voor onderzoekers in de jaren zeventig die verwachtten dat pre-mRNAs eiwitsequenties zou specificeren zonder verdere verwerking, zoals ze hadden waargenomen in prokaryoten. De genen van hogere eukaryoten bevatten vaak één of meer introns. Deze gebieden kunnen aan regelgevende opeenvolgingen corresponderen; nochtans, is de biologische betekenis van het hebben van vele introns of het hebben van zeer lange introns in een gen onduidelijk. Het is mogelijk dat introns genuitdrukking vertragen omdat het langer duurt om pre-mRNAs met veel introns te transcriberen. Als alternatief kunnen introns niet-functionele opeenvolgingsresten zijn die overblijven van de fusie van oude genen door evolutie. Dit wordt gesteund door het feit dat afzonderlijke exons vaak afzonderlijke eiwitsubeenheden of domeinen coderen. Voor het grootste deel, kunnen de opeenvolgingen van introns worden gemuteerd zonder uiteindelijk het eiwitproduct te beà nvloeden.
alle introns van een pre-mRNA moeten volledig en nauwkeurig worden verwijderd vóór de eiwitsynthese. Als het proces door zelfs één enkel nucleotide dwaalt, zou het lezingskader van de opnieuw aangesloten exons verschuiven, en de resulterende proteã ne dysfunctioneel zou zijn. Het proces van het verwijderen van introns en het opnieuw verbinden van exons wordt splicing genoemd (figuur 1). Introns worden verwijderd en gedegradeerd terwijl pre-mRNA nog in de kern is. Het verbinden komt door een opeenvolging-specifiek mechanisme voor dat introns verzekert zullen worden verwijderd en exons met de nauwkeurigheid en de precisie van één enkel nucleotide weer verenigd. Het verbinden van pre-mRNAs wordt geleid door complexen van proteã nen en de molecules van RNA genoemd spliceosomes.
praktijkvraag
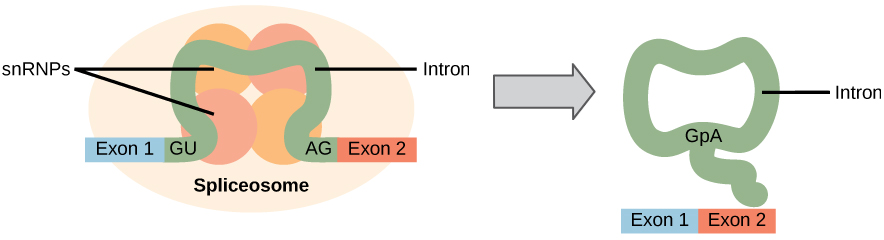
figuur 1. Het Pre-mRNA verbinden impliceert de nauwkeurige verwijdering van introns van het primaire transcript van RNA. Het verbindingsproces wordt gekatalyseerd door eiwitcomplexen genoemd spliceosomes die uit proteã nen en de molecules van RNA genoemd snRNAs worden samengesteld. Spliceosomes herkennen opeenvolgingen bij het einde van 5′ en 3′ van intron.
fouten in splicing zijn betrokken bij kanker en andere ziekten bij de mens. Welke soorten mutaties kunnen leiden tot splitsingsfouten?
merk op dat meer dan 70 individuele introns aanwezig kunnen zijn, en elk het proces moet ondergaan van het verbinden—naast 5′ het afdekken en de toevoeging van een poly-een staart—enkel om één enkel, vertaalbaar mRNA molecuul te produceren.
RNA-bewerking in trypanosomen
Figuur 2. Trypanosoma brucei is de veroorzaker van slaapziekte bij mensen. MRNAs van deze ziekteverwekker moet door de toevoeging van nucleotiden worden gewijzigd alvorens eiwitsynthese kan voorkomen. (credit: wijziging van het werk van Torsten Ochsenreiter)
de trypanosomen zijn een groep protozoa, waaronder het pathogeen Trypanosoma brucei, dat bij mensen slaapziekte veroorzaakt (Figuur 2). Trypanosomen, en vrijwel alle andere eukaryoten, hebben organellen genoemd mitochondriën die de cel van chemische energie voorzien. Mitochondria zijn organellen die hun eigen DNA uitdrukken en om de overblijfselen van een symbiotische relatie tussen een eukaryote en een overspoelde prokaryote worden verondersteld te zijn. Het mitochondriale DNA van trypanosomen vertoont een interessante uitzondering op het centrale Dogma: hun pre-mRNAs hebben niet de juiste informatie om een functioneel eiwit te specificeren. Gewoonlijk, is dit omdat mRNA verscheidene u-nucleotiden mist. De cel voert een extra stap van de verwerking van RNA genoemd het uitgeven van RNA uit om dit te verhelpen.
andere genen in het mitochondriale genoom coderen 40-tot 80-nucleotide guide RNAs. Een of meer van deze molecules interageert door het complementaire base in paren rangschikken met sommige nucleotiden in het pre-mRNA transcript. Nochtans, heeft gidsrna meer nucleotiden A dan pre-mRNA u nucleotiden heeft om mee te binden. In deze gebieden, de lijnen van gidsrna uit. De 3 ‘ einden van gids RNAs hebben een lange Poly-u-staart, en deze U-basissen worden ingevoegd in gebieden van het pre-mRNA-transcript waarbij de gids RNAs van een lus worden voorzien. Dit proces wordt volledig bemiddeld door de molecules van RNA. Dat wil zeggen, dient De Gids RNAs-eerder dan proteã nen-als katalysatoren in het uitgeven van RNA.RNA-bewerking is niet alleen een fenomeen van trypanosomen. In de mitochondriën van sommige planten worden bijna alle pre-mRNA ‘ s bewerkt. Het uitgeven van RNA is ook geà dentificeerd in zoogdieren zoals ratten, konijnen, en zelfs mensen. Wat zou de evolutionaire reden voor deze extra stap in pre-mRNA verwerking kunnen zijn? Een mogelijkheid is dat de mitochondriën, die resten van oude prokaryotes zijn, een eveneens Oude op RNA-gebaseerde methode hebben om genuitdrukking te regelen. Ter ondersteuning van deze hypothese, verschillen de bewerkingen aan pre-mRNAs afhankelijk van cellulaire voorwaarden. Hoewel speculatief, kan het proces van het uitgeven van RNA een holdover van een primordiale tijd zijn wanneer de molecules van RNA, in plaats van proteã nen, van het katalyseren reacties de oorzaak waren.
Probeer Het.
Draag Bij!
verbeter deze pagina meer