Creative Proteomics Blog
de sequentie van aminozuren in een eiwit of peptide kan worden geïdentificeerd door Edman degradation, die werd ontwikkeld door Pehr Edman. Deze methode kan peptide van n-terminal etiketteren en splitsen zonder de peptide banden tussen andere aminozuurresiduen te verstoren. De Edman degradatie reactie werd geautomatiseerd in 1967 door Edman en Beggs. Tegenwoordig, wordt de geautomatiseerde Edman degradatie (de eiwitsequenator) wijd gebruikt, en het kan peptides tot 50 aminozuren rangschikken.
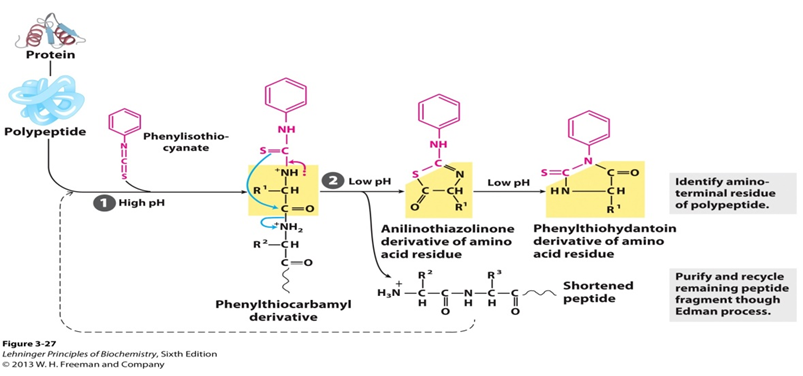
cyclische afbraak van peptiden op basis van de reactie van fenylisothiocyanaat met de vrije aminogroep van het n-eindresidu, zodat aminozuren één voor één worden verwijderd en als hun fenylthiohydantoïnederivaten worden geïdentificeerd. Sprekend aan het specifieke proces, wordt een uncharged peptide met phenylisothiocyanate (PITC) bij het aminoeind onder mild alkalische voorwaarden gereageerd om een phenylthiocarbamoyl derivaat (PTC-peptide) te geven. Dan, onder zure omstandigheden, valt de thiocarbonyl zwavel van het derivaat de carbonylkoolstof van het n-terminale aminozuur aan. Het eerste aminozuur wordt gespleten als anilinothiazolinone derivaat (ATZ-aminozuur) en de rest van peptide kan worden geà soleerd en aan de volgende degradatiecyclus worden onderworpen. Eenmaal gevormd, is dit thiazolonderivaat stabieler dan fenylthiocarbamylderivaat. Het ATZ-aminozuur wordt dan verwijderd door extractie met ethylaceraat en omgezet in een fenylthiohydantoïnederivaat (PTH-aminozuur). En de chromatografie kan worden gebruikt om het PTH-residu te identificeren dat door elke cyclus wordt geproduceerd.
wat de geautomatiseerde Edman-degradatie betreft, kunnen eiwitten worden geanalyseerd door ze in oplossing toe te passen op een TFA-filter en vervolgens te laden op het Edman-sequentieinstrument. De proteã nen in mengsels worden eerst gescheiden door 1D of 2D gels en dan gevlekt op een PVDF-membraan. De proteã nen worden ontdekt door coomassie blauw, Amido zwart of Poncau s bevlekken en de proteã nen van belang uitgesneden en het PVDF membraanstuk geladen op Edman sequencer.Bij de ontwikkeling van massaspectrometrie begon het gebruik van Edman-degradatiesequencing af te nemen. Nochtans, blijft het de methodes voor verscheidene types van eiwit structurele analysetoepassingen. Het kan worden gebruikt om de N-terminale grens van recombinante eiwitten te verifiëren of om de N-terminale grens van protease-resistente domeinen te bepalen, met name wanneer het eiwit of domein >40 tot 80 kDa is of niet gemakkelijk kan worden gezuiverd. Het kan ook worden gebruikt om de nieuwe n-eind en proteolytic splitsingsplaats in de eiwitfragmenten te identificeren. Bovendien, wat betreft sommige nieuwe proteã nen en peptides waar opeenvolgingsdatabases niet beschikbaar zijn voor het zoeken van het gegevensbestand van lidstaten/lidstaten, kan de degradatie van Edman voor analyse worden gebruikt.
het n-terminale aminozuur van het eiwit kan worden afgesplitst. Aldus, in het proces, identificeert de eerste cyclus zo het nauwkeurige n-eind aminozuur. Bovendien, omdat de vrijgegeven aminozuren door chromatografie worden geà dentificeerd en gekwantificeerd, kunnen de aminozuren met identiek molecuulgewicht worden geà dentificeerd. Bijvoorbeeld, hebben isoleucine en leucine een massa van 113 Da, maar zij hebben verschillende retentietijd. Bovendien kan Edman het rangschikken op PVDF vlekken van 1D en 2D gels worden uitgevoerd, die n-het eind rangschikken van proteã nen in het mengsel toelaat. Nochtans, zal Edman degradatie het rangschikken niet beschikbaar zijn wanneer peptide waarvan n-eindpunt chemisch, zoals acetylation is gewijzigd. En omdat de PITC niet kan reageren met niet-α-aminozuur, zal het sequencen stoppen als een niet-α-aminozuur wordt aangetroffen zoals isoaspartic zuur. Bovendien kunnen de grotere proteã nen niet door het Edman rangschikken worden gerangschikt.
bij Creative Proteomics biedt ons professionele eiwitsequentieplatform N-terminale sequentieanalyse door Edman-degradatie of massaspectrometrie (MS), met elkaar aanvullende voordelen. Ons team van experts met uitgebreide ervaring kan u helpen begrijpen wat u probeert te onderzoeken en aan uw eisen te voldoen. Bovendien kunnen wij andere eiwitidentificatiediensten leveren, met inbegrip van:
- peptide mass fingerprinting (PMF) service
- Sequence analysis of peptiden or proteins
- de novo protein sequence analysis service
- Shotgun protein identification service
- membraan proteomics service