Frontiers in Microbiology
Introduction
de familie Astroviridae bevat non-enveloped, positive-sense, single-stranded RNA virussen binnen twee geslachten, Mamastrovirus en Avastrovirus, die respectievelijk zoogdieren en vogels infecteren. Momenteel erkent het International Committee on Taxonomy of Viruses (International Committee on Taxonomy of Viruses , 2018) 19 soorten, namelijk Mamastrovirus-1 tot en met -19, binnen het Mamastrovirus genus; er zijn echter tal van stammen in afwachting van classificatie, waarvan sommige voorlopig als nieuwe soorten worden beschouwd (Donato and Vijaykrishna, 2017).Sinds 2010 zijn verschillende astrovirussen in toenemende mate erkend als neuro-invasief bij verschillende zoogdiersoorten, waaronder de mens (Quan et al., 2010; Naccache et al., 2015), mink (Blomström et al., 2010), vee (Li et al., 2013), schapen (Pfaff et al., 2017), en varkens (Boros et al., 2017). Na eerste erkenning van boviene astrovirus-geassocieerde encefalitis bij runderen in de Verenigde Staten (Li et al., 2013), een retrospectieve studie in gevallen van sporadische boviene encefalitis van Onbepaalde etiologie uit Zwitserland bleek dat dit neuro-invasieve astrovirus onopgemerkt was gebleven voor decennia (Selimovic-Hamza et al., 2016). Hoewel de epidemiologie en de transmissieroutes van deze astrovirussen onbekend zijn, is er een suggestie gedaan voor cross-species transmissie op basis van het hoge niveau van identiteit (>98%), gedeeld tussen neuro-invasieve astrovirussen van runderen en schapen op nucleotide-en aminozuurniveaus (Boujon et al., 2017).Boviene astrovirussen (BoAstVs), genaamd BoAstV-NeuroS1 (Li et al., 2013) en BoAstV-CH13 (Bouzalas et al., 2014), werden aanvankelijk gevonden in de hersenen van runderen met niet-suppurative encefalitis in de Verenigde Staten en Zwitserland, respectievelijk. Ondanks de verschillende nomenclatuur, vertegenwoordigen beide virussen dezelfde genotype species (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a) die nog in afwachting is van de officiële classificatie door de ICTV. In 2015 werd een voorheen onbekende boastv-stam, genaamd BoAstV-CH15, geïdentificeerd in de hersenen van koeien met encefalitis in Zwitserland. Volledige genoom fylogenetische vergelijking bleek een nauwere relatie van BoAstV-CH15 met een schaap astrovirus (OvAstV) dan met BoAstV-CH13 (Seuberlich et al., 2016). Co-infectie met BoAstV-CH13 en BoAstV-CH15 werd ook gedocumenteerd in een geval (Seuberlich et al., 2016). Hetzelfde jaar in Duitsland, Schlottau et al. (2016) meldde een nieuw astrovirus, namelijk BoAstV-BH89/14, bij een koe met encefalitis, dat het meest verwant was aan OvAstV en BoAstV-CH15. Vervolgens werd BoAstV-CH13 / NeuroS1 in 2017 geïdentificeerd in gevallen van runderencefalitis in Oost-en West-Canada (Spinato et al., 2017; Selimovic-Hamza et al., 2017b). In 2018, werd een nieuwe neuro-invasieve BoAstV nauw verwant met Noord-Amerikaanse en Europese BoAstV-NeuroS1/BoAstV-CH13, geïdentificeerd in een steer met niet-suppuratieve encefalomyelitis in Japan ,en het voorkomen van intra-genotypische recombinatie tussen de Noord-Amerikaanse en Europese stammen werd gesuggereerd (Hirashima et al., 2018).
hoewel gevallen van astrovirus-geassocieerde encefalitis zijn gemeld in Noord-Amerika, Europa en Azië, is hun aanwezigheid nooit gedocumenteerd op het zuidelijk halfrond. Hier beschrijven we een geval van astrovirus-geassocieerde encefalitis bij runderen in Uruguay, die de geografische verspreiding en genetische diversiteit van neuro-invasieve astrovirussen verbreedt en fylogeografisch bewijs levert dat suggereert dat dit virus vanuit Europa in Amerika werd geïntroduceerd en later naar Azië werd verspreid.
materialen en methoden
geschiedenis en signalering
in juni 2018 ontwikkelde een 22 maanden oude Holsteinse ossen in een groep van 37 ossen in een ∼300 hectare groot bedrijf in Colonia, Uruguay, progressieve neurologische zang, waaronder ongewoon gedrag, doelloos lopen, cirkelen, ataxie, repetitieve en ongecoördineerde tongbewegingen en liggend. De kudde graasde op een jaarlijkse haverweide en werd aangevuld met kuilvoer. Een vermoedelijke klinische diagnose van cerebrale listeriose door de dierenarts leidde tot behandeling met penicilline en streptomycine, maar het dier stierf spontaan na een klinisch verloop van 3 dagen.
pathologisch onderzoek, in situ hybridisatie (ISH) en immunohistochemie (IHC)
de kop van de OS werd van het karkas verwijderd en voor diagnostisch onderzoek aan het Inia ‘ s Veterinary Diagnostic Laboratory (Animal Health Platform) voorgelegd. De helft van de hersenen, een korte segment van de proximale cervicale ruggenmerg (C1), trigeminale ganglion en de wortel van het trigeminale zenuw, de speekselklieren, retropharyngeal lymfeklier, oropharynx, slokdarm -, tong-en skeletspieren, waren onderdompeling-opgelost in 10% neutraal gebufferde formaline gedurende 48-72 h. De weefsels werden routinematig verwerkt in de histologie, ingebed in paraffine, een microtoom-delig op 4-5 µm en gekleurd met hematoxyline en eosine (H&E) en Gram vlekken.
chromogene ISH werd handmatig uitgevoerd op 5 µm secties van formaline-fixed, paraffine-embedded (FFPE) hersenstam, cerebrum en cerebellum op Superfrost Plus-dia ‘ s (Thermo Fisher Scientific, Pittsburgh, PA, Verenigde Staten) met behulp van de Rnascope 2.5 Red assay kit (Cat #322360, Advanced Cell Diagnostics, Hayward, CA, Verenigde Staten) en de BoAstV probe Cat. #406921. De sonde bestaat uit 20ZZ paren gericht gebied 5232-6180 van het virus (GenBank KF233994. 1). Elke 5 µm sectie van weefsel werd voorbehandeld met warmte en protease voorafgaand aan probe hybridisatie gedurende 2 uur bij 40°C en verwerkt volgens de aanbevelingen van de fabrikant. De negatieve controles die voor de validatie van het signaal werden gebruikt, omvatten een niet-gerelateerde (GC-inhoud matched) sonde uitgevoerd op seriële secties en het onderzoeken van weefsel van niet-geïnfecteerde dieren. De dia ‘ s werden met hematoxyline tegengewerkt en gemonteerd met EcoMount (Biocare Medical, Concord, CA, Verenigde Staten).
Daarnaast werd IHC uitgevoerd in FFPE-secties van hersenstam, cerebrum en cerebellum, zoals eerder beschreven, voor de identificatie van het West-Nijlvirus (WNV, Flavivirus) (Palmieri et al., 2011), rabiësvirus (Lyssavirus) (Stein et al., 2010), en Chlamydia spp. (Giannitti et al., 2016) antigenen.
moleculaire virologie
Nucleïnezuurextractie werd uitgevoerd uit een gepoolde steekproef van bevroren (-20°C) hersenen met behulp van MagMAX Nucleïnezuurisolatie Kit® (Thermo Fisher Scientific). Voor astrovirus detectie werd reverse transcriptie (RT) uitgevoerd met RevertAid Reverse Transcriptase® (Thermo Fisher Scientific) en random hexamer primers (Qiagen). PCR werd uitgevoerd van cDNA met MangoMix® (Bioline) en primers die een 432-nucleotide fragment van het astrovirus polymerase gen (Tse et al., 2011). Het PCR-product werd gevisualiseerd in 2% agarose gel, gezuiverd met PureLink ® Quick Gel Extraction en PCR Purification Combo Kit (Invitrogen), en gesequenced bij Macrogen Inc. (Seoul, Zuid-Korea). Voor astrovirus whole genome amplification, Maxima H Minus Reverse Transcriptase(Thermo Fisher Scientific) en oligo (dT)18 voor het verkrijgen van cDNA, en MangoMix® (Bioline) of Ranger DNA Polymerase (Bioline) met primers beschreven door Hirashima et al., 2018, werden gebruikt. De PCR-producten werden gevisualiseerd in 1-2% agarose gel, gezuiverd en gerangschikt zoals hierboven vermeld. Sequence assembly werd uitgevoerd met SeqMan (Lasergene 8, DNASTAR). Zesentwintig complete genoomsequenties van neuro-invasieve astrovirus van runderen, schapen, varkens, mensen en nertsen, en enterische boviene astrovirus beschikbaar in GenBank werden gedownload en uitgelijnd met behulp van Clustal W in MEGA 7 software (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) werd gebruikt om het best passende model van sequence evolution (SYM+I+G4) te bepalen en om een phylogenetische boom met de bijna volledige sequenties van de boastv gedetecteerd in dit geval te construeren, en die volledige sequenties gedownload van GenBank, met behulp van bootstrap als statistische methode om clades robuustheid te beoordelen. Gelijkenis plot werd uitgevoerd met SimPlot software (Lole et al., 1999). P-afstanden op aminozuurniveau van de ORF2 werden geschat met MEGA 7 software (Kumar et al., 2016).
Daarnaast werd een Bayesiaanse fylogeografische analyse uitgevoerd met het BEAST v1. 8. 4 pakket (Drummond et al., 2012), met behulp van: het volledige codeergebied van BoAstV CH13 / NeuroS1 lineage, ORF1ab (non-structural genes), ORF2 (structural gen), ORF1a (protease) en een partiële ORF1b (polymerase genomic region, waarvoor Canadese stammen beschikbaar waren), met alle sequenties beschikbaar in GenBank (laatste toetreding 18 April 2019), om de evolutionaire snelheid, de leeftijden/jaren van de gemeenschappelijke voorouders te bepalen, en de meest waarschijnlijke route van virale circulatie per land (Zwitserland, Uruguay, Verenigde Staten, Canada en Japan). Het gebrek aan recombinatie in de dataset werd bepaald gebruikend programma van de Recombinatiedetectie 4. Het substitutiemodel dat het best past bij elke uitlijning werd bepaald met behulp van MEGA 7 software door middel van Bayesian information criterion (Bic) waarden, en de temporele structuur van elke dataset werd geëvalueerd met behulp van TempEst (Rambaut et al., 2016). De lognormale ontspannen moleculaire klok met Bayesiaanse Skyline analyse werd geselecteerd door Bayes Factor uit de verschillende combinaties van moleculaire klokken en coalescente boom priors gebruikt. Het land van opsporing werd gebruikt als kenmerk. De Markov keten Monte Carlo lengte was 100 miljoen generaties, zorgen voor de convergentie van de analyse, geëvalueerd in Tracer v1.6.0, en de posterieure waarschijnlijkheid werd gebruikt om clades te evalueren. De maximale clade geloofwaardigheid boom (MCCT) werd verkregen met behulp van TreeAnnotator software van BEAST en gevisualiseerd in FigTree v1.4.3.Ten slotte werd DNA geëxtraheerd uit bevroren hersenen verwerkt door PCR voor de detectie van runderherpesvirussen 1 en 5 (BHV-1 en -5), zoals eerder beschreven (Ashbaugh et al., 1997).
Bacteriologie
verse monsters van cerebrum en hersenstam werden routinematig verwerkt voor aërobe bacterieculturen in bloed en MacConkey-agars, en selectieve kweek voor Listeria monocytogenes (Al-Zoreky en Sandine, 1990).
resultaten en discussie
de klinische symptomen en epidemiologische bevindingen in het hier beschreven geval, hoewel niet-specifiek, waren vergelijkbaar met die beschreven in andere gevallen van boviene astrovirus-geassocieerde encefalitis, die gewoonlijk als sporadisch wordt beschreven (Selimovic-Hamza et al., 2016), met een verscheidenheid aan neurologische tekorten (Deiss et al., 2017), met een duur van klinische symptomen die meestal varieert van 1 dag tot 3 weken (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
macroscopisch onderzoek van de hersenen, het C1-segment van het ruggenmerg en andere weefsels van het hoofd toonde geen significante Grove anatomische laesies aan. Histologisch was er sprake van matige tot ernstige lymfocytische, histiocytische en plasmacytische meningoencephalomyelitis die het telencephalon (inclusief de cerebrale hemisfeer en hippocampus), de hersenstam en het enige onderzochte segment van het ruggenmerg aantastte. Laesies werden voornamelijk gedistribueerd in de grijze stof en de beperkende gebieden van de witte stof. In de getroffen gebieden was er perivasculaire cuffing en lymfoplasmacytaire en histiocytische ontsteking en neuronale necrose / neuronofagie met gliose in de aangrenzende neuropil. Er was satellitosis van aangetaste, necrotische neuronen (figuren 1A–D). De laesies waren veel minder frequent en ernstig in het cerebellaire parenchym, hoewel er multifocale matige cerebellaire leptomeningitis was. Er werden geen intralesionale bacteriën gevonden met H&E en Gramvlekken. Er werden geen significante histologische veranderingen gevonden in de andere onderzochte weefsels.

figuur 1. Histologische laesies in de hersenstam (A, B) en cerebrale cortex (C,D) en detectie van BoAstV RNA in de cerebrale cortex (E,G). Beelden (A-D) zijn delen van de hersenen gekleurd met H&E; beelden E en G zijn secties van cerebrale cortex die hybridisatie demonstreren gebruikend chromogene ISH gebruikend de specifieke sonde van BoAstV, die met hematoxylin wordt tegenbehandeld; beeld F is een seriële sectie van de sonde van dapb van de cerebrale cortex (negatieve controle), die met hematoxylin wordt tegenbehandeld. (A) een perivenulaire ruimte wordt duidelijk uitgebreid door ontstekingscellen (meestal lymfocyten en histiocyten) die ook de aangrenzende neuropil infiltreren. (B) het neuron in het centrum heeft hypereosinofiele perikaryon en karyorrhexis (necrose) en het neuronale lichaam is omgeven door verhoogde aantallen gliale (satellitosis) en ontstekingscellen. C) de leptomeninge wordt geïnfiltreerd door lymfocyten en histiocyten. (D) een gebied van de hersenschors met meerdere hypereosinofiele (necrotische) neuronen en een groot vat met perivasculaire lymfatische manchetten. (E) in een seriële sectie van cerebrale cortex, wordt de overvloedige intracytoplasmische etikettering van BoAstV RNA afgebeeld door sterke, korrelig rode chromogeenafzetting binnen neuronaal cytoplasma van het soma en neuronale uitbreidingen (E,G), die niet met hybridisatie met behulp van de negatieve controlesonde (F) aanwezig is.
een neuro-invasieve virale infectie werd vermoed na histologisch onderzoek van het centrale zenuwstelsel. Runderen met encefalitis zijn zorgwekkend omdat veel neuropathogenen bij herkauwers zoönotisch zijn (Cantile and Youssef, 2016); zo zou een diagnose van encefalitis uitgebreide laboratoriumtests moeten veroorzaken om waar mogelijk op infectieuze agentia te screenen. In het hierin beschreven geval, ihcs voor WNV, rabiësvirus en Chlamydia spp. en PCR voor BHV-1 en -5 waren allemaal negatief, en er werden geen pathogene bacteriën gekweekt uit hersenweefsel. Omdat de OS < 2 jaar oud was en er geen spongiforme veranderingen in de hersenstam werden waargenomen, werd het dier niet getest op boviene spongiforme encefalopathie (BSE), een exotische ziekte bij volwassen runderen die nooit in Uruguay is gemeld. Bovendien is BSE niet inflammatoir (Cantile and Youssef, 2016).
in situ hybridisatie werd uitgevoerd met behulp van een sonde gegenereerd uit BoAstV-NeuroS1, en er was hybridisatie van de sonde overvloedig aanwezig in, en beperkt tot het cytoplasma van neuronen in de cerebrale hemisfeer en hippocampus (figuur 1E–G). In deze gebieden colokaliseerde de probe hybridisatie met necrotische neuronen en gebieden van gliosis, zonder probe hybridisatie die in de gliacellen, of ontstekingscellen van de perivasculaire manchetten Kan worden ontdekt. Er werd geen viraal nucleïnezuur gedetecteerd door ISH in het cerebellum, dat slechts minimale ontstekingslaesies in het parenchym had, maar matige leptomeningitis, of in de hersenstam, inclusief secties met ernstige ontsteking. Dit betekent dat, topografisch, de detectie van virale distributie door ISH beperkter was dan encefalitis in de onderzochte secties, die af en toe is beschreven in gevallen van boastv-CH13/NeuroS1-geassocieerde encefalitis bij runderen (Selimovic-Hamza et al., 2017a, b). Een reden voor dit incidentele gebrek aan virale RNA detectie in lesioned gebieden van de hersenen zou de detectielimiet van de ISH, of de klaring van het virus in ontstoken gebieden van de hersenen tegen het tijdstip van overlijden, zoals eerder gesuggereerd (Selimovic-Hamza et al., 2017b). Zoals verwacht, werd geen probe hybridisatie ontdekt door ISH in het hersenenweefsel dat als negatieve controle wordt gebruikt.
Astrovirus werd gedetecteerd in de hersenen door RT-PCR. De bijna volledige analyse van de genoomopeenvolging onthulde een mamastrovirus stam binnen de ch13/NeuroS1 clade, noemden wij BoAstV-Neuro-Uy, werd de opeenvolging in GenBank onder toetredingsnummer MK386569 gedeponeerd. De fylogenetische analyse toonde nabijheid aan met andere neuro-invasieve astrovirussen binnen de Virginia/Human-Nertsen-Ovine (VA / HMO) clade (Figuur 2), die de meest bekende neuro-invasieve astrovirussen bevat (Hirashima et al., 2018; Reuter et al., 2018). De bijna volledige opeenvolging van BoAstV-Neuro-Uy is 6427 bp in lengte en heeft een opeenvolging identiteit van 94% met kagoshimasr28-462 stam. BoAstV-Neuro-Uy heeft gelijkaardige eigenschappen als andere spanningen van lijn CH13 / NeuroS1: een 5 ‘ UTR gebied van 51 nt, ORF1a (protease) van 861 aminozuren (aa), ORF1b van 523 aa (RNA-afhankelijke polymerase van RNA), en ORF2 van 758 aa (capsid proteã ne). Helaas kon de 3 ‘ UTR niet worden gesequenced, maar een poly(A) staart wordt verondersteld aanwezig te zijn omdat oligo(dT)18 werd gebruikt om cDNA te verkrijgen. Daarnaast is de heptamerische aaaaaaac-sequentie, een ribosomaal frameshiftsignaal, aanwezig. P-afstanden op het aminozuurniveau van de ORF2 bevestigden de toewijzing van deze stam aan de Ch13/NeuroS1-klasse. P-afstanden < 0.35 tussen BoAstV-Neuro-Uy en andere leden van deze clade (Tabel 1) zou een classificatie van deze virale stammen binnen één zelfde soort ondersteunen; Mamastrovirus-13 is onlangs voorgesteld door andere auteurs (Donato and Vijaykrishna, 2017; Hirashima et al., 2018), hoewel definitieve soortenaanwijzing door de ICTV in behandeling is. De sonde die voor ISH wordt gebruikt, die van BoAstV-NeuroS1 wordt geproduceerd, had 92.7% opeenvolgingsidentiteit met BoAstV-Neuro-Uy.
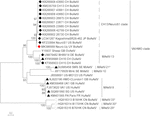
Figuur 2. Fylogenetische analyse van nucleotideopeenvolgingen van de volledige lengte door middel van de methode van de maximumwaarschijnlijkheid. BoAstV-Neuro-Uy is gemarkeerd met een rode ruit. Andere sequenties van boviene neuro-invasieve astrovirussen zijn gemarkeerd met zwarte rhombi, sequenties van niet-boviene neuro-invasieve astrovirussen zijn gemarkeerd met zwarte driehoeken, en sequenties van enterische boviene astrovirussen zijn gemarkeerd met witte cirkels. Mamastrovirus soorten zijn afgebeeld; sterretjes geven soorten aan die nog door de ICTV herkend moeten worden. Stammen binnen de Ch13 / NeuroS1-klasse die nog niet door de Ictv aan een soort zijn toegewezen, worden geïdentificeerd met een stippellijn. Bootstrap waarden worden aangegeven in knooppunten. CH, Zwitserland; JP, Japan; US, Verenigde Staten Van Amerika; UY, Uruguay; GB, Verenigd Koninkrijk van Groot-Brittannië; de, Duitsland; SE, Zweden; FR, Frankrijk; CN, China. BoAstV, boviene astrovirus; OvAstV, schapen astrovirus; PoAstV, porcine astrovirus; HuAstV, humaan astrovirus; MiAstV, nerts astrovirus.
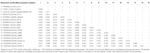
Tabel 1. Schattingen van evolutionaire divergentie op het aminozuurniveau van het volledige ORF2-gebied tussen sequenties van mamastrovirus-13 van runderen en schapen en stammen binnen de Ch13/NeuroS1-klasse die nog niet aan een soort zijn toegewezen door het Internationaal Comité voor taxonomie van virussen (Ictv).
Studies gebaseerd op neuropathologische onderzoeken en astrovirus nucleïnezuur en eiwit detectie hebben geconcludeerd dat er een waarschijnlijk causaal verband bestaat tussen astrovirus infectie en neurologische ziekte en laesies bij runderen (Selimovic-Hamza et al., 2017a; Reuter et al., 2018). Voor zover wij weten is astrovirus-geassocieerde encefalitis nog niet experimenteel gereproduceerd. Dit vereist isolatie van neuro-invasieve astrovirussen uit klinische gevallen, wat in ons geval niet is geprobeerd.
de bron van BoAstV-Neuro-Uy kon in dit geval niet worden vastgesteld. Echter, reservoir vee en wilde dieren moeten worden overwogen, omdat het vee werd gefokt onder uitgebreide buitenomstandigheden. Het aangetaste dier was in februari 2018 gekocht en samen met andere 9 ossen naar de boerderij verhuisd. Helaas weigerde de eigenaar verdere bemonstering en testen van andere dieren in het pand, en een gedetailleerder epidemiologisch onderzoek. Geen van de andere dieren in de groep had neurologische ziekte ontwikkeld vanaf augustus 2018, de laatste keer dat contact werd opgenomen met de dierenarts. Een seizoensgebondenheid van het begin van de winter tot het einde van de lente is gesuggereerd voor gevallen van astrovirus-geassocieerde encefalitis in Zwitserland (Selimovic-Hamza et al., 2016). Interessant is dat het hier beschreven geval plaatsvond in juni, wat overeenkomt met de herfst-winter overgangsperiode op het zuidelijk halfrond.
terwijl neurotrope astrovirussen zijn geïdentificeerd in Noord-Amerika (Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) en Azië (Hirashima et al., 2018), is hun aanwezigheid nooit gemeld op het zuidelijk halfrond, dus deze mededeling verbreedt de geografische verspreiding van astrovirus-geassocieerde encefalitis. Om te beoordelen of de virusstam die in Uruguay werd gedetecteerd mogelijk afkomstig is uit Europa, Noord-Amerika of Azië, hebben we de evolutionaire snelheid geschat en een fylogeografische analyse uitgevoerd met behulp van neuro-invasieve BoAstV-sequenties die beschikbaar zijn in GenBank. De evolutionaire snelheid geschat met behulp van de volledige codering gebied was 4.27 × 10-4 (95% hoogste kansdichtheid-HPD-, 2,19–6,46 × 10-4) nucleotide substituties/plaats/jaar, die wordt verwacht voor een RNA-virus (Jenkins et al., 2002), maar lager dan geschat voor enterische menselijke astrovirussen (Babkin et al., 2012, 2014). Het ORF1ab-gebied vertoonde een vergelijkbaar evolutionair percentage (4,20 × 10-4, 95% HPD 1,66–6,46 × 10-4 substituties/locatie/jaar) als het volledige coderingsgebied, terwijl de ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6.46 × 10-4 substituties/locatie/jaar) en ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 substituties / plaats/jaar) vertoonde een iets snellere evolutionaire snelheid, en de partiële polymerase genomische regio (ORF1b) vertoonde een iets langzamere evolutionaire snelheid (5,39 × 10-4, 95% HPD 6,41 × 10-7-1, 10 × 10-3 substituties/plaats/jaar).
zoals bepaald door de fylogeografische analyse met het volledige coderingsgebied, en weergegeven in de MCTT (Figuur 3), zijn er twee sublijnen (CH13 en NeuroS1) gebaseerd op referentiestammen, die een gemeenschappelijke voorouder hebben. De meest recente gemeenschappelijke voorouder van deze sublijnen (lijn CH13/NeuroS1) ontstond in Europa ongeveer in 1885 (95% HPD, 1794-1940). Aan het begin van de 1900 ‘ s, de twee sub-lineages diverged, de ch13 sub-lineage bleef circuleren in Europa, terwijl de NeuroS1 sub-lineage verspreid naar Amerika en Azië. Het meest waarschijnlijke scenario is dat de NeuroS1 sub-lineage werd geïntroduceerd in Uruguay uit Europa rond het jaar 1921 (95% HPD, 1849-1967), vermoedelijk door middel van veehandel, vervolgens verspreid naar Noord-Amerika, en later naar Japan (Figuur 3). Wegens de beperking in het aantal beschikbare opeenvolgingen in GenBank, die de analyse zouden kunnen hebben bevooroordeeld, werden de verkregen resultaten gebruikend het volledige codeergebied vergeleken met die verkregen met andere genomic gebieden (ORF1ab, ORF2, ORF1a, en ORF1b) beschikbaar voor een groter aantal spanningen (d.w.z., Canadese spanningen). In alle analyses is het meest waarschijnlijke scenario dat de insleep van het virus in Uruguay vanuit Europa plaatsvond (aanvullende cijfers S1A–D). Bovendien was de geschatte datum voor deze introductie, verkregen met het orf1ab en partieel polymerasegenoomgebied (Orf1b) (aanvullende cijfers S1A,D), vergelijkbaar met die verkregen met het volledige coderingsgebied, terwijl de geschatte datum van introductie verkregen met ORF2 en ORF1a eerder was,maar met een breder 95% HPD-interval (aanvullende cijfers S1B, C). Een introductie van de sub-lineage NeuroS1 rechtstreeks naar Canada vanuit Europa, met latere verspreiding naar de Verenigde Staten en Japan, is ook plausibel, s getoond in aanvullend cijfer S1D.

Figuur 3. Maximale klade geloofwaardigheid boom verkregen door analyse van de volledige codering regio. De kleur van de takken vertegenwoordigt het meest waarschijnlijke land waar de voorouders circuleerden, posterieure waarschijnlijkheidswaarden worden getoond in de takken en de getallen in elke knoop vertegenwoordigen de jaren van oorsprong voor elke clade met het 95% HPD interval. Sublijnen worden aangegeven met labels.
nader onderzoek is nodig om de geografische verspreiding, pathogene mechanismen (met name mechanismen van transmissie en binnenkomst), Moleculaire Epidemiologie en potentiële interspecies-overdracht van neuro-invasieve astrovirussen te beoordelen.
beschikbaarheid van gegevens
de voor deze studie gegenereerde datasets zijn te vinden in GenBank, MK386569.
bijdragen van auteurs
FG, RDC en MC droegen bij aan de opzet van de studie. FG en RDC hebben het pathologische onderzoek en de bemonstering uitgevoerd. PP voerde de kruising in situ uit. FU voerde de immunohistochemie uit. LM, RC en MC voerden moleculaire virologie testen uit. MC voerde de sequentie en fylogeografische analyses en bijbehorende cijfers uit. FG en PP verkregen de histologische beelden. MF voerde de bacteriële culturen uit. FG en MC schreven de eerste versie van het manuscript. RDC, PP, FU, LM, MF en RC schreven delen van het manuscript. Alle auteurs hebben bijgedragen aan de revisie van het manuscript, hebben de ingediende versie gelezen en goedgekeurd.
financiering
dit werk werd gefinancierd door Grants PL-015 N-15156 van INIA en 158 van het “Programa de Iniciación a la Investigación 2017” van “Comisión Sectorial de Investigación Científica” (CSIC). MC en RDC erkennen steun van het “National Agency for Research and Innovation” (ANII) en Inia, respectievelijk, door middel van Ph.D. beurzen. FG erkent steun van ANII via mobiliteitsbeurs MOV_CA_2018_1_150021.
belangenverstrengeling verklaring
de auteurs verklaren dat het onderzoek werd uitgevoerd zonder enige commerciële of financiële relatie die als een potentieel belangenconflict kon worden opgevat.
de recensent TS verklaarde een verleden coauteurschap met een van de auteurs PP aan de handling editor.
Dankbetuigingen
de auteurs danken Yisell Perdomo en Cecilia Monesiglio van INIA, en Karen Sverlow en Juliann Beingesser van CAHFS voor technische bijstand.
aanvullend materiaal
het aanvullende materiaal voor dit artikel is online te vinden op: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
figuur S1 / maximale klade geloofwaardigheid bomen (Mcct ‘ s) verkregen door analyse van de volledige lengte ORF1ab (A), Volledige lengte ORF2 (B), Volledige lengte ORF1a (C), en gedeeltelijke ORF1b (D). De kleur van de takken vertegenwoordigt het meest waarschijnlijke land waar de voorouders circuleerden, posterieure waarschijnlijkheidswaarden worden getoond in de takken, en de getallen in elke knoop vertegenwoordigen de jaren van oorsprong voor elke clade met het 95% HPD-interval. Sublijnen worden aangegeven met labels.
voetnoten
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., and Sandine, W. E. (1990). Zeer selectief medium voor de isolatie van Listeria monocytogenes uit voedsel. Appl. Environ. Microbiol. 56, 3154–3157.
PubMed Abstract / Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chowdhury, S., and Collins, J. K. (1997). Specifieke detectie van uitscheiding en latentie van boviene herpesvirus 1 en 5 met behulp van een geneste polymerasekettingreactie. J. Vet. Diagn. Investeren. 9, 387–394. doi: 10.1177/104063879700900408
PubMed Abstract / CrossRef Full Text / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., en Tikunova, N. V. (2014). Recombinatieanalyse gebaseerd op de volledige genomen van HAstV-2 en HastV-4. Infecteren. Genet. Evol. 22, 94–102. doi: 10.1016 / j. meegid.2014.01.010
PubMed Abstract / CrossRef Full Text / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V., en Tikunova, N. V. (2012). Hoge evolutionaire snelheid van menselijk astrovirus. Infecteren. Genet. Evol. 12, 435–442. doi: 10.1016 / j. meegid.2012.01.019
PubMed Abstract / CrossRef Full Text / Google Scholar
Blomström, A. L., Widén, F., Hammer, A. S., Belák, S., and Berg, M. (2010). Detectie van een nieuw astrovirus in hersenenweefsel van nertsen die lijden aan het schudden van het nertssyndroom door gebruik van virale metagenomics. J. Clin. Microbiol. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
PubMed Abstract / CrossRef Full Text / Google Scholar
Boros, Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et al. (2017). Uitbraken van neuro-invasieve astrovirus geassocieerd met encefalomyelitis, zwakte en verlamming bij gespeende varkens, Hongarije. Emerg. Infecteren. Dis. 23, 1982–1993. doi: 10.3201 / eid2312. 170804
PubMed Abstract / CrossRef Full Text / Google Scholar
Boujon, C. L., Koch, M. C., Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Indicatie van cross-species transmissie van astrovirus geassocieerd met encefalitis bij schapen en runderen. Emerg. Infecteren. Dis. 23, 1604–1608. doi: 10.3201 / eid2309.170168
PubMed Abstract / CrossRef Full Text / Google Scholar
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., en Seuberlich, T. (2016). Volledig genoom gebaseerde moleculaire karakterisatie van encefalitis-geassocieerde boviene astrovirussen. Infecteren. Genet. Evol. 44, 162-168. doi: 10.1016 / J. meegid.06/052/2016
PubMed Abstract / CrossRef Full Text / Google Scholar
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C., Zurbriggen, A., Vandevelde, M., et al. (2014). Neurotropisch astrovirus bij runderen met niet-suppuratieve encefalitis in Europa. J. Clin. Microbiol. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
PubMed Abstract / CrossRef Full Text / Google Scholar
Cantile, C., and Youssef, S. (2016). “Nervous System,” in Jubb, Kennedy, and Palmer ‘ s Pathology of Domestic Animals, 6th Edn, ed. M. G. Maxie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T., and Meylan, M. (2017). Neurologische klinische symptomen bij runderen met astrovirus-geassocieerde encefalitis. J. Vet. Stagiair. Med. 31, 1209–1214. doi: 10.1111 / jvim.14728
PubMed Abstract / CrossRef Full Text / Google Scholar
Donato, C., and Vijaykrishna, D. (2017). Het brede gastheerbereik en de genetische diversiteit van zoogdierastrovirussen en aviaire astrovirussen. Virussen 9: E102. doi: 10.3390 / v9050102
PubMed Abstract / CrossRef Full Text / Google Scholar
Drummond, A. J., Suchard, M. A., Xie, D., and Rambaut, A. (2012). Bayesiaanse fylogenetica met BEAUti en het beest 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev / mss075
PubMed Abstract / CrossRef Full Text / Google Scholar
Giannitti, F., Anderson, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Chlamydia pecorum: foetale en placentale laesies bij sporadische capriene abortus. J. Vet. Diagn. Investeren. 28, 184–189. doi: 10.1177/1040638715625729
PubMed Abstract / CrossRef Full Text / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Gehele genoomanalyse van een nieuw neurotropisch boviene astrovirus gedetecteerd in een Japanse zwarte OS met niet-suppuratieve encefalomyelitis in Japan. Boog. Virol. 163, 2805–2810. doi: 10.1007/s00705-018-3898-3
PubMed Abstract / CrossRef Full Text / Google Scholar
International Committee on Taxonomy of Viruses (2018). Master Species List (MSL32), Versie 1. Beschikbaar op: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (geraadpleegd in Maart 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G., and Holmes, E. C. (2002). Snelheid van de moleculaire evolutie in RNA-virussen: een kwantitatieve fylogenetische analyse. J. Mol. Evol. 54, 156–165. doi: 10.1007 / s00239-001-0064-3
PubMed Abstract / CrossRef Full Text / Google Scholar
Kumar, S., Stecher, G., en Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis version 7.0 voor grotere datasets. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093 / molbev / msw054
PubMed Abstract / CrossRef Full Text / Google Scholar
Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Afwijkend astrovirus geassocieerd met neurologische ziekte bij runderen. Emerg. Infecteren. Dis. 19, 1385–1392. doi: 10.3201 / eid1909. 130682
PubMed Abstract / CrossRef Full Text / Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Volledig humaan immunodeficiëntievirus type 1 genomen van subtype C-geïnfecteerde seroconverters in India, met bewijs van intersubtype recombinatie. J. Virol. 73, 152–160.
PubMed Abstract / Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et al. (2015). Diagnose van neuro-invasieve astrovirus infectie in een immunogecompromitteerde volwassene met encefalitis door onbevooroordeelde volgende generatie sequencing. Clin. Infecteren. Dis. 60, 919–923. doi: 10.1093/cid / ciu912
PubMed Abstract / CrossRef Full Text / Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Pathologie en immunohistochemische bevindingen van West Nile virus infectie in psittaciformes. Dierenarts. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
PubMed Abstract / CrossRef Full Text / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Een nieuw astrovirus geassocieerd met encefalitis en ganglioneuritis bij gedomesticeerde schapen. Grensoverschrijdend. Emerg. Dis. 64, 677–682. doi: 10.1111 / tbed.12623
PubMed Abstract / CrossRef Full Text / Google Scholar
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Astrovirus encefalitis bij jongen met X-linked agammaglobulinemie. Emerg. Infecteren. Dis. 16, 918–925. doi: 10.3201 / eid1606. 091536
PubMed Abstract / CrossRef Full Text / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L., en Pybus, O. G. (2016). Het verkennen van de temporale structuur van heterochrone sequenties met behulp van TempEst (voorheen Path-O-Gen). Virus Evol. 2: vew007. doi: 10.1093 / ve / vew007
PubMed Abstract / CrossRef Full Text / Google Scholar
Reuter, G., Pankovics, P., en Boros, A. (2018). Niet-suppuratieve (aseptische) meningoencephalomyelitis geassocieerd met neurovirulente astrovirus infecties bij mens en dier. Clin. Microbiol. Rev. 31, e00040-18. doi: 10.1128/CMR.00040-18
PubMed Abstract / CrossRef Full Text / Google Scholar
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Beer, M., et al. (2016). Detectie van een nieuw boviene astrovirus bij een koe met encefalitis. Grensoverschrijdend. Emerg. Dis. 63, 253–259. doi: 10.1111 / tbed.12493
PubMed Abstract / CrossRef Full Text / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A., en Seuberlich, T. (2017a). Frequentie en pathologisch fenotype van boviene astrovirus ch13/NeuroS1 infectie bij neurologisch-ziekte runderen: towards assessment of causality. Virussen 9:E12. doi: 10.3390 / v9010012
PubMed Abstract / CrossRef Full Text / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., en Seuberlich, T. (2017b). Boviene astrovirus infectie bij feedlot runderen met neurologische ziekte in West-Canada. Kunnen. Dierenarts. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., en Seuberlich, T. (2016). Detectie van astrovirus in historische gevallen van Europese sporadische boviene encefalitis, Zwitserland 1958-1976. Voorkant. Dierenarts. Sci. 3:91. doi: 10.3389 / fvets.2016.00091
PubMed Abstract / CrossRef Full Text / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identificatie van een tweede encefalitis-geassocieerd astrovirus bij runderen. Emerg. Microben Infecteren. 5: e5. doi: 10.1038 / emi.2016.5
PubMed Abstract / CrossRef Full Text / Google Scholar
Spinato, M. T., Vince, A., Cai, H., and Ojkic, D. (2017). Identificatie van boviene astrovirus in gevallen van boviene niet-suppuratieve encefalitis in Oost-Canada. Kunnen. Dierenarts. J. 58, 607-609.
Google Scholar
Stein, L. T., Rech, R. R., Harrison, L., and Brown, C. C. (2010). Immunohistochemische studie van rabiësvirus binnen het centrale zenuwstelsel van gedomesticeerde en in het wild levende soorten. Dierenarts. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
PubMed Abstract / CrossRef Full Text / Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A., En Minh, B. Q. (2016). W-IQ-TREE: een snelle online fylogenetische tool voor maximale waarschijnlijkheid analyse. Nucleic Acids Res.44, W232–W235. doi: 10.1093 / nar / gkw256
PubMed Abstract / CrossRef Full Text / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Herontdekking en genomische karakterisering van boviene astrovirussen. J.-Generaal Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0,030817-0
PubMed Abstract / CrossRef Full Text / Google Scholar