Biologia dla kierunków i
efekty uczenia się
- zrozum różnicę między pre-mRNA a mRNA
po transkrypcji eukariotyczne pre-mRNA muszą przejść kilka etapów przetwarzania, zanim będą mogły zostać przetłumaczone. Eukariotyczne (i prokariotyczne) tRNA i rRNA również przechodzą proces przetwarzania, zanim będą mogły funkcjonować jako składniki w maszynach syntezy białek.
przetwarzanie mRNA
eukariotyczny pre-mRNA przechodzi intensywne przetwarzanie, zanim będzie gotowy do przetłumaczenia. Dodatkowe etapy związane z dojrzewaniem eukariotycznego mRNA tworzą cząsteczkę o znacznie dłuższym okresie półtrwania niż prokariotyczny mRNA. MRNA eukariotyczne trwają kilka godzin, podczas gdy typowy mRNA E. coli trwa nie dłużej niż pięć sekund.
Pre-mRNA są najpierw pokryte białkami stabilizującymi RNA; chronią one pre-mRNA przed degradacją, gdy jest przetwarzany i eksportowany z jądra. Trzy najważniejsze etapy przetwarzania pre-mRNA to dodanie czynników stabilizujących i sygnalizujących na końcach 5′ I 3 ’ cząsteczki oraz usunięcie sekwencji interweniujących, które nie określają odpowiednich aminokwasów. W rzadkich przypadkach transkrypt mRNA może być „edytowany” po jego transkrypcji.
5′ Capping
podczas gdy pre-mRNA jest nadal syntetyzowany, Kap 7-metyloguanozyna jest dodawana do 5 ’ końca rosnącego transkryptu przez połączenie fosforanowe. To ugrupowanie (Grupa funkcjonalna) chroni rodzący się mRNA przed degradacją. Ponadto czynniki zaangażowane w syntezę białek rozpoznają cap, aby pomóc w inicjacji translacji przez rybosomy.
3′ ogon Poli-A
po zakończeniu wydłużenia pre-mRNA jest rozszczepiany przez endonukleazę między sekwencją konsensusu aauaaa a sekwencją bogatą w gu, pozostawiając sekwencję aauaaa na pre-mRNA. Enzym zwany polimerazą Poli-a następnie dodaje ciąg około 200 reszt a, zwany ogonem Poli-A. Ta modyfikacja dodatkowo chroni pre-mRNA przed degradacją i sygnalizuje eksport czynników komórkowych, których transkrypt potrzebuje do cytoplazmy.
pre-mRNA Splicing
geny eukariotyczne składają się z egzonów, które odpowiadają sekwencjom kodującym białka (ex-on oznacza, że są one ekspresowane), oraz sekwencji interweniujących zwanych intronami (intron oznacza ich rolę interweniującą), które mogą być zaangażowane w regulację genów, ale są usuwane z pre-mRNA podczas przetwarzania. Sekwencje intronowe w mRNA nie kodują funkcjonalnych białek.
odkrycie intronów było zaskoczeniem dla naukowców w latach 70., którzy spodziewali się, że pre-mRNA będą określać sekwencje białkowe bez dalszego przetwarzania, jak zaobserwowano u prokariotów. Geny wyższych eukariotów bardzo często zawierają jeden lub więcej intronów. Regiony te mogą odpowiadać sekwencjom regulatorowym; jednak biologiczne znaczenie posiadania wielu intronów lub posiadania bardzo długich intronów w genie jest niejasne. Możliwe, że introny spowalniają ekspresję genów, ponieważ transkrypcja pre-mRNA z dużą ilością intronów zajmuje więcej czasu. Alternatywnie, introny mogą być niefunkcjonalnymi resztkami sekwencji pozostałymi po fuzji starożytnych genów w trakcie ewolucji. Jest to poparte faktem, że oddzielne egzony często kodują oddzielne podjednostki lub domeny białkowe. W przeważającej części sekwencje intronów mogą być zmutowane bez wpływu na produkt białkowy.
wszystkie introny pre-mRNA muszą zostać całkowicie i dokładnie usunięte przed syntezą białek. Jeśli proces przebiega nieprawidłowo nawet przez pojedynczy nukleotyd, rama odczytu połączonych eksonów ulegnie przesunięciu, a powstałe białko będzie dysfunkcyjne. Proces usuwania intronów i ponownego łączenia egzonów nazywa się splicingiem (ryc. 1). Introny są usuwane i degradowane, gdy pre-mRNA jest jeszcze w jądrze. Splicing zachodzi za pomocą specyficznego dla sekwencji mechanizmu, który zapewnia usunięcie intronów i ponowne połączenie eksonów z dokładnością i precyzją pojedynczego nukleotydu. Splicing pre-mRNA jest przeprowadzany przez kompleksy białek i cząsteczek RNA zwane spliceosomami.
pytanie praktyczne
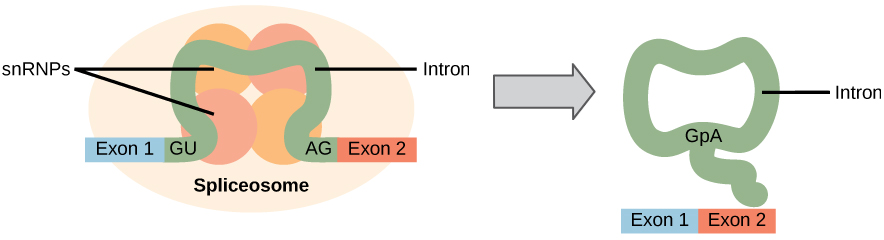
Rysunek 1. Splicing Pre-mRNA polega na precyzyjnym usunięciu intronów z pierwotnego transkryptu RNA. Proces splicingu jest katalizowany przez kompleksy białkowe zwane spliceosomami, które składają się z białek i cząsteczek RNA zwanych snRNA. Spliceosomy rozpoznają sekwencje na 5′ I 3 ’ końcu intronu.
błędy w splicingu są związane z nowotworami i innymi chorobami człowieka. Jakie rodzaje mutacji mogą prowadzić do błędów splicingu?
zauważ, że może być obecnych ponad 70 pojedynczych intronów, a każdy musi przejść proces splicingu-oprócz 5 ’ capping i dodania ogona Poli-a-tylko w celu wytworzenia pojedynczej, translowalnej cząsteczki mRNA.
Edycja RNA w Trypanosomach
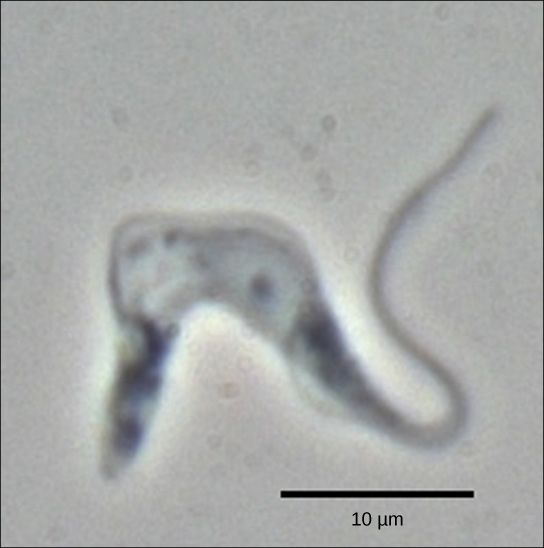
Rysunek 2. Trypanosoma brucei jest czynnikiem sprawczym śpiączki u ludzi. MRNA tego patogenu muszą zostać zmodyfikowane przez dodanie nukleotydów, zanim dojdzie do syntezy białek. (źródło: modyfikacja pracy Torstena Ochsenreitera)
trypanosomy są grupą pierwotniaków, które obejmują patogen Trypanosoma brucei, który powoduje śpiączkę u ludzi (ryc. 2). Trypanosomy i praktycznie wszystkie inne eukarioty mają organelle zwane mitochondriami, które dostarczają komórce energię chemiczną. Mitochondria są organellami, które wyrażają własne DNA i uważa się, że są pozostałościami symbiotycznej relacji między eukariotem a pochłoniętym prokariotem. Mitochondrialne DNA trypanosomów wykazuje interesujący wyjątek od dogmatu Centralnego: ich pre-mRNA nie mają prawidłowych informacji, aby określić funkcjonalne białko. Zazwyczaj dzieje się tak dlatego, że w mRNA brakuje kilku nukleotydów U. Komórka wykonuje dodatkowy etap przetwarzania RNA zwany edycją RNA, aby temu zaradzić.
inne geny w genomie mitochondrialnym kodują 40-do 80-nukleotydowe RNA. Jedna lub więcej z tych cząsteczek oddziałuje przez komplementarne parowanie Zasady z niektórymi nukleotydami w transkrypcji pre-mRNA. Jednak przewodnik RNA ma więcej nukleotydów a niż pre-mRNA ma nukleotydy U do wiązania. W tych regionach RNA przewodnika zapętla się. 3 ’ końce prowadnic RNA mają długi ogon Poli-U, a te podstawy U są wstawiane w regiony transkryptu pre-mRNA, w których prowadnice RNA są zapętlone. Proces ten jest w całości pośredniczony przez cząsteczki RNA. Oznacza to, że prowadnice RNA—zamiast białek—służą jako katalizatory w edycji RNA.
edytowanie RNA to nie tylko zjawisko trypanosomów. W mitochondriach niektórych roślin prawie wszystkie pre-mRNA są edytowane. Edycja RNA została również zidentyfikowana u ssaków, takich jak szczury, króliki, a nawet ludzie. Jaka może być ewolucyjna przyczyna tego dodatkowego kroku w przetwarzaniu pre-mRNA? Jedną z możliwości jest to, że mitochondria, będące pozostałościami starożytnych prokariotów, mają równie starożytną metodę regulacji ekspresji genów opartą na RNA. Na poparcie tej hipotezy zmiany dokonane na pre-mRNA różnią się w zależności od warunków komórkowych. Chociaż spekulatywny, proces edycji RNA może być opóźnieniem z pierwotnego czasu, kiedy cząsteczki RNA, zamiast białek, były odpowiedzialne za katalizowanie reakcji.
Spróbuj
Przyczyń Się!
popraw tę stronęucz się więcej