Biologia dla kierunku II
efekty uczenia się
- zrozumienie przeszłych i powstających systemów klasyfikacji wirusów
ponieważ większość wirusów prawdopodobnie wyewoluowała z różnych przodków, metody systematyczne, które naukowcy zastosowali do klasyfikacji komórek prokariotycznych i eukariotycznych, nie są zbyt użyteczne. Jeśli wirusy stanowią „pozostałości” różnych organizmów, to nawet analiza genomowa lub białkowa nie jest przydatna. Dlaczego?, Ponieważ wirusy nie mają wspólnej sekwencji genomowej, którą wszystkie dzielą. Na przykład Sekwencja 16S rRNA tak przydatna do konstruowania prokaryote phylogenies nie jest przydatna dla stworzenia bez rybosomów! Biolodzy stosowali w przeszłości kilka systemów klasyfikacji. Wirusy były początkowo pogrupowane według wspólnej morfologii. Później grupy wirusów były klasyfikowane według rodzaju kwasu nukleinowego, który zawierały, DNA lub RNA oraz tego, czy ich kwas nukleinowy był jedno – czy dwuniciowy. Jednak te wcześniejsze metody klasyfikacji grupowały wirusy inaczej, ponieważ opierały się na różnych zestawach znaków wirusa. Najczęściej stosowaną dziś metodą klasyfikacji jest tzw. schemat klasyfikacji Baltimore ’ a i opiera się na sposobie generowania messenger RNA (mRNA) w każdym konkretnym typie wirusa.
wcześniejsze systemy klasyfikacji
wirusy zawierają tylko kilka elementów, według których można je sklasyfikować: Genom wirusowy, Typ kapsydu i strukturę otoczki dla wirusów otoczkowych. Wszystkie te elementy były wykorzystywane w przeszłości do klasyfikacji wirusów (Tabela 1 i rycina 1). Genomy wirusowe mogą różnić się rodzajem materiału genetycznego (DNA lub RNA) i jego organizacją (jedno – lub dwuniciowe, liniowe lub kołowe oraz segmentowane lub nie segmentowane). W niektórych wirusach dodatkowe białka potrzebne do replikacji są związane bezpośrednio z genomem lub zawarte w wirusowym kapsydzie.
| Tabela 1. Klasyfikacja wirusów w strukturze genomu i jądra | |
|---|---|
| Podstawowe klasyfikacji | Przykłady |
| RNA | Wirus wścieklizny, retrowirusy |
| DNA | Wirusy opryszczki, wirus ospy |
| Одноцепочечный | Wirus wścieklizny, retrowirusy |
| Двухцепочечные | Wirusy opryszczki, wirus ospy |
| Liniowy | Wirus wścieklizny, retrowirusy, wirusy opryszczki, wirus ospy |
| Okólnik | Papillomawirusy, wiele bakteriofagów |
| bez segmentacji: Genom składa się z pojedynczego segmentu materiału genetycznego | wirusów parainfluenzy |
| segmentacja: Genom jest podzielony na wiele segmentów | wirusy grypy |
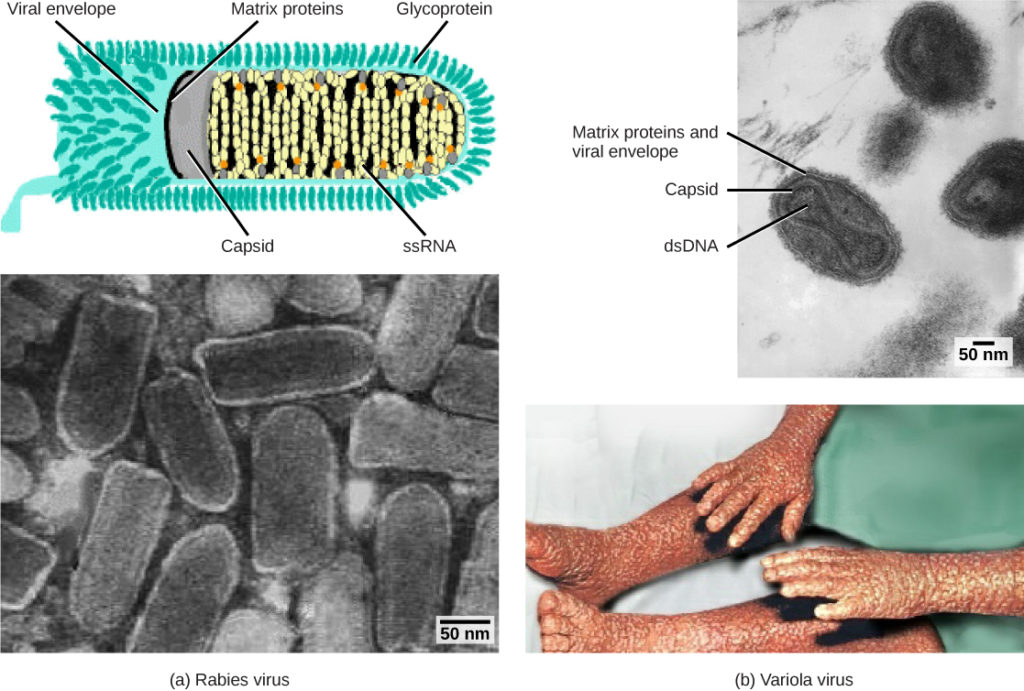
Rysunek 1. Wirusy są klasyfikowane na podstawie ich podstawowego materiału genetycznego i konstrukcji kapsydu. a) wirus wścieklizny ma jednoniciowy rdzeń RNA (ssRNA) i otoczkę spiralnego kapsydu, podczas gdy B) wirus variola, czynnik sprawczy ospy, ma dwuniciowy rdzeń DNA (dsDNA) i złożony kapsyd. (kredyt „rabies diagram”: modyfikacja pracy przez CDC; „rabies micrograph”: modyfikacja pracy przez dr Fred Murphy, CDC; kredyt „small pox micrograph”: modyfikacja pracy przez dr Fred Murphy, Sylvia Whitfield, CDC; kredyt „smallpox photo”: modyfikacja pracy przez CDC; skala-dane barowe od Matta Russella)
wirusy mogą być również klasyfikowane na podstawie konstrukcji ich kapsydów (Tabela 2 i rysunek 2). Capsids są klasyfikowane jako nagie icosahedral, otoczone icosahedral, otoczone spiralne, nagie spiralne i złożone. Rodzaj materiału genetycznego (DNA lub RNA) i jego struktura (jedno – lub dwuniciowe, liniowe lub okrągłe, segmentowane lub nie segmentowane) są wykorzystywane do klasyfikacji struktur rdzenia wirusa (Tabela 2).
| Tabela 2. Klasyfikacja wirusów według struktury kapsydu | |
|---|---|
| Klasyfikacja Capsid | przykłady |
| Naga ikosahedral | wirus zapalenia wątroby typu A, poliowirusy |
| wirus Epsteina-Barr, wirus opryszczki pospolitej, wirus różyczki, wirus żółtej febry, HIV-1 | |
| wirusy grypy, wirus świnki, wirus odry, wirus wścieklizny | |
| Naked helical | wirus mozaiki tytoniowej |
| kompleks z wieloma białkami; niektóre mają kombinacje struktur ikosahedralnych i spiralnych kapsydów | herpeswirusy, wirus ospy, wirus zapalenia wątroby typu B, bakteriofag T4 |

Rysunek 2. Transmisyjne mikrofotografie elektronów różnych wirusów pokazują ich struktury. Kapsyd wirusa (a) polio jest nagi, ikosahedryczny; (b) kapsyd wirusa Epsteina-Barra jest otoczony, ikosahedryczny; (c) kapsyd wirusa świnki jest otoczoną spiralą; (d) kapsyd wirusa mozaiki tytoniowej jest nagi, spiralny; oraz (e) kapsyd herpeswirusa jest złożony. (kredyt a: modyfikacja pracy przez Dr. Fred Murphy, Sylvia Whitfield; kredyt b: modyfikacja pracy przez Liza Gross; kredyt c: modyfikacja pracy przez Dr. F. A. Murphy, CDC; kredyt d: modyfikacja pracy przez USDA ARS; kredyt e: modyfikacja pracy Lindy Stannard, Department of Medical Microbiology, University of Cape Town, South Africa, NASA; dane w skali barowej od Matta Russella)
Klasyfikacja Baltimore
najczęściej używany system klasyfikacji wirusów został opracowany przez biologa, laureata Nagrody Nobla Davida Baltimore ’ a we wczesnych latach 70. XX wieku.oprócz wspomnianych wyżej różnic w morfologii i genetyce, schemat klasyfikacji Baltimore grupuje wirusy zgodnie z tym, w jaki sposób mRNA jest wytwarzane podczas cyklu replikacyjnego wirusa.
wirusy z grupy I zawierają dwuniciowy DNA (dsDNA) jako swój genom. Ich mRNA jest wytwarzany przez transkrypcję w taki sam sposób, jak w przypadku dna komórkowego.
wirusy z grupy II mają jednoniciowe DNA (ssDNA) jako swój genom. Przekształcają swoje jednoniciowe genomy w półprodukt dsDNA przed transkrypcją do mRNA.
wirusy z grupy III wykorzystują dsRNA jako swój genom. Nici rozdzielają się, a jedna z nich jest używana jako szablon do wytwarzania mRNA przy użyciu zależnej od RNA polimerazy RNA kodowanej przez wirusa.
wirusy z grupy IV mają ssRNA jako swój genom z dodatnią polaryzacją. Dodatnia polaryzacja oznacza, że genomowy RNA może służyć bezpośrednio jako mRNA. Półprodukty dsRNA, zwane replikacyjnymi półproduktami, powstają w procesie kopiowania genomowego RNA. Z tych półproduktów powstają liczne, pełnej długości nici RNA o ujemnej polaryzacji (komplementarne do dodatniego genomowego RNA), które mogą następnie służyć jako szablony do produkcji RNA o dodatniej polaryzacji, w tym zarówno genomowego RNA pełnej długości, jak i krótszych wirusowych mRNA.
wirusy z grupy V zawierają genomy ssRNA o ujemnej polaryzacji, co oznacza, że ich sekwencja jest komplementarna do mRNA. Podobnie jak w przypadku wirusów z grupy IV, półprodukty dsRNA są używane do tworzenia kopii genomu i wytwarzania mRNA. W tym przypadku Genom ujemny może zostać przekształcony bezpośrednio do mRNA. Dodatkowo, dodatnie nici RNA o Pełnej długości służą jako szablony do produkcji genomu o ujemnej nici.
wirusy z grupy VI mają diploidalne (dwie kopie) genomy ssRNA, które muszą zostać przekształcone, przy użyciu enzymu odwrotnej transkryptazy, do dsDNA; dsDNA jest następnie transportowany do jądra komórki gospodarza i wstawiany do genomu gospodarza. Następnie mRNA może być wytwarzany przez transkrypcję wirusowego DNA, który został zintegrowany z genomem gospodarza.
wirusy z grupy VII mają częściowe genomy dsDNA i tworzą półprodukty ssRNA, które działają jako mRNA, ale są również przekształcane z powrotem w genomy dsDNA przez odwrotną transkryptazę, niezbędną do replikacji genomu. Charakterystykę każdej grupy w klasyfikacji Baltimore podsumowano w tabeli 3 z przykładami każdej grupy.
| Tabela 3. Klasyfikacja Baltimore | |||
|---|---|---|---|
| Grupa | charakterystyka | tryb produkcji mRNA | przykład |
| I | dwuniciowe DNA | mRNA jest transkrybowane bezpośrednio z szablonu DNA | Herpesvirus) |
| II | jednoniciowe DNA | DNA jest przekształcane do postaci dwuniciowej przed transkrypcją RNA | parwowirus psów (parwowirus) |
| III | dwuniciowy RNA | mRNA jest transkrybowany z Genom RNA | dziecięce zapalenie żołądka i jelit (rotawirus) |
| IV | jednoniciowy RNA (+) | Genom funkcjonuje jako mRNA | przeziębienie (pircornawirus) |
| V | jednoniciowy RNA (−) | mRNA jest transkrybowany z genomu RNA | wścieklizna (rabdowirus) |
| VI | jednoniciowe wirusy RNA z odwrotną transkryptazą | odwrotna transkryptaza wytwarza DNA z genomu RNA; DNA jest następnie włączane do genomu gospodarza; | ludzki wirus niedoboru odporności (HIV) |
| VII | dwuniciowe wirusy DNA z odwrotną transkryptazą | genomem wirusowym jest dwuniciowe DNA, ale DNA wirusowe jest replikowane przez pośredni RNA; RNA może służyć bezpośrednio jako mRNA lub jako szablon do tworzenia mRNA | wirus zapalenia wątroby typu B (hepadnavirus) |