Creative Proteomics Blog
sekwencja aminokwasów w białku lub peptydzie może być zidentyfikowana przez degradację Edmana, która została opracowana przez Pehra Edmana. Ta metoda może oznaczać i rozszczepiać peptyd Z N-terminala bez zakłócania wiązań peptydowych między innymi reszt aminokwasowych. Reakcja degradacji Edmana została zautomatyzowana w 1967 roku przez Edmana i Beggsa. Obecnie powszechnie stosuje się automatyczną degradację Edmana (sekwenator białek) i może sekwencjonować peptydy do 50 aminokwasów.
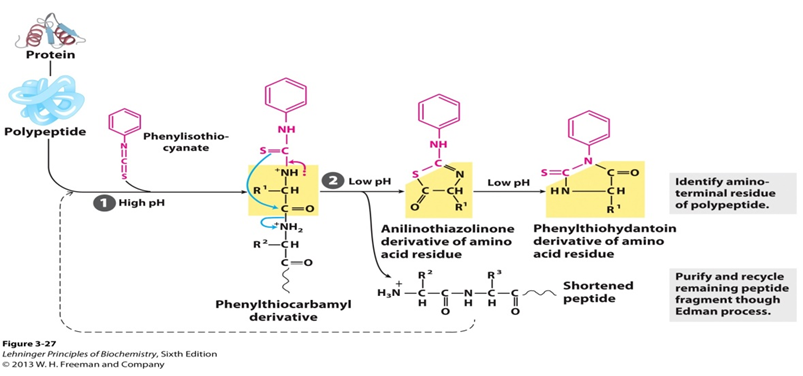
cykliczny rozkład peptydów oparty na reakcji fenyloizotiocyjanianu z wolną grupą aminową N-końcowej reszty, tak że aminokwasy są usuwane pojedynczo i identyfikowane jako ich pochodne fenylotiohydantoiny. Mówiąc o specyficznym procesie, niezaładowany peptyd poddaje się reakcji z fenyloizotiocyjanianem (PITC) na końcu aminowym w Warunkach lekko zasadowych, otrzymując pochodną fenylotiokarbamoilową (peptyd PTC). Następnie, w warunkach kwaśnych, Siarka tiokarbonylowa pochodnej atakuje węgiel karbonylowy N-końcowego aminokwasu. Pierwszy aminokwas jest rozszczepiany jako pochodna anilinotiazolinonu (aminokwas ATZ), a pozostała część peptydu może być wyizolowana i poddana kolejnemu cyklowi degradacji. Po utworzeniu pochodna tiazolonu jest bardziej stabilna niż pochodna fenylotiokarbamylu. Aminokwas ATZ jest następnie usuwany przez ekstrakcję aceratem etylu i przekształcany w pochodną fenylotiohydantoiny (aminokwas PTH). Chromatografia może być wykorzystana do identyfikacji pozostałości PTH generowanych przez każdy cykl.
jeśli chodzi o automatyczną degradację Edmana, białka mogą być analizowane przez zastosowanie ich w roztworze na filtrze TFA, a następnie załadowane na przyrząd do sekwencjonowania Edmana. Białka w mieszaninach są najpierw oddzielane żelami 1D lub 2D, a następnie blazonowane na błonie PVDF. Białka są wykrywane przez barwienie coomassie blue, amido black lub Poncau S, a białka będące przedmiotem zainteresowania wycinane i kawałek membrany PVDF ładowany na sekwencer Edmana.
dzięki opracowaniu spektrometrii masowej, zastosowanie sekwencjonowania degradacji Edmana zaczęło się zmniejszać. Jednak nadal pozostaje metody dla kilku rodzajów zastosowań analizy strukturalnej białek. Może być stosowany do weryfikacji N-końcowej granicy rekombinowanych białek lub określenia N-końcowej granicy domen opornych na proteazy, szczególnie gdy białko lub domena jest >40 do 80 kDa lub nie może być łatwo oczyszczona. Można go również wykorzystać do identyfikacji nowego N-końcowego i proteolitycznego miejsca rozszczepiania fragmentów białka. Ponadto, w przypadku niektórych nowych białek i peptydów, w których bazy danych sekwencji nie są dostępne do przeszukiwania baz danych MS/MS, degradacja Edmana może być wykorzystana do analizy.
N-końcowy aminokwas białka można oddzielić. Tak więc, w procesie, pierwszy cykl identyfikuje dokładny N-końcowy aminokwas. Ponadto, ponieważ uwolnione aminokwasy są identyfikowane i oznaczane ilościowo za pomocą chromatografii, można zidentyfikować aminokwasy o identycznej masie cząsteczkowej. Na przykład izoleucyna i leucyna mają masę 113 Da, ale mają inny czas retencji. Co więcej, sekwencjonowanie Edmana można przeprowadzić na BLOTACH PVDF z żeli 1D i 2D, co umożliwia N-końcowe sekwencjonowanie białek w mieszaninie. Jednakże, sekwencjonowanie degradacji Edmana nie będzie dostępne, gdy peptyd, którego N-KONIEC został chemicznie zmodyfikowany, taki jak acetylacja. A ponieważ PITC nie może reagować z nie-α-aminokwasem, sekwencjonowanie zatrzyma się, jeśli napotkany zostanie Nie-α-aminokwas, jak kwas izoaspartynowy. Co więcej, większe białka nie mogą być zsekwencjonowane przez sekwencjonowanie Edmana.
w Creative Proteomics, nasza profesjonalna platforma sekwencjonowania białek zapewnia analizę sekwencji N-końcowej za pomocą degradacji Edmana lub spektrometrii masowej (MS), z komplementarnymi zaletami. Nasz zespół ekspertów z dużym doświadczeniem pomoże Ci zrozumieć, co próbujesz zbadać i spełnić twoje wymagania. Ponadto możemy świadczyć inne usługi identyfikacji białek, w tym:
- usługa Peptide mass fingerprinting (PMF)
- analiza sekwencji peptydów lub białek
- De novo usługa analizy sekwencji białek
- usługa identyfikacji białek Shotgun
- usługa proteomiki membranowej