Frontiers in Microbiology
wprowadzenie
Rodzina Astroviridae zawiera bezotoczkowe, dodatnio sensowne, jednoniciowe wirusy RNA w obrębie dwóch rodzajów, Mamastrovirus i Avastrovirus, które infekują odpowiednio ssaki i ptaki. Obecnie Międzynarodowy Komitet taksonomii wirusów (International Committee on Taxonomy of Viruses , 2018) uznaje 19 gatunków, a mianowicie Mamastrovirus-1 do -19, w obrębie rodzaju Mamastrovirus; jednak istnieje wiele szczepów oczekujących na klasyfikację, z których niektóre są wstępnie uważane za nowe gatunki (Donato and Vijaykrishna, 2017).
od 2010 r.kilka astrovirusów jest coraz częściej rozpoznawanych jako neuroinwazyjne u różnych gatunków ssaków, w tym u ludzi (Quan et al., 2010; Naccache et al., 2015), norka (Blomström et al., 2010), bydło (Li et al., 2013), Owce (Pfaff et al., 2017) i świnie (Boros et al., 2017). Po początkowym rozpoznaniu zapalenia mózgu związanego z astrowirusem bydła u bydła w Stanach Zjednoczonych (Li et al., 2013), retrospektywne badanie przypadków sporadycznego bydlęcego zapalenia mózgu o nieokreślonej etiologii ze Szwajcarii wykazało, że ten neuroinwazyjny astrovirus pozostawał niewykryty przez dziesięciolecia (Selimovic-Hamza et al., 2016). Chociaż epidemiologia i drogi transmisji tych astrovirusów są nieznane, zasugerowano przenoszenie międzygatunkowe na podstawie wysokiego poziomu tożsamości (>98%), dzielonego między astrovirusy neuroinwazyjne bydła i owiec na poziomie nukleotydów i aminokwasów (Boujon et al., 2017).
astrowirusy bydlęce (BoAstVs) o nazwie BoAstV-NeuroS1 (Li et al., 2013) i BoAstV-CH13 (Bouzalas et al., 2014), zostały początkowo Znalezione w mózgu bydła z ropnym zapaleniem mózgu odpowiednio w Stanach Zjednoczonych i Szwajcarii. Pomimo odmiennej nomenklatury, oba wirusy reprezentują ten sam gatunek genotypu (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a), który wciąż czeka na oficjalną klasyfikację przez ICTV. W 2015 r. w mózgu krów z zapaleniem mózgu w Szwajcarii zidentyfikowano nieznany wcześniej szczep BoAstV o nazwie BoAstV-CH15. Pełne porównanie filogenetyczne genomu wykazało bliższe pokrewieństwo BoAstV-CH15 z astrowirusem owiec (OvAstV) niż z BoAstV-ch13 (Seuberlich et al., 2016). Koinfekcja z BoAstV-CH13 i BoAstV-CH15 została również udokumentowana w jednym przypadku (Seuberlich et al., 2016). W tym samym roku w Niemczech, Schlottau et al. (2016) poinformował o nowym astrowirusie, mianowicie BoAstV-BH89/14, u krowy z zapaleniem mózgu, który był najbliżej spokrewniony z OvAstV i BoAstV-CH15. Następnie boastv-CH13/NeuroS1 zidentyfikowano w 2017 r. w przypadkach bydlęcego zapalenia mózgu we wschodniej i zachodniej Kanadzie(Spinato et al., 2017; Selimovic-Hamza et al.,2017b). W 2018 r. zidentyfikowano nowy neuroinwazyjny BoAstV ściśle związany z północnoamerykańskim i europejskim Boastv-NeuroS1/BoAstV-CH13 w sterku z nie ropnym zapaleniem mózgu i rdzenia kręgowego w Japonii i zasugerowano występowanie rekombinacji wewnątrzgenotypowej między szczepami północnoamerykańskimi i europejskimi(Hirashima et al., 2018).
chociaż przypadki zapalenia mózgu związanego z astrowirusem odnotowano w Ameryce Północnej, Europie i Azji, ich obecność nigdy nie została udokumentowana na półkuli południowej. Opisujemy tutaj przypadek zapalenia mózgu związanego z astrowirusem u bydła w Urugwaju, które rozszerza rozmieszczenie geograficzne i różnorodność genetyczną neuroinwazyjnych astrovirusów i dostarcza dowodów filogeograficznych, które sugerują, że wirus ten został wprowadzony do obu Ameryk z Europy, a później rozprzestrzenił się na Azję.
materiały i metody
Historia i sygnalizacja
w czerwcu 2018 roku 22-miesięczny wer Holsztyński w grupie 37 wołów na 300-hektarowej farmie w Colonia w Urugwaju rozwinął progresywne neurologiczne śpiewy, w tym niezwykłe zachowanie, bezcelowe chodzenie, krążenie, ataksja, powtarzające się i nieskoordynowane ruchy języka i cofanie się. Stado pasło się na jednorocznym pastwisku owsianym i uzupełniano kiszonką z kukurydzy. Przypuszczalna diagnoza kliniczna listeriozy mózgowej przez lekarza weterynarii skłoniła do leczenia penicyliną i streptomycyną, jednak zwierzę zmarło spontanicznie po 3-dniowym kursie klinicznym.
badanie patologiczne, hybrydyzacja in situ (ISH) i Immunohistochemia (IHC)
głowę wołu usunięto z tuszy i przekazano do weterynaryjnego laboratorium diagnostycznego Inia (Animal Health Platform) w celu przeprowadzenia prac diagnostycznych. Połowę mózgu, krótki odcinek proksymalnego szyjnego rdzenia kręgowego (C1), zwoju trójdzielnego i korzenia nerwu trójdzielnego, gruczoł ślinowy, węzeł chłonny poza gardłem, gardło, gardło, przełyk, język i mięśnie szkieletowe zamocowano zanurzeniowo w 10% neutralnej buforowanej formalinie przez 48-72 godziny. tkanki rutynowo przetwarzano do histologii, osadzano w parafinie, mikrotom odcinano w temperaturze 4-5 µm i barwiono hematoksyliną i eozyną (H&E) i plamy gramowe.
Chromogenne badanie ISH przeprowadzono ręcznie na 5 µm odcinkach pnia mózgu, móżdżku i móżdżku na szkiełkach Superfrost Plus (Thermo Fisher Scientific, Pittsburgh, PA, Stany Zjednoczone) przy użyciu zestawu Rnascope 2.5 Red assay kit (Nr kat. 322360, Advanced Cell Diagnostics, Hayward, CA, Stany Zjednoczone) i sondy BoAstV Cat. #406921. Sonda składa się z 20 par ukierunkowanych na region 5232-6180 wirusa (GenBank KF233994.1). Każdy 5 µm odcinek tkanki poddano wstępnej obróbce termicznej i proteazą przed hybrydyzacją sondy przez 2 godziny w temperaturze 40°C i poddano obróbce zgodnie z zaleceniami producenta. Kontrole negatywne stosowane do walidacji sygnału obejmowały niepowiązaną (dopasowaną do zawartości GC) sondę prowadzoną na odcinkach seryjnych i sondowanie tkanki niezakażonych zwierząt. Szkiełka przeciwstawiono hematoksyliną i zamontowano za pomocą EcoMount (Biocare Medical, Concord, CA, Stany Zjednoczone).
dodatkowo, IHC przeprowadzono w sekcjach FFPE pnia mózgu, móżdżku i móżdżku, jak wcześniej opisano, w celu identyfikacji wirusa Zachodniego Nilu (WNV, Flawiwirus) (Palmieri i wsp ., 2011), wirus wścieklizny (Lyssavirus) (Stein et al., 2010) oraz Chlamydia spp. (Giannitti et al., 2016).
wirusologia molekularna
ekstrakcję kwasu nukleinowego przeprowadzono z połączonej próbki zamrożonego (-20°C) mózgu za pomocą MagMAX Nucleic Acid Isolation Kit® (Thermo Fisher Scientific). W celu wykrycia astrowirusa przeprowadzono odwrotną transkrypcję (RT) za pomocą RevertAid Reverse Transcriptase® (Thermo Fisher Scientific) i random hexamer starters (Qiagen). PCR wykonano z cDNA przy użyciu MangoMix® (Bioline) i starterów, które amplifikują fragment nukleotydu 432 genu polimerazy astrowirusa (Tse i wsp., 2011). Produkt PCR wizualizowano w 2% żelu agarozowym, oczyszczono za pomocą zestawu PureLink® Quick Gel Extraction i PCR Purification Combo Kit (Invitrogen) i zsekwencjonowano w Macrogen Inc. (Seul, Korea Południowa). Dla amplifikacji całego genomu astrowirusa, Maxima H Minus odwrotna Transkryptaza (Thermo Fisher Scientific) i oligo(DT)18 dla uzyskania cDNA i MangoMix® (Bioline) lub polimeraza DNA Rangera (Bioline) z starterami opisanymi przez Hirashima i wsp., 2018, zostały wykorzystane. Produkty PCR wizualizowano W 1-2% żelu agarozowym, oczyszczono i zsekwencjonowano, jak wspomniano powyżej. Montaż sekwencji przeprowadzono za pomocą Seqmana (Lasergene 8, DNASTAR). Dwadzieścia sześć kompletnych sekwencji genomu neuroinwazyjnego astrowirusa bydła, owiec, świń, ludzi i norek oraz jelitowego astrowirusa bydła dostępnego w GenBank pobrano i wyrównano za pomocą Clustal W w oprogramowaniu MEGA 7 (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) został użyty do określenia najlepiej dopasowanego modelu ewolucji sekwencji (SYM+I+G4) i skonstruowania drzewa filogenetycznego o maksymalnym prawdopodobieństwie z prawie kompletnymi sekwencjami boastv wykrytymi w tym przypadku i tymi kompletnymi sekwencjami pobranymi z GenBank, przy użyciu bootstrap jako metody statystycznej do oceny solidności kladów. Wykres podobieństwa przeprowadzono za pomocą oprogramowania SimPlot (Lole et al., 1999). Odległości P na poziomie aminokwasów ORF2 oszacowano za pomocą oprogramowania MEGA 7 (Kumar i wsp ., 2016).
dodatkowo przeprowadzono Bayesowską analizę filogeograficzną z pakietem the BEAST v1.8.4 (Drummond et al., 2012), wykorzystując: kompletny region kodujący linii BoAstV ch13/NeuroS1, ORF1ab (geny niestrukturalne), ORF2 (Gen strukturalny), ORF1a (proteaza) i częściowy ORF1b (region genomowy polimerazy, dla którego dostępne były szczepy kanadyjskie), ze wszystkimi sekwencjami dostępnymi w GenBank (Ostatnie przystąpienie 18 kwietnia 2019 r.), w celu określenia tempa ewolucji, wieku/lat wspólnych przodków i najbardziej prawdopodobnej drogi krążenia wirusa według kraju (Szwajcaria, Urugwaj, Stany Zjednoczone, Kanada i Japonia). Brak rekombinacji w zbiorze danych określono za pomocą programu wykrywania rekombinacji 4. Model substytucji, który najlepiej pasuje do każdego wyrównania, określono za pomocą oprogramowania MEGA 7 za pomocą wartości bayesowskiego kryterium informacyjnego (BIC), a strukturę czasową każdego zbioru danych oceniono za pomocą TempEst (Rambaut et al., 2016). Lognormalny zegar molekularny z analizą Bayesa został wybrany przez współczynnik Bayesa spośród różnych kombinacji zegarów molekularnych i koalescencyjnych drzew. Jako cechę użyto kraju wykrycia. Długość Łańcucha Markowa wynosiła 100 milionów generacji, co zapewniało zbieżność analizy, ocenianej w Tracer v1. 6.0, a do oceny kladów użyto prawdopodobieństwa tylnego. Maksymalne drzewo wiarygodności kladu (MCCT) zostało uzyskane za pomocą oprogramowania TreeAnnotator od BEAST i wizualizowane w FigTree v1.4.3.
wreszcie DNA wyekstrahowane z zamrożonego mózgu zostało przetworzone metodą PCR w celu wykrycia opryszczek bydlęcych 1 i 5 (BHV-1 i -5), jak wcześniej opisano (Ashbaugh i wsp., 1997).
Bakteriologia
świeże próbki móżdżku i pnia mózgu były rutynowo przetwarzane na tlenowe kultury bakterii we krwi i agarach MacConkey ’ a oraz na selektywną hodowlę Listeria monocytogenes (Al-Zoreky and Sandine, 1990).
wyniki i dyskusja
objawy kliniczne i wyniki epidemiologiczne w opisanym tu przypadku, choć niespecyficzne, były podobne do tych opisanych w innych przypadkach zapalenia mózgu związanego z astrowirusem bydła, które jest zwykle opisywane jako sporadyczne (Selimovic-Hamza i wsp., 2016), z różnymi deficytami neurologicznymi (Deiss et al., 2017), z czasem trwania objawów klinicznych, które zwykle wahają się od 1 dnia do 3 tygodni (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).
badanie makroskopowe mózgu, odcinka C1 rdzenia kręgowego i innych tkanek głowy nie wykazało znaczących zmian anatomicznych. Histologicznie stwierdzono umiarkowane do ciężkiego, limfocytowe, histiocytarne i plazmocytowe zapalenie opon mózgowo-rdzeniowych, które dotyczyło telencefalonu (w tym półkuli mózgowej i hipokampa), pnia mózgu i jedynego zbadanego odcinka rdzenia kręgowego. Zmiany były rozmieszczone głównie w istocie szarej i ograniczonych obszarach istoty białej. W dotkniętych obszarach stwierdzono zapalenie okołonaczyniowe i limfoplazmatyczne i histiocytarne oraz martwicę neuronów / neuronofagię z gliozą w sąsiednim neuropilu. Stwierdzono satellitozę uszkodzonych, martwiczych neuronów (ryc. 1a-D). Zmiany te były znacznie rzadsze i ciężkie w miąższu móżdżku, chociaż występowało wieloogniskowe umiarkowane zapalenie leptomening móżdżku. Nie stwierdzono bakterii wewnątrzustnych z Plamami H&E i grama. W pozostałych badanych tkankach nie stwierdzono istotnych zmian histologicznych.

Rysunek 1. Zmiany histologiczne w pniu mózgu (A, B)i korze mózgowej (C, D) oraz wykrywanie RNA BoAstV w korze mózgowej (E, G). Obrazy (A-D)to fragmenty mózgu zabarwione H& E; obrazy E i G są sekcjami kory mózgowej wykazującymi hybrydyzację przy użyciu chromogennego ISH przy użyciu specyficznej sondy BoAstV, przeciwstawionej hematoksyliną; zdjęcie F to szeregowy odcinek sondy dapb kory mózgowej (Kontrola negatywna), przeciwstawionej hematoksyliną. A) przestrzeń okołoporodowa jest znacznie rozszerzona przez komórki zapalne (głównie limfocyty i histiocyty), które również infiltrują sąsiedni neuropil. (B) neuron w centrum ma hipereozynofilowe perikaryon i karyorrhexis (martwica), a ciało neuronalne jest otoczone zwiększoną liczbą glejowych (satellitosis) i komórek zapalnych. C) leptomening jest naciekany przez limfocyty i histiocyty. (D) obszar kory mózgowej z wieloma hipereozynofilowymi (martwiczymi) neuronami i dużym naczyniem z okołonaczyniowymi mankietami limfocytowymi. (E) w seryjnym odcinku kory mózgowej, obfite wewnątrzcytoplazmatyczne etykietowanie BoAstV RNA jest przedstawione przez silne, ziarniste osadzanie czerwonego chromogenu w neuronalnej cytoplazmie soma i rozszerzeniach neuronalnych (E,G), które nie występuje przy hybrydyzacji przy użyciu ujemnej sondy kontrolnej (F).
podczas badania histologicznego ośrodkowego układu nerwowego podejrzewano neuroinwazyjne zakażenie wirusowe. Bydło z zapaleniem mózgu jest niepokojące, ponieważ wiele neuropatogenów przeżuwaczy jest odzwierzęcych (Cantile i Youssef, 2016); tak więc diagnoza zapalenia mózgu powinna skłaniać do rozległych badań laboratoryjnych w celu wykrycia czynników zakaźnych, jeśli to możliwe. W przypadku opisanym w niniejszym dokumencie, IHC dla WNV, wirusa wścieklizny i Chlamydia spp. i PCR dla BHV-1 i -5 były ujemne, a z tkanki mózgowej nie wyhodowano żadnych patogennych bakterii. Ponieważ byk miał < 2 lata i nie zaobserwowano zmian gąbczastych w pniu mózgu, zwierzę nie zostało zbadane pod kątem gąbczastej encefalopatii bydła (BSE), która jest egzotyczną chorobą dorosłego bydła, która nigdy nie została zgłoszona w Urugwaju. Ponadto BSE nie jest stanem zapalnym (Cantile and Youssef, 2016).
hybrydyzację in situ przeprowadzono za pomocą sondy Wygenerowanej z Boastv-NeuroS1, a hybrydyzację sondy przeprowadzono obficie wewnątrz i ograniczono ją do cytoplazmy neuronów półkuli mózgowej i hipokampa (Fig.1e–G). W tych obszarach hybrydyzacja sondy kolokalizuje się z neuronami martwiczymi i regionami gliozy, bez hybrydyzacji sondy wykrywalnej w komórkach glejowych lub komórkach zapalnych mankietów okołonaczyniowych. Nie wykryto wirusowego kwasu nukleinowego w móżdżku, który miał tylko minimalne zmiany zapalne w miąższu, ale umiarkowane zapalenie leptomening lub pnia mózgu, w tym sekcje z ciężkim stanem zapalnym. Oznacza to, że topograficznie wykrywanie dystrybucji wirusa przez ISH było bardziej ograniczone niż zapalenie mózgu w badanych sekcjach, które czasami opisywano w przypadkach zapalenia mózgu związanego z BoAstV-CH13 / NeuroS1 u bydła(Selimovic-Hamza i wsp .,2017a, b). Powodem tego sporadycznego braku wykrywania wirusowego RNA w uszkodzonych obszarach mózgu może być granica wykrywalności ISH lub klirens wirusa w zapalnych obszarach mózgu do czasu śmierci, jak wcześniej sugerowano (Selimovic-Hamza et al.,2017b). Zgodnie z oczekiwaniami, nie wykryto hybrydyzacji sondy przez ISH w tkance mózgowej wykorzystywanej jako kontrola negatywna.
Astrowirus wykryto w mózgu metodą RT-PCR. Prawie kompletna analiza sekwencji genomu ujawniła szczep Mamastrovirus w kladzie Ch13 / NeuroS1, nazwaliśmy BoAstV-Neuro-Uy, sekwencja została zdeponowana w GenBank pod numerem mk386569. Analiza filogenetyczna wykazała bliskość z innymi neuroinwazyjnymi astrovirusami w obrębie kladu Virginia / Human-Nork-Sheep (VA / HMO) (Rysunek 2), który zawiera najbardziej znane neuroinwazyjne astrovirusy (Hirashima et al., 2018; Reuter et al., 2018). Prawie kompletna Sekwencja BoAstV-Neuro-Uy ma długość 6427 bp I 94% identyczności sekwencji ze szczepem KagoshimaSR28-462. BoAstV-Neuro-Uy ma podobne cechy jak inne szczepy linii ch13 / NeuroS1: region 5 ’ UTR 51 nt, orf1a (proteaza) 861 aminokwasów (aa), ORF1b 523 aa (polimeraza RNA zależna od RNA) i ORF2 758 aa (białko kapsyd). Niestety, 3 ’ UTR nie mógł być zsekwencjonowany, ale przypuszcza się, że istnieje ogon Poli(a), ponieważ oligo(dT)18 został użyty do uzyskania cDNA. Ponadto występuje heptameryczna Sekwencja aaaaac, rybosomalny sygnał zmiany ramki. P-odległości na poziomie aminokwasów ORF2 potwierdziły przypisanie tego szczepu do kladu CH13 / NeuroS1. P-odległości < 0.35 między BoAstV-Neuro-Uy i innymi członkami tego kladu (Tabela 1) poparłoby klasyfikację tych szczepów wirusowych w obrębie jednego gatunku; Mamastrovirus-13 został niedawno zaproponowany przez innych autorów (Donato and Vijaykrishna, 2017; Hirashima et al., 2018), choć w toku jest definitywne przypisanie gatunku przez ICTV. Sonda używana do ISH, wygenerowana z Boastv-NeuroS1, miała 92,7% identyczności sekwencji z BoAstV-Neuro-Uy.
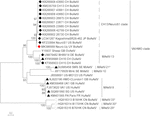
Rysunek 2. Filogenetyczna analiza pełnowymiarowych sekwencji nukleotydowych metodą maksimum prawdopodobieństwa. BoAstV-Neuro-Uy jest oznaczony czerwonym rombem. Inne sekwencje bydlęcych neuroinwazyjnych astrovirusów są oznaczone czarnym rombem, sekwencje z nie-bydlęcych neuroinwazyjnych astrovirusów są oznaczone czarnymi trójkątami, a sekwencje z jelitowych astrovirusów bydlęcych są oznaczone białymi kółkami. Pokazano gatunki mamastrowirusów; gwiazdki wskazują na gatunki, które nie zostały jeszcze rozpoznane przez ICTV. Szczepy należące do kladu CH13/NeuroS1, które nie zostały jeszcze przypisane do gatunku przez ICTV, są identyfikowane z linią przerywaną. Wartości Bootstrap są wskazywane w węzłach. CH, Szwajcaria; JP, Japonia; USA, Stany Zjednoczone Ameryki; UY, Urugwaj; GB, Zjednoczone Królestwo Wielkiej Brytanii; DE, Niemcy; SE, Szwecja; FR, Francja; CN, Chiny. BoAstV, astrowirus bydła; OvAstV, astrowirus owiec; PoAstV, astrowirus świń; HuAstV, astrowirus ludzki; MiAstV, astrowirus norek.
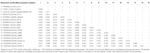
Tabela 1. Oszacowanie różnic ewolucyjnych na poziomie aminokwasów w całym regionie ORF2 pomiędzy sekwencjami Mamastrowirusa-13 bydła i owiec a szczepami w obrębie kladu Ch13 / NeuroS1, które nie zostały jeszcze przypisane do gatunku przez Międzynarodowy Komitet taksonomii wirusów (ICTV).
badania oparte na badaniach neuropatologicznych i wykrywaniu kwasu nukleinowego i białka astrowirusa wykazały, że istnieje prawdopodobny związek przyczynowy między zakażeniem astrowirusem a chorobą neurologiczną i zmianami u bydła (Selimovic-Hamza i in., 2017a; Reuter et al., 2018). Zgodnie z naszą najlepszą wiedzą astrowirusowe zapalenie mózgu nie zostało jeszcze odtworzone eksperymentalnie. Wymagałoby to izolacji neuroinwazyjnych astrovirusów od przypadków klinicznych, czego w naszym przypadku nie próbowano.
źródło BoAstV-Neuro-Uy w tym przypadku nie może być określone. Należy jednak wziąć pod uwagę bydło wodne i dzikie zwierzęta, ponieważ bydło hodowano w rozległych warunkach zewnętrznych. Dotknięte zwierzę zostało zakupione i przeniesione do gospodarstwa w lutym 2018 roku wraz z innymi 9 wołami. Niestety właściciel odmówił dalszego pobierania próbek i testowania innych zwierząt w nieruchomości oraz bardziej szczegółowego badania epidemiologicznego. Żadne z pozostałych zwierząt w grupie nie rozwinęło choroby neurologicznej w sierpniu 2018 r., po raz ostatni skontaktowano się z lekarzem weterynarii. Sezonowość od początku zimy do końca wiosny została zasugerowana w przypadkach zapalenia mózgu związanego z astrowirusem w Szwajcarii(Selimovic-Hamza et al., 2016). Co ciekawe, opisany tu przypadek miał miejsce w czerwcu, odpowiadającym jesienno-zimowemu okresowi przejściowemu na półkuli południowej.
podczas gdy astrovirusy neurotropowe zostały zidentyfikowane w Ameryce Północnej (Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) i Azji (Hirashima et al., 2018), ich obecność nigdy nie została zgłoszona na półkuli południowej, więc ta komunikacja rozszerza rozmieszczenie geograficzne związanego z astrowirusem zapalenia mózgu. Aby ocenić, czy szczep wirusa wykryty w Urugwaju mógł pochodzić z Europy, Ameryki Północnej lub Azji, oszacowaliśmy tempo ewolucji i przeprowadziliśmy analizę filogeograficzną z wykorzystaniem neuroinwazyjnych sekwencji BoAstV dostępnych w GenBank. Tempo ewolucji oszacowane za pomocą całego regionu kodującego wynosiło 4.27 × 10-4 (95% najwyższej gęstości prawdopodobieństwa-HPD-, 2,19–6,46 × 10-4) podstawienia nukleotydów / miejsce/rok, co jest spodziewane dla wirusa RNA (Jenkins i wsp ., 2002), ale niższe niż szacowane dla jelitowych ludzkich astrovirusów (Babkin et al., 2012, 2014). Region ORF1ab wykazywał podobną ewolucję (4,20 × 10-4, 95% HPD 1,66–6,46 × 10-4 podstawienia/miejsce/rok) jak kompletny region kodujący, podczas gdy ORF1a (2,92 × 10-4, 95% HPD 1,19 × 10-6-6, 46 × 10-4 podstawienia/miejsce/rok) i ORF2 (2,86 × 10-4, 95% HPD 4,13 × 10-6-5.79 × 10-4 podstawienia / miejsce / rok) wykazywało nieco szybszą ewolucję, a częściowy region genomowy polimerazy (ORF1b) wykazywał nieco wolniejszą ewolucję (5,39 × 10-4, 95% HPD 6,41 × 10-7-1, 10 × 10-3 podstawienia/miejsce/rok).
zgodnie z analizą filogeograficzną z kompletnym regionem kodującym i pokazaną w MCTT (Fig. 3), istnieją dwie podrodziny (CH13 i NeuroS1) oparte na szczepach referencyjnych, które mają wspólnego przodka. Ostatni wspólny przodek tych podrodzin (rodowód CH13/NeuroS1) powstał w Europie około 1885 roku (95% HPD, 1794-1940). Na początku 1900 roku, dwie pod-linie rozeszły się, pod – linia CH13 pozostała w obiegu w Europie, podczas gdy pod-linia NeuroS1 rozprzestrzeniła się do Ameryki i Azji. Najbardziej prawdopodobnym scenariuszem jest to, że Podrodzina NeuroS1 została wprowadzona w Urugwaju z Europy około roku 1921 (95% HPD, 1849-1967), prawdopodobnie poprzez handel żywym inwentarzem, a następnie rozprzestrzeniła się do Ameryki Północnej, a później do Japonii (Rysunek 3). Ze względu na ograniczenie liczby sekwencji dostępnych w GenBank, które mogło spowodować stronniczość analizy, wyniki uzyskane przy użyciu pełnego regionu kodującego porównano z wynikami uzyskanymi z innymi regionami genomowymi (ORF1ab, ORF2, ORF1a i ORF1b) dostępnymi dla większej liczby szczepów (tj. szczepów Kanadyjskich). We wszystkich analizach najbardziej prawdopodobny scenariusz zakłada, że wprowadzenie wirusa do Urugwaju nastąpiło z Europy (dodatkowe dane S1A–D). Ponadto szacowana data wprowadzenia, uzyskana z regionem genomowym ORF1ab i częściowej polimerazy (ORF1b) (dodatkowe figury S1A,D), była podobna do daty uzyskanej z kompletnym regionem kodującym, podczas gdy szacowana data wprowadzenia uzyskana z ORF2 i ORF1a była wcześniejsza,ale z szerszym 95% odstępem HPD (dodatkowe figury S1B, C). Prawdopodobne jest również wprowadzenie sub-lineage NeuroS1 bezpośrednio do Kanady z Europy, z późniejszym rozprzestrzenianiem się na Stany Zjednoczone i Japonię, pokazane na rysunku uzupełniającym S1D.

Rysunek 3. Maksymalne drzewo wiarygodności kladu uzyskane na podstawie analizy całego regionu kodowania. Kolor gałęzi reprezentuje najbardziej prawdopodobny kraj, w którym przodkowie krążyli, tylne wartości prawdopodobieństwa są pokazane w gałęziach, a liczby w każdym węźle reprezentują lata pochodzenia dla każdego kladu z interwałem 95% HPD. Podtytuły są oznaczone etykietami.
potrzebne są dalsze badania w celu oceny rozkładu geograficznego, mechanizmów patogennych (w szczególności mechanizmów przenoszenia i wprowadzania), epidemiologii molekularnej i potencjalnego międzygatunkowego przenoszenia neuroinwazyjnych astrovirusów.
dostępność danych
zbiory danych wygenerowane dla tego badania można znaleźć w GenBank, MK386569.
wkład autora
FG, RDC i MC przyczyniły się do koncepcji badania. FG i RDC przeprowadziły badanie patologiczne i pobieranie próbek. PP przeprowadził hybrydyzację in situ. FU przeprowadził immunohistochemię. Lm, RC i MC przeprowadziły badania wirusologiczne molekularne. MC przeprowadził analizy sekwencyjne i filogeograficzne oraz związane z nimi figury. FG i PP uzyskali obrazy histologiczne. MF przeprowadziło hodowle bakterii. FG i MC napisali pierwszy szkic rękopisu. RDC, PP, FU, LM, MF i RC napisał fragmenty rękopisu. Wszyscy autorzy przyczynili się do rewizji manuskryptu, przeczytali i zaakceptowali nadesłaną wersję.
Funding
This work was funded by Grants PL-015 N-15156 from INIA and 158 from the „program inicjowania badań 2017” from „sektorowa Komisja badań naukowych” (CSIC). MC and RDC acknowledge support from the „National Research and Innovation Agency” (ANII) and INIA, respectively, through Ph. D. scholarships. FG acknowledges support from ANII through mobility grant MOV_CA_2018_1_150021.
Oświadczenie o konflikcie interesów
autorzy oświadczają, że badanie zostało przeprowadzone przy braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
recenzent TS zadeklarował wcześniejsze współautorstwo z jednym z autorów.
podziękowania
autorzy dziękują Yisellowi Perdomo i Cecilii Monesiglio z INIA, a Karen Sverlow i Juliann Beingesser z CAHFS za pomoc techniczną.
Materiały uzupełniające
Materiały uzupełniające do tego artykułu można znaleźć w Internecie pod adresem: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
rysunek S1 / maksimum drzew wiarygodności kladów (Mcct) uzyskane przez Analizę pełnej długości ORF1ab (A), Pełnej długości ORF2 (B), Pełnej długości ORF1a (C) i częściowej orf1b (D). Kolor gałęzi reprezentuje najbardziej prawdopodobny kraj, w którym przodkowie krążyli, tylne wartości prawdopodobieństwa są pokazane w gałęziach, a liczby w każdym węźle reprezentują lata pochodzenia dla każdego kladu z interwałem 95% HPD. Podtytuły są oznaczone etykietami.
Przypisy
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., and Sandine, W. E. (1990). Wysoce selektywne podłoże do izolacji Listeria monocytogenes z pożywienia. Appl. Environ. Mikrobiol. 56, 3154–3157.
PubMed Abstract / Google Scholar
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chowdhury, S., and Collins, J. K. (1997). Specyficzne wykrywanie zrzucania i opóźnień opryszczek bydła 1 i 5 przy użyciu zagnieżdżonej reakcji łańcuchowej polimerazy. J. Vet. Diagn. Inwestuj. 9, 387–394. doi: 10.1177/104063879700900408
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., and tikunova, N. V. (2014). Analiza rekombinacji oparta na kompletnych genomach HAstV-2 i hastv-4. Zarazić. Genet. Evol. 22, 94–102. doi: 10.1016 / j.meegid.2014.01.010
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V., and tikunova, N. V. (2012). Wysoki poziom ewolucyjny ludzkiego astrowirusa. Zarazić. Genet. Evol. 12, 435–442. doi: 10.1016 / j.meegid.2012.01.019
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Blomström, A. L., Widén, F., Hammer, A. S., Belák, S., and Berg, M. (2010). Wykrycie nowego astrowirusa w tkance mózgowej norek cierpiących na zespół drżenia norek za pomocą metagenomiki wirusowej. J. Clin. Mikrobiol. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
PubMed Streszczenie / CrossRef Pełny Tekst / Google Scholar
Boros, Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et al. (2017). Ogniska neuroinwazyjnego astrowirusa związanego z zapaleniem mózgu i rdzenia, osłabieniem i paraliżem u odsadzonych od maciory świń, Węgry. Emerg. Zarazić. Dis. 23, 1982–1993. doi: 10.3201 | Eid2312. 170804
PubMed Abstract / CrossRef Full Text / Google Scholar
Boujon, C. L., Koch, M. C.,Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Wskazanie przeniesienia międzygatunkowego astrowirusa związanego z zapaleniem mózgu u owiec i bydła. Emerg. Zarazić. Dis. 23, 1604–1608. doi: 10.3201 / eid2309.170168
Streszczenie PubMed / Pełny tekst CrossRef / Google Scholar
Buzalas I. G., Vyutrich D., Selimovich-Hamza S., Dregemüller S., Bruggmann R. i Seuberlich T. (2016). Full-genome based molecular characterization of encephalitis-associated byka Astro viruses. Infect. Genet. Evol. 44, 162-168. doi: 10.1016 / j..2016.06.052
Streszczenie PubMed / Pełny tekst CrossRef / Google Scholar
Buzalas,I. G., Vütrich, D., Walland, J., Dregemüller, K., Zurbriggen, A., Vandevelde, M. i in. (2014). Astrovirus neurotropowy u bydła z niesupuratacyjnym zapaleniem mózgu w Europie. J. Clin. Mikrobiol. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Cantile, C., and Youssef, S. (2016). „Nervous System,” in Jubb, Kennedy, and Palmer ’ s Pathology of Domestic Animals, 6th Edn, ed. M. G. Maxie (Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T., and Meylan, M. (2017). Neurologiczne objawy kliniczne u bydła z towarzyszącym astrowirusowi zapaleniem mózgu. J. Vet. Stażystka. Med. 31, 1209–1214. doi: 10.1111 / jvim.14728
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Donato, C., and Vijaykrishna, D. (2017). Szeroki zakres żywicieli i różnorodność genetyczna astrowirusów ssaków i ptaków. Wirusy 9: E102. doi: 10.3390 / v9050102
PubMed Abstract / CrossRef Full Text / Google Scholar
Drummond, A. J., Suchard, M. A., Xie, D., and Rambaut, A. (2012). Bayesian filogenetics with BEAUti and the BEAST 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev / mss075
PubMed Abstract / CrossRef Full Text / Google Scholar
Giannitti, F., Anderson, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Chlamydia pecorum: zmiany płodu i łożyska w sporadycznych przypadkach poronień u kóz. J. Vet. Diagn. Inwestuj. 28, 184–189. doi: 10.1177/1040638715625729
PubMed Abstract / CrossRef Pełny tekst / Google Scholar
Hirashima, Y., Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Analiza całego genomu nowego neurotropowego astrowirusa bydlęcego wykrytego u japońskiego czarnego wołu z nie-ropnym zapaleniem mózgu i rdzenia w Japonii. Arch. Virol. 163, 2805–2810. doi: 10.1007 / s00705-018-3898-3
PubMed Abstract / CrossRef Pełny tekst / Google Scholar
International Committee on Taxonomy of Viruses (2018). Master Species List (MSL32), Version 1. Dostępne pod adresem: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185 (dostęp: Marzec 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G., and Holmes, E. C. (2002). Tempo ewolucji molekularnej wirusów RNA: ilościowa analiza filogenetyczna. J. Mol. Evol. 54, 156–165. doi: 10.1007 / s00239-001-0064-3
PubMed Abstract / CrossRef Pełny tekst / Google Scholar
Kumar, S., Stecher, G., and Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis Wersja 7.0 dla większych zbiorów danych. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093/molbev / msw054
PubMed Abstract / CrossRef Full Text / Google Scholar
Li, L., Diab, S., McGraw, S., Barr, B., Traslavina, R., Higgins, R., et al. (2013). Rozbieżne astrowirusy związane z chorobą neurologiczną u bydła. Emerg. Zarazić. Dis. 19, 1385–1392. doi: 10.3201 / eid1909.130682
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Lole, K. S., Bollinger, R. C., Paranjape, R. S., Gadkari, D., Kulkarni, S. S., Novak, N. G., et al. (1999). Pełnowymiarowe genomy ludzkiego wirusa niedoboru odporności typu 1 z podtypu C-zakażonych serokonwerterów w Indiach, z dowodami rekombinacji intersubtyp. J. Virol. 73, 152–160.
PubMed Abstract / Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et al. (2015). Diagnostyka neuroinwazyjnego zakażenia astrowirusem u osoby dorosłej z obniżoną odpornością z zapaleniem mózgu za pomocą bezstronnego sekwencjonowania nowej generacji. Clin. Zarazić. Dis. 60, 919–923. doi: 10.1093/cid / ciu912
PubMed Abstract / CrossRef Full Text / Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Patologia i immunohistochemiczne odkrycia infekcji wirusem Zachodniego Nilu u psittaciformes. Weterynarz. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Nowy astrovirus związany z zapaleniem mózgu i ganglioneuritis u owiec domowych. Transbound. Emerg. Dis. 64, 677–682. doi: 10.1111 / tbed.12623
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Astrowirusowe zapalenie mózgu u chłopca z agammaglobulinemią związaną z X. Emerg. Zarazić. Dis. 16, 918–925. doi: 10.3201 / eid1606.091536
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Rambaut, A., Lam, T. T., Max Carvalho, L., and Pybus, O. G. (2016). Badanie struktury czasowej sekwencji heterochronicznych przy użyciu TempEst (dawniej Path-O-Gen). Virus Evol. 2: vew007 doi: 10.1093/ve / vew007
PubMed Abstract / CrossRef Full Text / Google Scholar
Reuter, G., Pankovics, P., and Boros, A. (2018). Nonsuppurative (aseptyczne) meningoencephalomyelitis związane z infekcjami neurovirulent astrovirus u ludzi i zwierząt. Clin. Mikrobiol. Rev. 31, e00040-18. doi: 10.1128 / CMR.00040-18
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Beer, M., et al. (2016). Wykrycie nowego astrowirusa bydlęcego u krowy z zapaleniem mózgu. Transbound. Emerg. Dis. 63, 253–259. doi: 10.1111 / tbed.12493
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A., and Seuberlich, T. (2017a). Częstość i patologiczny fenotyp zakażenia astrowirusem ch13/NeuroS1 bydła u bydła z chorobami neurologicznymi: w kierunku oceny związku przyczynowego. Wirusy 9: E12. doi: 10.3390 / v9010012
PubMed Abstract / CrossRef Full Text / Google Scholar
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., and Seuberlich, T. (2017b). Zakażenie astrowirusem bydła u bydła z chorobą neurologiczną w zachodniej Kanadzie. Może. Weterynarz. J. 58, 601-603.
Google Scholar
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., and Seuberlich, T. (2016). Detection of astrovirus in historical cases of European sporadyc bovine encephalitis, Switzerland 1958-1976. Przód. Weterynarz. Sci. 3:91. doi: 10.3389 / fvets.2016.00091
PubMed Streszczenie / CrossRef Pełny Tekst / Google Scholar
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identyfikacja drugiego astrowirusa związanego z zapaleniem mózgu u bydła. Emerg. Zarazić Drobnoustroje. 5: e5. doi: 10.1038 / emi.2016.5
PubMed Streszczenie / CrossRef Pełny tekst / Google Scholar
Spinato, M. T., Vince, A., Cai, H., and Ojkic, D. (2017). Identyfikacja astrowirusa bydła w przypadkach bydlęcego nieuleczalnego zapalenia mózgu we wschodniej Kanadzie. Może. Weterynarz. J. 58, 607-609.
Google Scholar
(2010) Badanie immunohistochemiczne wirusa wścieklizny w obrębie ośrodkowego układu nerwowego gatunków domowych i dzikich zwierząt. Weterynarz. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
PubMed Abstract / CrossRef Pełny tekst / Google Scholar
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A., and Minh, B. Q. (2016). W-IQ-TREE: szybkie narzędzie filogenetyczne online do analizy maksymalnego prawdopodobieństwa. Kwasy Nukleinowe Res. 44, W232–W235. doi: 10.1093/nar / gkw256
PubMed Abstract / CrossRef Full Text / Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Ponowne odkrycie i charakterystyka genomiczna astrowirusów bydlęcych. J. Gen. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0.030817-0
PubMed Abstract / CrossRef Full Text / Google Scholar