Biologia para Cursos I
Resultados da Aprendizagem
- Entender a diferença entre o pré-mRNA mRNA e
Após a transcrição, eucarióticas pré-mRNAs devem ser submetidos a várias etapas de processamento antes que eles possam ser traduzido. Os eucarióticos (e procarióticos) tRNAs e rRNAs também são submetidos a processamento antes de poderem funcionar como componentes na máquina de síntese de proteínas.
processamento de mRNA
o pré-mRNA eucariótico sofre um processamento extenso antes de estar pronto para ser traduzido. Os passos adicionais envolvidos na maturação do mRNA eucariótico criam uma molécula com uma semi-vida muito mais longa do que um mRNA procariótico. O mRNAs eucariótico dura várias horas, enquanto o típico mRNA E. coli dura não mais de cinco segundos.
pré-mRNAs são primeiro revestidos em proteínas estabilizadoras do ARN; estas protegem o pré-mRNA da degradação enquanto é processado e exportado para fora do núcleo. As três etapas mais importantes do processamento pré-mRNA são a adição de fatores estabilizadores e sinalizadores nas extremidades 5′ e 3′ da molécula, e a remoção de sequências intermediárias que não especificam os aminoácidos apropriados. Em casos raros, a transcrição do mRNA pode ser “editada” depois de transcrita.
5′ tapping
While the pre-mRNA is still being synthesized, a 7-methylguanosine Caps is added to the 5′ end of the growing transcript by a phosphate linkage. Esta fracção (grupo funcional) protege o ARNm nascente da degradação. Além disso, fatores envolvidos na síntese de proteínas reconhecem a tampa para ajudar a iniciar a tradução por ribossomas.
3′ Poly-A Tail
Once elongation is complete, the pre-mRNA is clived by an endonuclease between an AAUAAA consensus sequence and a GU-rich sequence, leaving the AAUAAA sequence on the pre-mRNA. Uma enzima chamada poly – a polimerase então adiciona uma cadeia de aproximadamente 200 a resíduos, chamada Poly-a tail. Esta modificação protege ainda mais o pré-ARNm da degradação e sinaliza a exportação dos fatores celulares que a transcrição precisa para o citoplasma.
Pré-mRNA Splicing
Eucarióticas genes são compostos de exões, que correspondem aos codificadores de proteínas de sequências (ex-no significa que eles estão expressas), e intervir seqüências de chamadas de íntrons (intron denota o seu papel intervencionista), que podem estar envolvidos na regulação de genes, mas são removidos do pré-mRNA durante o processamento. Sequências de Intron em ARNm não codificam proteínas funcionais.
a descoberta dos intrões veio como uma surpresa para os pesquisadores na década de 1970 que esperavam que o pré-mRNAs iria especificar sequências de proteínas sem mais processamento, como eles tinham observado em procariontes. Os genes dos eucariotas superiores contêm muitas vezes um ou mais intrões. Estas regiões podem corresponder a sequências reguladoras; no entanto, o significado biológico de ter muitos intrões ou ter intrões muito longos em um gene não é claro. É possível que os intrões abranjam a expressão genética porque demora mais tempo a transcrever pré-mRNAs com muitos intrões. Alternativamente, os intrões podem ser remanescentes de sequência não-funcional deixados da fusão de genes antigos ao longo da evolução. Isto é suportado pelo fato de que exons separados muitas vezes codificam subunidades proteicas separadas ou domínios. Na maior parte das vezes, as sequências de intrões podem sofrer mutações sem, Em última análise, afectar o produto proteico.Todos os intrões de um pré-ARNm devem ser completa e precisamente removidos antes da síntese proteica. Se o processo errar por um único nucleótido, o quadro de leitura dos exons reunidos mudaria, e a proteína resultante seria disfuncional. O processo de remoção de intrões e reconectação de exões é chamado splicing (Figura 1). Os intrões são removidos e degradados enquanto o pré-ARNm ainda está no núcleo. O Splicing ocorre por um mecanismo específico de sequência que garante que os intrões serão removidos e os exons reunidos com a precisão e precisão de um único nucleótido. O splicing de pré-mRNAs é conduzido por complexos de proteínas e moléculas de RNA chamadas spliceossomas.
a pergunta prática
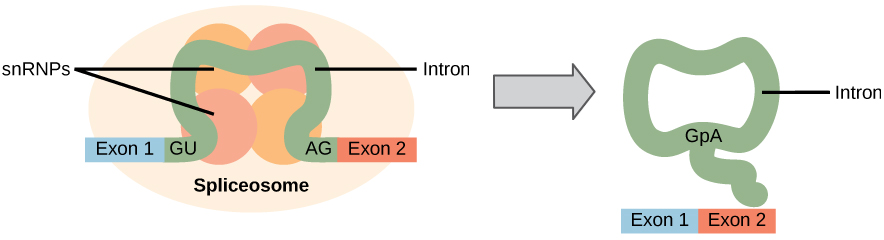
Figura 1. O splicing pré-mRNA envolve a remoção precisa dos intrões da transcrição primária de RNA. O processo de splicing é catalizado por complexos proteicos chamados spliceossomas que são compostos de proteínas e moléculas de RNA chamadas snRNAs. Os spliceossomas reconhecem sequências na extremidade 5′ e 3′ do intrão.
os erros na articulação estão implicados em cancros e outras doenças humanas. Que tipo de mutações podem levar a erros de splicing?
Note que mais de 70 intrões individuais podem estar presentes, e cada um tem que passar pelo processo de splicing—além de 5′ capping e a adição de um poli-uma cauda—apenas para gerar uma única molécula de mRNA translatável.
edição de ARN em tripanossomas
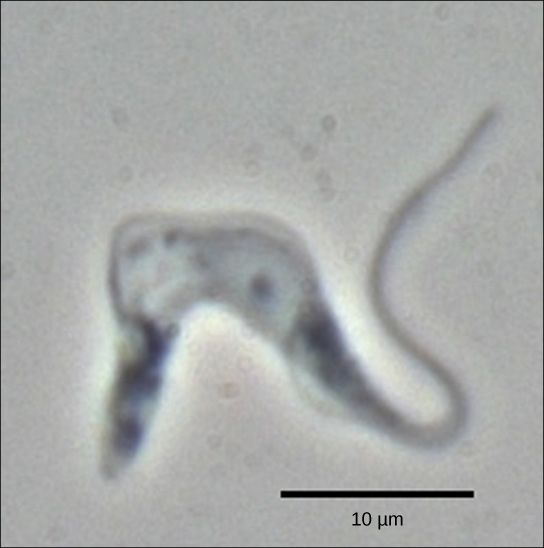
Figura 2. Trypanosoma brucei é o agente causador da doença do sono em humanos. O mRNAs deste patógeno deve ser modificado pela adição de nucleótidos antes que a síntese proteica possa ocorrer. (crédito: Alteração do trabalho de Torsten Ochsenreiter)
os tripanossomas são um grupo de protozoários que incluem o agente patogénico Trypanosoma brucei, que causa doença do sono em seres humanos (Figura 2). Tripanossomas, e praticamente todos os outros eucariontes, têm organelas chamadas mitocôndrias que fornecem a célula com energia química. As mitocôndrias são organelas que expressam seu próprio DNA e acredita-se que sejam remanescentes de uma relação simbiótica entre um eucariota e um procariota engolfado. O ADN mitocondrial dos tripanossomas apresenta uma excepção interessante ao Dogma Central: os seus pré-mRNAs não têm a informação correcta para especificar uma proteína funcional. Normalmente, isto é porque o ARNm está faltando vários nucleótidos U. A célula executa um passo adicional de processamento de RNA chamado edição de RNA para remediar isso.
outros genes do genoma mitocondrial codificam o guia RNAs de 40 a 80 nucleótidos. Uma ou mais destas moléculas interagem por pares de bases complementares com alguns dos nucleótidos na transcrição pré-mRNA. No entanto, o ARN-guia tem mais nucleótidos do que o pré-ARNm tem nucleótidos U com os quais se ligar. Nestas regiões, o ARN-guia desloca-se. As extremidades 3′ do Guia RNAs têm uma longa cauda poli-U, e estas bases U são inseridas em regiões da transcrição pré-mRNA em que o guia RNAs é looped. Este processo é inteiramente mediado por moléculas de RNA. Isto é, o guia RNAs—ao invés de proteínas-serve como catalisadores na edição de RNA.
a edição de ARN não é apenas um fenómeno de tripanossomas. Na mitocôndria de algumas plantas, quase todas as pré-mRNAs são editadas. A edição de RNA também foi identificada em mamíferos como ratos, coelhos e até mesmo humanos. Qual poderia ser a razão evolutiva para este passo adicional no processamento pré-mRNA? Uma possibilidade é que as mitocôndrias, sendo remanescentes de procariontes antigos, têm um método igualmente antigo baseado em RNA para regular a expressão genética. Em apoio a esta hipótese, as edições feitas para pré-mRNAs diferem dependendo das condições celulares. Embora especulativo, o processo de edição de RNA pode ser um adiamento de um tempo primordial quando moléculas de RNA, em vez de proteínas, foram responsáveis por catalisar reações.
Tente Isso
Contribuir!Teve alguma ideia para melhorar este conteúdo? Adoraríamos a tua opinião.
Melhorar este pageLearn Mais