Biologia para Cursos II
Resultados da Aprendizagem
- Entender o passado e emergentes de sistemas de classificação de vírus
Porque a maioria dos vírus, provavelmente, evoluíram a partir de ancestrais diferentes, a sistemática de métodos que os cientistas têm utilizado para classificar os procariontes e células eucarióticas não são muito úteis. Se os vírus representam” restos ” de organismos diferentes, então mesmo a análise genômica ou proteína não é útil. Por quê?, Porque os vírus não têm uma sequência genômica comum que todos compartilham. Por exemplo, a sequência rRNA 16S tão útil para a construção de filogenias procariotas é inútil para uma criatura sem ribossomas! Biólogos têm usado vários sistemas de classificação no passado. Os vírus foram inicialmente agrupados por morfologia compartilhada. Mais tarde, grupos de vírus foram classificados pelo tipo de ácido nucleico que continham, DNA ou RNA, e se seu ácido nucleico era de cadeia simples ou dupla. No entanto, estes métodos de classificação anteriores agruparam vírus de forma diferente, porque eles foram baseados em diferentes conjuntos de caracteres do vírus. O método de classificação mais comumente usado hoje é chamado de esquema de classificação de Baltimore, e é baseado em como o ARN mensageiro (mRNA) é gerado em cada tipo particular de vírus.
os sistemas anteriores de classificação
os vírus contêm apenas alguns elementos pelos quais podem ser classificados: o genoma viral, o tipo de capsid, e a estrutura envolvente para os vírus com envelope. Todos estes elementos foram utilizados no passado para a classificação viral (Tabela 1 e Figura 1). Genomas virais podem variar no tipo de material genético (DNA ou RNA) e sua organização (cadeia simples ou dupla, linear ou circular, e segmentada ou não segmentada). Em alguns vírus, proteínas adicionais necessárias para a replicação estão diretamente associadas ao genoma ou contidas no capsídeo viral.
| Quadro 1. Vírus de Classificação por Estrutura do Genoma e do Núcleo de | |
|---|---|
| Núcleo Classificações | Exemplos |
| RNA | o vírus da Raiva, os retrovírus |
| DNA | Herpesviruses, vírus da varíola |
| Single-stranded | o vírus da Raiva, os retrovírus |
| Double-stranded | Herpesviruses, vírus da varíola |
| Linear | o vírus da Raiva, os retrovírus, herpesviruses, vírus da varíola |
| Circular | Papilomavírus, muitos bacteriófagos |
| Não-segmentado: genoma consiste de um único segmento de material genético | Parainfluenza vírus |
| Segmentada: o genoma é dividida em vários segmentos | o vírus da Influenza |
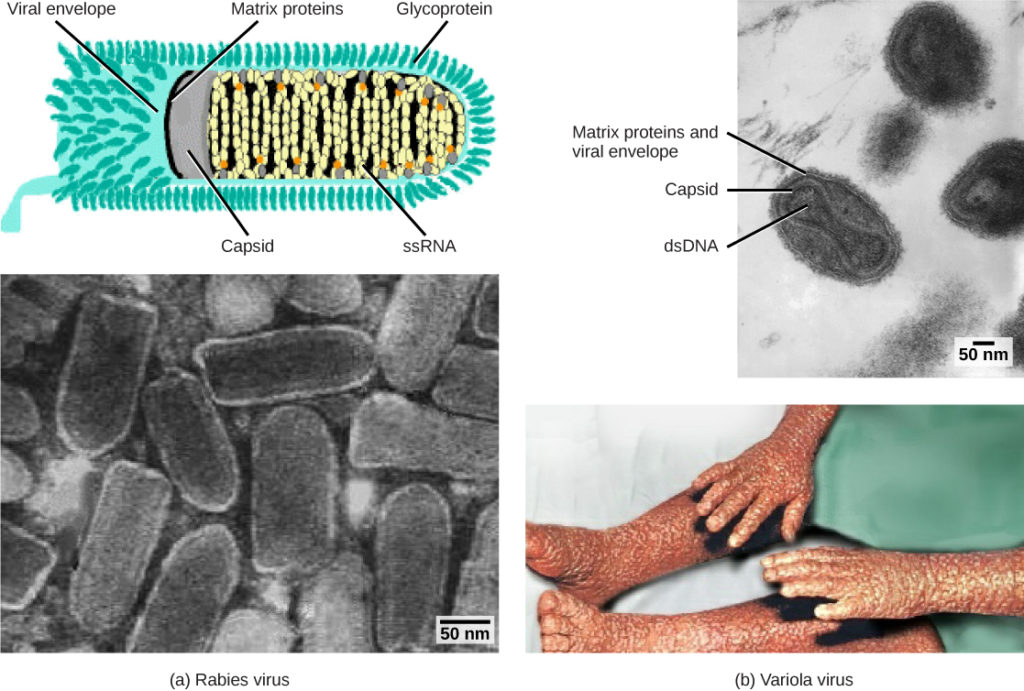
Figura 1. Os vírus são classificados com base no seu material genético e desenho capsid. a) o vírus da raiva tem um núcleo Rna de cadeia simples (ssRNA) e um capsídeo helicoidal envolvido, enquanto b) o vírus da varíola, o agente causador da varíola, tem um núcleo de ADN de cadeia dupla (dsDNA) e um capsídeo complexo. (credit “rabies diagram”: modification of work by CDC; “rabies micrograph”: modification of work by Dr. Fred Murphy, CDC; credit “small pox micrograph”: modification of work by Dr. Fred Murphy, Sylvia Whitfield, CDC; credit “smallpox photo”: modification of work by CDC; dados de barras de escala de Matt Russell)
os vírus também podem ser classificados pela concepção dos seus capsídeos (Quadro 2 e Figura 2). Os capsídeos são classificados como icosaédricos nus, icosaédricos Envelopados, céticos, céticos nus e complexos. O tipo de material genético (ADN ou ARN) e a sua estrutura (de cadeia simples ou dupla, linear ou circular, segmentada ou não segmentada) são utilizados para classificar as estruturas do núcleo do vírus (Quadro 2).
| Quadro 2. Vírus de Classificação por Cápside Estrutura | |
|---|---|
| Cápside Classificação | Exemplos |
| Nu icosahedral | o vírus da Hepatite A, polioviruses |
| Envolto icosahedral | vírus de Epstein-Barr, vírus do herpes simples, vírus da rubéola, vírus da febre amarela, o HIV-1 |
| Envolto helicoidal | o vírus da Influenza, o vírus da caxumba, vírus do sarampo, vírus da raiva |
| Nu helicoidal | vírus do mosaico do Tabaco |
| Complexo, com muitas proteínas; alguns possuem combinações de icosahedral e helicoidal cápside estruturas | Herpesviruses, vírus da varíola, da hepatite B e vírus bacteriófago T4 |

Figura 2. Micrografos de elétrons de transmissão de vários vírus mostram suas estruturas. A cápside do (a) vírus da poliomielite está nu icosahedral; (b) o vírus de Epstein-Barr cápside é envolvido icosahedral; (c) o vírus da caxumba cápside é um envolto de hélice; (d) o vírus do mosaico do tabaco cápside é nu helicoidal; e (e) o herpesvirus cápside é complexo. (credit a: modification of work by Dr. Fred Murphy, Sylvia Whitfield; credit b: modification of work by Liza Gross; credit c: modification of work by Dr. F. A. Murphy, CDC; credit d: modification of work by USDA ARS; credit e: modificação de trabalho por Linda Stannard, Departamento de Microbiologia Médica da Universidade da Cidade do Cabo, África do Sul, a NASA; escala-barra de dados a partir de Matt Russell)
Baltimore Classificação
O sistema mais utilizado de vírus classificação foi desenvolvida pelo ganhador do Prêmio Nobel biólogo David Baltimore, no início da década de 1970. Além das diferenças na morfologia e genética mencionado acima, o Baltimore esquema de classificação de grupos de vírus de acordo com a forma como o mRNA é produzido durante o ciclo de replicação do vírus.
os vírus do Grupo I contêm ADN de cadeia dupla (dsDNA) como genoma. O ARNm é produzido pela transcrição da mesma forma que o ADN celular.
os vírus do Grupo II têm DNA de cadeia simples (ssDNA) como seu genoma. Eles convertem seus genomas de cadeia simples em um intermediário dsDNA antes que a transcrição para mRNA possa ocorrer.
os vírus do Grupo III usam dsRNA como seu genoma. As cadeias se separam, e uma delas é usada como um modelo para a geração de ARNm usando a RNA-dependente polimerase codificada pelo vírus.
os vírus do Grupo IV têm ssRNA como seu genoma com uma polaridade positiva. Polaridade positiva significa que o ARN genômico pode servir diretamente como ARNm. Intermediários de dsRNA, chamados intermediários replicativos, são feitos no processo de cópia do RNA genômico. Múltiplas cadeias de RNA de comprimento completo de polaridade negativa (complementar ao RNA genômico de cadeia positiva) são formadas a partir destes intermediários, que podem então servir como modelos para a produção de RNA com polaridade positiva, incluindo tanto RNA genômico de comprimento completo e RNNs virais mais curtos.
os vírus do Grupo V contêm genomas ssRNA com uma polaridade negativa, o que significa que a sua sequência é complementar ao ARNm. Tal como acontece com os vírus do Grupo IV, os intermediários dsRNA são usados para fazer cópias do genoma e produzir mRNA. Neste caso, o genoma encalhado negativo pode ser convertido diretamente em ARNm. Além disso, cadeias de RNA positivas de comprimento total são feitas para servir como modelos para a produção do genoma de cadeia negativa.
os vírus do grupo VI têm genomas diplóides (duas cópias) ssRNA que devem ser convertidos, utilizando a enzima transcriptase reversa, para dsDNA .; o dsDNA é então transportado para o núcleo da célula hospedeira e inserido no genoma do hospedeiro. Em seguida, o ARNm pode ser produzido por transcrição do DNA viral que foi integrado no genoma do hospedeiro.
os vírus do grupo VII têm genomas dsDNA parciais e fazem intermediários ssRNA que atuam como mRNA, mas também são convertidos de volta em genomas dsDNA pela transcriptase reversa, necessária para a replicação do genoma. As características de cada grupo na classificação de Baltimore são resumidas no quadro 3 com exemplos de cada grupo.
| Quadro 3. Baltimore Classificação | |||
|---|---|---|---|
| Grupo | Características | Modo de mRNA de Produção | Exemplo |
| I | Double-stranded DNA | mRNA é transcrito diretamente a partir do molde de DNA | Herpes simplex (herpesvirus) |
| II | Single-stranded DNA | DNA é convertido para double-stranded formulário antes de o RNA é transcrito | parvovírus Canino (parvovírus) |
| III | Double-stranded RNA | mRNA é transcrito a partir de o genoma de RNA | Infância gastroenterite (rotavírus) |
| IV | Single stranded RNA (+) | Genoma funciona como mRNA | resfriado Comum (pircornavirus) |
| V | Single stranded RNA (−) | mRNA é transcrito a partir do genoma de RNA | Raiva (rhabdovirus) |
| VI | Single stranded RNA vírus com transcriptase reversa | transcriptase Reversa faz de DNA do genoma de RNA; o DNA é então incorporada no genoma de host; o mRNA é transcrito a partir da incorporação de DNA | vírus da imunodeficiência Humana (HIV) |
| VII | Double stranded DNA do vírus com transcriptase reversa | O genoma viral é double-stranded DNA, mas o DNA viral é replicado através de um RNA intermediário; o RNA pode servir diretamente como mRNA ou como um modelo para fazer mRNA | o vírus da Hepatite B (hepadnavirus) |