Creative Proteomics Blog
the sequence of amino acids in a protein or peptide can be identified by Edman degradation, which was developed by Pehr Edman. Este método pode rotular e clivar o peptídeo a partir de N-terminal sem perturbar as ligações peptídicas entre outros resíduos de aminoácidos. A reação de degradação de Edman foi automatizada em 1967 por Edman e Beggs. Atualmente, a degradação automatizada de Edman (o sequenador de proteínas) é amplamente utilizada, e pode sequenciar peptídeos até 50 aminoácidos.
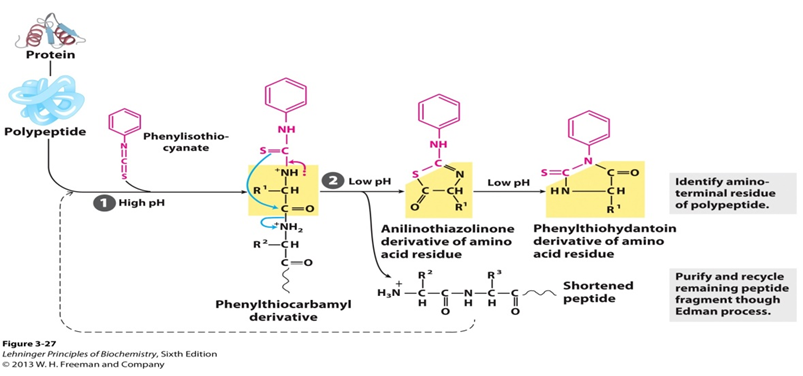
Cíclica degradação de peptídeos com base na reação de phenylisothiocyanate com o grupo amino livre do N-terminal de resíduos, tais que os aminoácidos são removidas uma a uma, identificados como seus phenylthiohydantoin derivados. Falando do processo específico, um peptídeo não carregado é reagido com fenilisotiocianato (PITC) no amino terminal em condições ligeiramente alcalinas para dar um derivado feniltiocarbamoílo (peptídeo PTC). Em seguida, sob condições ácidas, o enxofre tiocarbonil do derivado ataca o carbono carbonilo do aminoácido N-terminal. O primeiro aminoácido é clivado como derivado da anilinotiazolinona (ácido ATZ-amino) e o restante do peptídeo pode ser isolado e submetido ao ciclo de degradação seguinte. Uma vez formado, este derivado tiazolona é mais estável do que o derivado feniltiocarbamilo. O aminoácido ATZ é então removido por extração com acerato de etilo e convertido em um derivado da feniltiohidantoína (PTH-aminoácido). E a cromatografia pode ser usada para identificar o resíduo PTH gerado por cada ciclo.
quanto à degradação automatizada de Edman, as proteínas podem ser analisadas aplicando-as em solução em um filtro TFA e, em seguida, carregadas no instrumento de sequenciação Edman. As proteínas em misturas são primeiro separadas por géis 1D ou 2D e depois misturadas numa membrana PVDF. As proteínas são detectadas por coloração azul Coomassie, preto de Amido ou Poncau e as proteínas de interesse cortadas e a membrana PVDF carregada no sequenciador Edman.Com espectrometria de massa foi desenvolvido o uso de sequenciação de degradação de Edman começou a diminuir. No entanto, continua a ser o método para vários tipos de aplicações de análise estrutural de proteínas. Pode ser utilizado para verificar o limite N-terminal das proteínas recombinantes ou determinar o N-terminal de domínios resistentes à protease, particularmente quando a proteína ou domínio é >40 a 80 kDa ou não pode ser prontamente purificado. Também pode ser usado para identificar o novo local de clivagem N-terminal e proteolítica nos fragmentos proteicos. Além disso, quanto a algumas novas proteínas e peptídeos onde bases de dados de sequência não estão disponíveis para pesquisa de base de dados MS/MS, a degradação de Edman pode ser usada para análise.
o aminoácido N-terminal da proteína pode ser clivado. Assim, no processo, o primeiro ciclo identifica o aminoácido N-terminal exato. Além disso, uma vez que os aminoácidos libertados são identificados e quantificados por cromatografia, os aminoácidos com peso molecular idêntico podem ser identificados. Por exemplo, a isoleucina e a leucina têm uma massa de 113 Da, mas têm um tempo de retenção diferente. Além disso, a sequenciação de Edman pode ser realizada em blots de PVDF a partir de géis 1D e 2D, o que permite a sequenciação N-terminal de proteínas na mistura. No entanto, o sequenciamento de degradação de Edman não estará disponível quando o peptídeo cujo n-terminus foi modificado quimicamente, como a acetilação. E como o PITC não pode reagir com não-α-aminoácido, a sequenciação irá parar se um não-α-aminoácido for encontrado como ácido isoaspártico. Além disso, proteínas maiores não podem ser sequenciadas pela sequenciação de Edman.
na Creative Proteomics, a nossa plataforma profissional de sequenciamento de proteínas fornece análise de sequência N-terminal por degradação de Edman ou espectrometria de massa (MS), com vantagens complementares entre si. Nossa equipe de especialistas com vasta experiência pode ajudá-lo a entender o que você está tentando investigar e atender às suas necessidades. Além disso, podemos fornecer outros serviços de identificação de proteínas, incluindo:
- Peptide mass fingerprinting (PMF) de serviço
- análise da Sequência de peptídeos ou proteínas
- De novo De seqüência de proteína de serviço de análise
- Espingarda de proteína serviço de identificação
- Membrana proteômica serviço