Estrutura Molecular Revela Como o Antibiótico Estreptomicina Obras | BNL Newsroom
Estrutura Molecular Revela Como o Antibiótico Estreptomicina Obras
5 de setembro, 2013
Estreptomicina foi o primeiro antibiótico desenvolvido para tratar a tuberculose, contudo, até recentemente, os cientistas não entender completamente como funciona a nível molecular. Eles sabiam que a estreptomicina bloqueia um processo crítico, a síntese de proteínas por ribossomas levando à morte celular bacteriana, mas certos detalhes da interação permaneceram por descobrir. No Brookhaven National Laboratory’s National Synchrotron Light Source, pesquisadores têm usado cristalografia de raios-x para completar o quadro.
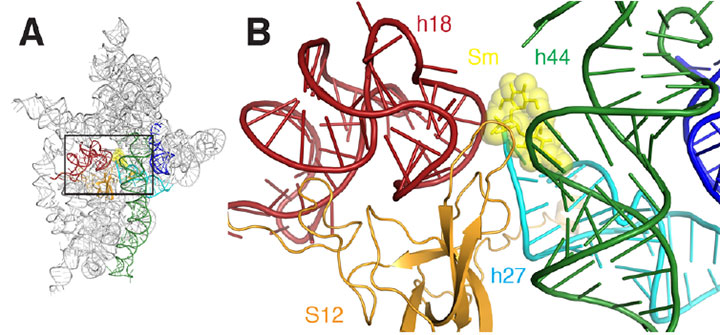
a) diagrama da fita do local de ligação da estreptomicina do ribossoma. B) Um close-up da área retangular delineada em A. Streptomicina é representado como sHcks amarelos e esferas, hélices são de cor vermelha, verde escuro, Ciano, Laranja e azul.
estreptomicina é um membro de uma família de antibióticos que trabalham interrompendo a função dos ribossomas das células bacterianas, as complexas máquinas moleculares que criam proteínas ligando aminoácidos juntos. Os ribossomas, um dos principais alvos para os antibióticos que funcionam inibindo a síntese de proteínas, têm duas partes principais ou “subunidades”.”
a subunidade maior faz a construção proteica, guiada por um tipo de RNA chamado ARN mensageiro (ARNm), que se liga a ele. The small subunit “reads” the mRNA and selects the matching transfer RNA (tRNA) molecule, which selects and delivers the next amino acid to the ribosome. É aqui que a estreptomicina desempenha um papel. Liga-se perto da pequena subunidade, fazendo com que interprete severamente a sequência. Isto resulta na síntese de proteínas aleatórias, que em última análise mata as bactérias. Mas como essa má leitura ocorreu permaneceu um mistério, até que um estudo recente por pesquisadores da Universidade Brown e da equipe multi-instituição Northeastern Collaborative Access em Argonne National Laboratory (gerenciado pela Universidade Cornell).
criando um cristal – uma disposição ordenada de unidades idênticas – da pequena subunidade ribossomal vinculado ao mRNA na presença de estreptomicina, os pesquisadores gerado várias informações detalhadas de “instantâneos”, que revelou-chave a nível molecular detalhes da interação, em última análise, mostrando como a estreptomicina prejudica a função da subunidade. Na NSLS beamline X25, eles usaram uma técnica chamada cristalografia de raios-x, na qual um feixe de raios-x é dirigido ao cristal, interage com as moléculas, e produz um padrão de difração intrincado. A partir do padrão, com a ajuda de software de computador, o grupo construiu representações visuais do complexo subunit-mRNA-streptomicina.
in short, the researchers could “see” for the first time the subunit’s decoding site, causing it to incorrectly read the mRNA. Por exemplo, a ligação da estreptomicina reduz a distância entre dois dos muitos hélices que compõem a estrutura molecular da subunidade. Isto é particularmente significativo porque estas hélices formam o local de decodificação real, e decodificação só ocorre corretamente se estes elementos são orientados exatamente para o mRNA e o tRNA selecionado. A ligação da estreptomicina também induz uma mudança na relação entre uma destas duas hélices e uma terceira hélice, fazendo com que uma se retraia da outra ou “desengate”.”
o resultado final de todas estas ligeiras alterações é que a estreptomicina desestabiliza a ligação entre a subunidade e o tRNA “correto”, enquanto simultaneamente estabiliza a ligação da subunidade ao tRNA “errado”, removendo assim efetivamente a discriminação entre o tRNA correto e o errado. Isto causa o caos na cadeia de abastecimento bacteriana de novas proteínas, perturbando a reprodução das bactérias e o ciclo de vida.
“Nossos estudos estruturais revelou que a estreptomicina induz surpreendentemente grandes distorções no ribossoma bacteriano, o que nos ajuda a entender como esse antibiótico interfere com a síntese de proteínas em bactérias”, disse o pesquisador principal Gerwald Jogl, professor associado de biologia na Brown biologia Molecular, Biologia Celular & Departamento de Bioquímica. “Continuando a partir de nossas descobertas atuais, estamos agora estudando como as mutações nos ribossomas bacterianos podem neutralizar esses rearranjos estruturais e permitir que as bactérias sobrevivam à ação letal da estreptomicina.”
esta pesquisa foi publicada na edição de 15 de janeiro de 2013 da Nature Communications, sob o título ” a structural basis for streptomicina-induced misreading of the genetic code.”O apoio veio dos Institutos Nacionais de Saúde e do Departamento de energia.
2013-4251 / INT / EXT / Newsroom