Fronteiras em Microbiologia
Introdução
O Astroviridae família contém não-envelopado, positivo-sentido único-stranded RNA vírus dentro de dois gêneros, Mamastrovirus e Avastrovirus, que infectam mamíferos e aves, respectivamente. Atualmente, o Comitê Internacional de Taxonomia de Vírus (Comitê Internacional de Taxonomia de Vírus , 2018) reconhece 19 espécies, nomeadamente Mamastrovirus-1 através de -19, dentro do Mamastrovirus gênero; no entanto, existem inúmeras variedades aguardando classificação, alguns dos quais estão provisoriamente considerado novas espécies (Donato e Vijaykrishna, 2017).
desde 2010, vários astrovírus têm sido cada vez mais reconhecidos como neuroinvasivos em várias espécies de mamíferos, incluindo humanos (Quan et al., 2010; Naccache et al., 2015), mink (Blomström et al., 2010), cattle (Li et al., 2013), sheep (Pfaff et al., 2017), e suínos (Boros et al., 2017). Após o reconhecimento inicial da encefalite associada ao astrovírus bovino nos Estados Unidos da América (Li et al., 2013), um estudo retrospectivo em casos de encefalite esporádica bovina de etiologia indeterminada da Suíça revelou que este astrovírus neuroinvasivo não tinha sido detectado por décadas (Selimovic-Hamza et al., 2016). Embora as vias de Epidemiologia e transmissão destes astrovírus sejam desconhecidas, a transmissão entre espécies foi sugerida com base no elevado nível de identidade (>98%), partilhado entre os astrovírus neuroinvasivos bovinos e ovinos nos níveis de nucleótidos e aminoácidos (Boujon et al., 2017).
Bovine astrovirus( Ostvs), named Ostv-NeuroS1 (Li et al., 2013) e Ostv-CH13 (Bouzalas et al., 2014), foram inicialmente encontrados no cérebro de bovinos com encefalite não supurativa nos Estados Unidos e Suíça, respectivamente. Apesar da nomenclatura diferente, ambos os vírus representam a mesma espécie de genótipo (Bouzalas et al., 2016; Selimovic-Hamza et al., 2017a) que ainda aguarda classificação oficial pela ICTV. Em 2015, uma estirpe anteriormente desconhecida, chamada “Ostv-CH15”, foi identificada no cérebro de vacas com encefalite na Suíça. A comparação filogenética do genoma completo revelou uma relação mais próxima do Ostv-CH15 com um astrovírus ovino (OvAstV) do que com o Ostv-CH13 (Seuberlich et al., 2016). A co-infecção com Ostv-CH13 e Ostv-CH15 também foi documentada num caso (Seuberlich et al., 2016). No mesmo ano na Alemanha, Schlottau et al. (2016) reported a novel astrovirus, namely BoAstV-BH89/14, in a cow with encephalitis, that was most closely related to OvAstV and BoAstV-CH15. Subsequentemente, o BoAstV-CH13 / NeuroS1 foi identificado em 2017 em casos de encefalite bovina no leste e oeste do Canadá (Spinato et al., 2017; Selimovic-Hamza et al., 2017b). Em 2018, um novo BoAstV neuroinvasivo intimamente relacionado com o BoAstV-NeuroS1/BoAstV-CH13 da América do Norte e Europa, foi identificado em um boi com encefalomielite não supurativa no Japão, e a ocorrência de recombinação intra-genotípica entre as estirpes norte-americanas e europeias foi sugerida (Hirashima et al., 2018).Embora tenham sido relatados casos de encefalite associada ao astrovírus na América do Norte, Europa e Ásia, sua presença nunca foi documentada no Hemisfério Sul. Aqui nós descrevemos um caso de astrovirus-associado da encefalite bovina no Uruguai, o que amplia a distribuição geográfica e diversidade genética de neuroinvasive astroviruses e fornecer filogeográficos evidência que sugere que este vírus foi introduzido nas Américas, da Europa e, mais tarde, se espalhou para a Ásia.
Materiais e Métodos
História e Reconhecimento
Em junho de 2018, de 22 meses de idade, Holstein dirigir um grupo de 37 bois em uma ∼300 hectares, a fazenda, em Colonia, Uruguai, desenvolvido progressiva neurológica canta, incluindo o comportamento incomum, andando sem rumo, andar em círculos, ataxia, repetitivas e descoordenadas os movimentos da língua, e decúbito. O rebanho pastou em uma pastagem anual de aveia e foi complementado com silagem de milho. Um diagnóstico clínico presumível de listeriose cerebral por parte do médico veterinário provocou o tratamento com penicilina e estreptomicina, no entanto o animal morreu espontaneamente após um ciclo clínico de 3 dias.
exame patológico, hibridização in situ (ISH) e imunohistoquímica (IHC)
a cabeça do boi foi removida da carcaça e submetida ao Laboratório Veterinário de diagnóstico INIA (plataforma de Saúde Animal) para trabalho de diagnóstico. Metade do cérebro, um curto segmento proximal cervical da medula espinhal (C1), gânglio trigeminal e raiz do nervo trigeminal, glândulas salivares, linfonodos retrofaríngeos, orofaringe, esôfago, a língua e o músculo-esquelético, foram imersão, fixo em 10% neutra tamponada de formalina por 48-72 h. Os tecidos foram processados rotineiramente para histologia, incorporado em parafina, micrótomo-seccionados em 4-5 µm e corados com hematoxilina e eosina (H&E) e manchas de Grama.
Cromogênico ISH foi realizada manualmente em 5 µm seções fixadas em formol, incluídas em parafina (FFPE) do tronco cerebral, cérebro e cerebelo em Superfrost Além de slides (Thermo Fisher Scientific, Pittsburgh, PA, Estados Unidos) usando o RNAscope 2.5 Vermelho kit de ensaio (Cat #322360, Advanced Cell Diagnóstico, Hayward, CA, Estados Unidos da américa) e o BoAstV sonda Gato. #406921. A sonda é composta por 20 pares de 20 ZZ, visando a região 5232-6180 do vírus (GenBank KF233994.1). Cada secção de tecido de 5 µm foi pré-tratada com calor e protease antes da hibridação por sonda durante 2 h a 40°C e processada de acordo com as recomendações do fabricante. Os controlos negativos utilizados para a validação do sinal incluíam uma sonda não relacionada (com o teor de GC combinado) que funcionava em secções seriais e a sondar tecidos de animais não infectados. Slides foram contrastados com hematoxilina e montados com EcoMount (Biocare Medical, Concord, CA, Estados Unidos).
adicionalmente, o IHC foi realizado em secções FFPE do tronco cerebral, cerebro e cerebelo, Como anteriormente descrito, para a identificação do vírus do Nilo Ocidental (WNV, Flavivirus) (Palmieri et al., 2011), raiva virus (Lyssavirus) (Stein et al., 2010), e Clamydia spp. (Giannitti et al., 2016) antigénios.
virologia Molecular
a extracção de ácido nucleico foi realizada a partir de uma amostra conjunta de cérebro congelado (-20°C) utilizando MagMAX Nucleic Acid Isolation Kit® (Thermo Fisher Scientific). Para a detecção de astrovírus, a transcrição reversa (RT) foi realizada com Transcriptase reversa reversa reversa reversa reversível® (Thermo Fisher Scientific) e primers hexamer aleatórios (Qiagen). A PCR foi realizada a partir do cDNA utilizando MangoMix® (Biolina) e primers que amplificam um fragmento de 432 nucleótidos do gene da polimerase do astrovírus (Tse et al., 2011). O produto PCR foi visualizado em gel de agarose a 2%, purificado utilizando o PureLink® Quick Gel Extraction e o PCR Purification Combo Kit (Invitrogen), e sequenciado na Macrogen Inc. (Seul, Coreia Do Sul). Para a amplificação de todo o genoma do astrovírus, o máximo H menos a Transcriptase reversa (Thermo Fisher Scientific)e o oligo(dT) 18 para a obtenção do cDNA, e o MangoMix® (Biolina) ou a polimerase do ADN Ranger (Biolina) com primers descritos por Hirashima et al., 2018, foram utilizados. Os produtos PCR foram visualizados em 1-2% de gel de agarose, purificado e sequenciado como mencionado acima. A montagem da sequência foi realizada com SeqMan (Lasergene 8, DNASTAR). Vinte e seis sequências completas de genoma de astrovírus neuroinvasivo de bovinos, ovinos, porcos, humanos e vison, e astrovírus bovino entérico disponíveis Em GenBank foram baixadas e alinhadas usando Clustal W em software MEGA 7 (Kumar et al., 2016). W-IQ-TREE1 (Trifinopoulos et al., 2016) foi usado para determinar o melhor modelo de evolução de sequência (SYM+I+G4) e para construir uma árvore filogenética de máxima probabilidade com as sequências quase completas do Ostv detectado neste caso, e essas sequências completas baixadas de GenBank, usando bootstrap como o método estatístico para avaliar a robustez de clades. Similarity plot foi realizado com SimPlot software (Lole et al., 1999). As distâncias P ao nível dos aminoácidos do ORF2 foram estimadas com o software MEGA 7 (Kumar et al., 2016).
adicionalmente, uma análise filogeográfica Bayesiana foi realizada com o pacote BEAST v1. 8. 4 (Drummond et al., 2012), usando: a completar a codificação de região do BoAstV CH13/NeuroS1 linhagem, ORF1ab (não-estruturais genes), ORF2 (estrutural do gene), ORF1a (protease) e uma parte de um ORF1b (polimerase genômica região, para o qual Canadense estirpes estavam disponíveis), com todas as seqüências disponíveis no GenBank (última acessão a 18 de abril de 2019), para determinar a taxa evolutiva, a idade/anos de ancestrais comuns, e a rota mais provável circulação viral por país (Suíça, Uruguai, Estados Unidos, Canadá e Japão). A falta de recombinação no conjunto de dados foi determinada utilizando o programa de detecção de recombinação 4. O modelo de substituição que melhor se encaixa em cada alinhamento foi determinado usando o software MEGA 7 através dos valores do critério de informação Bayesiana (BIC), e a estrutura temporal de cada conjunto de dados foi avaliada usando TempEst (Rambaut et al., 2016). O relógio molecular relaxado lognormal com análise do horizonte Bayesiano foi selecionado pelo fator Bayes entre as diferentes combinações de relógios moleculares e priores de árvore coalescentes usados. O país de detecção foi usado como característica. O comprimento da cadeia Markov Monte Carlo foi de 100 milhões de gerações, garantindo a convergência da análise, avaliada no Tracer v1. 6.0, e a probabilidade posterior foi usada para avaliar clades. The maximum clade credibility tree (MCCT) was obtained using TreeAnnotator software from BEAST and visualized in FigTree v1.4.3.Por último, o ADN extraído do cérebro congelado foi processado por PCR para detecção de herpesvírus bovino 1 e 5 (BHV-1 e -5), como descrito anteriormente (Ashbaugh et al., 1997).
Bacteriologia
Fresco amostras do cerebelo e do tronco cerebral foram processados rotineiramente para aeróbica, culturas bacterianas no sangue e MacConkey ágares, seletiva e cultura para Listeria monocytogenes (Al-Zoreky e Sandine, 1990).
Resultados e Discussão
Os sinais clínicos e achados epidemiológicos no caso aqui descrito, embora não específico, foram semelhantes aos descritos em outros casos, de bovinos astrovirus associadas a encefalite, que é geralmente descrito como esporádica (Selimovic-Hamza et al., 2016), com uma variedade de déficits neurológicos (Deiss et al., 2017), com uma duração de sinais clínicos que tipicamente varia de 1 dia a 3 semanas (Schlottau et al., 2016; Deiss et al., 2017; Spinato et al., 2017; Hirashima et al., 2018).O exame macroscópico do cérebro, o segmento C1 da medula espinhal e outros tecidos da cabeça não revelaram lesões anatômicas grosseiras significativas. Histologicamente, houve meningoencefalomielite moderada a grave, linfocítica, histiocítica e plasmacítica que afetou o telencéfalo (incluindo o hemisfério cerebral e hipocampo), o tronco cerebral e o único segmento examinado da medula espinhal. As lesões foram predominantemente distribuídas na matéria cinzenta e áreas limitantes da matéria branca. Nas áreas afectadas houve abafamento perivascular e inflamação linfoplasmacítica e histiocítica e necrose neuronal/neuronofagia com gliose no neuropilo adjacente. Houve satellitose de neurônios necróticos afetados (figuras 1A–D). As lesões foram muito menos frequentes e graves no parênquima cerebelar, embora tenha havido leptomeningite cerebelar moderada multifocal. Não foram encontradas bactérias intralesionais com manchas H&E E Gram. Não foram encontradas alterações histológicas significativas nos outros tecidos examinados.

Figura 1. Lesões histológicas no tronco cerebral (A,B) e no córtex cerebral (C,D) e detecção de RNA Ostv no córtex cerebral (e,G). Imagens (A-D) são secções do cérebro manchadas com H &E; imagens E E E G são seções do córtex cerebral demonstrando hibridização usando o ISH cromogênico usando a sonda específica do BoAstV, contrastada com hematoxilina; imagem F é uma seção serial da sonda DapB do córtex cerebral (controle negativo), contrastada com hematoxilina. (A) um espaço perivenular é acentuadamente expandido por células inflamatórias (principalmente linfócitos e histiócitos) que também se infiltram no neuropil adjacente. B) o neurónio do centro tem perikaryon hipereosinofílico e karyorrhexis (necrose) e o corpo neuronal está rodeado por um número crescente de células gliais (satellitose) e inflamatórias. C) a leptomeninge é infiltrada por linfócitos e histiócitos. D) uma região do córtex cerebral com múltiplos neurónios hipereosinófilos (necróticos) e um grande recipiente com algemas linfocíticas perivasculares. E) numa secção serial do córtex cerebral, a abundante rotulagem intracitoplasmática do RNA é representada por uma forte deposição de cromogénio vermelho granular dentro do citoplasma neuronal das extensões soma e neuronal (e, G), que não está presente com hibridização utilizando a sonda de controlo negativo (F).Após exame histológico do sistema nervoso central, suspeitou-se de uma infecção viral neuroinvasiva
uma infecção viral neuroinvasiva. Bovinos com encefalite são motivo de preocupação porque muitos neuropatógenos ruminantes são zoonóticos (Cantile e Youssef, 2016); assim, um diagnóstico de encefalite deve levar a testes laboratoriais extensos para rastrear agentes infecciosos, quando possível. No caso descrito no presente documento, IHCs para WNV, vírus da raiva e clamídia spp., e PCR para BHV-1 e -5 foram todos negativos, e nenhuma bactéria patogênica foi cultivada a partir do tecido cerebral. Como o boi tinha < 2 anos de idade e não foram observadas alterações espongiformes no tronco cerebral, o animal não foi testado para a encefalopatia espongiforme bovina (EEB), que é uma doença exótica de bovinos adultos que nunca foi relatada no Uruguai. Além disso, a EEB não é inflamatória (Cantile e Youssef, 2016).
a hibridização in situ foi realizada usando uma sonda gerada a partir de Ostv-Neuro1, e houve hibridização por sonda abundante dentro, e limitada ao citoplasma de neurônios no hemisfério cerebral e hipocampo (figuras 1E–G). Nestas áreas a hibridização da sonda colocalizada com neurônios necróticos e regiões da gliose, sem hibridização de sonda detectável nas células gliais, ou células inflamatórias das algemas perivasculares. Nenhum ácido nucleico viral foi detectado pela ISH no cerebelo, que teve apenas lesões inflamatórias mínimas no parênquima, mas leptomeningite moderada, ou no tronco cerebral, incluindo seções com inflamação grave. Isto significa que, topograficamente, a detecção da distribuição viral pela ISH foi mais limitada do que a encefalite nas secções examinadas, que tem sido ocasionalmente descrita em casos de encefalite associada a Ostv-CH13/Neuro1 em bovinos (Selimovic-Hamza et al., 2017a, b). Uma razão para esta ocasional falta de detecção de ARN viral em áreas lesionadas do cérebro pode ser o limite de detecção da ISH, ou a depuração do vírus em áreas inflamadas do cérebro até a hora da morte, como sugerido anteriormente (Selimovic-Hamza et al., 2017b). Como esperado, nenhuma hibridação de sonda foi detectada pela ISH no tecido cerebral usado como controle negativo.
astrovírus foi detectado no cérebro por RT-PCR. A análise quase completa da sequência do genoma revelou uma estirpe de Mamastrovírus dentro do clado CH13/NeuroS1, que nomeamos de Ostv-Neuro-Uy, a sequência foi depositada em GenBank sob o número de adesão MK386569. A análise filogenética revelou a proximidade com outros astrovirus neuroinvasivos dentro do clado Virginia/humano-Vison-Ovinos (VA/HMO) (Figura 2), que contém os astrovirus neuroinvasivos mais conhecidos (Hirashima et al., 2018; Reuter et al., 2018). A sequência quase completa de Ostv-Neuro-Uy tem 6427 bp de comprimento e tem uma identidade sequencial de 94% com a estirpe KagoshimaSR28-462. BoAstV-Neuro-Uy tem características semelhantes como outras cepas de linhagem CH13/NeuroS1: 5’UTR região de 51 nt, ORF1a (protease) de 861 aminoácidos (aa), ORF1b de 523 aa (RNA-dependente de RNA polimerase), e ORF2 de 758 aa (cápside proteica). Infelizmente, o 3’UTR não pôde ser sequenciado, mas presume-se que uma cauda poli(a) esteja presente porque oligo(dT)18 foi usado para obter cDNA. Além disso, a sequência heptamérica de AAAAAAC, um sinal de deslocamento de rameshift ribossomal, está presente. P-distances at the amino acid level of the ORF2 confirmed the assignation of this strain to the CH13 / NeuroS1 clade. P-distâncias < 0.35 entre BoAstV-Neuro-Uy e outros membros do clado (Tabela 1) para oferecer suporte a uma classificação destas cepas virais dentro de uma mesma espécie; Mamastrovirus-13 foi recentemente proposto por outros autores (Donato e Vijaykrishna, 2017; Hirashima et al., 2018), embora a atribuição definitiva de espécies pelo ICTV esteja pendente. A sonda usada para a ISH, gerada a partir de Ostv-Neuro1, tinha 92,7% de identidade de sequência com Ostv-Neuro-Uy.
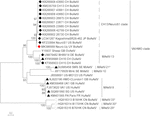
Figura 2. Phylogenetic analysis of full-length nucleotide sequences by maximum-likelihood method. Ostv-Neuro-Uy está marcado com um losango vermelho. Outras sequências de astrovirus neuroinvasivos bovinos estão marcadas com rombo preto, sequências de astrovirus neuroinvasivos não bovinos estão marcadas com triângulos negros e sequências de astrovirus bovinos entéricos estão marcadas com círculos brancos. As espécies de mamastrovírus são mostradas; os asteriscos indicam espécies ainda a serem reconhecidas pela ICTV. As estirpes do clado CH13 / NeuroS1 ainda não atribuídas a uma espécie pelo ICTV são identificadas com uma linha tracejada. Os valores de Bootstrap são indicados nos nós. CH, Suíça; JP, Japão; EUA, Estados Unidos da América; UY, Uruguai; GB, Reino Unido da Grã-Bretanha; de, Alemanha; SE, Suécia; FR, França; CN, China. Ostv, astrovírus bovino; OvAstV, astrovírus ovino; PoAstV, astrovírus porcino; HuAstV, astrovírus humano; MiAstV, astrovírus mink.
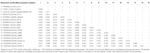
Quadro 1. Estimativas de divergência evolutiva no aminoácido nível de completa ORF2 região entre sequências de bovinos e ovinos Mamastrovirus-13 e tensões dentro da CH13/NeuroS1 clado ainda não atribuído a uma espécie pelo Comitê Internacional de Taxonomia de Vírus (ICTV).
estudos baseados em exames neuropatológicos e na detecção de ácidos nucleicos e proteínas do astrovírus concluíram que existe uma relação causal provável entre a infecção por astrovírus e a doença neurológica e lesões em bovinos (Selimovic-Hamza et al., 2017a; Reuter et al., 2018). Tanto quanto sabemos, a encefalite associada ao astrovírus ainda não foi reproduzida experimentalmente. Isso exigiria isolamento de astrovirus neuroinvasivos de casos clínicos, o que não foi tentado no nosso caso.
neste caso, não foi possível determinar a fonte de Uy-Neurologista. No entanto, o gado reservatório e a vida selvagem devem ser considerados, uma vez que o gado foi criado sob extensas condições ao ar livre. O animal afetado foi comprado e transferido para a fazenda em fevereiro de 2018, juntamente com outros 9 Bois. Infelizmente, o proprietário recusou mais amostras e testes de outros animais na propriedade, e uma investigação epidemiológica mais detalhada. Nenhum dos outros animais do grupo tinha desenvolvido doença neurológica a partir de agosto de 2018, a última vez que o médico veterinário foi contactado. Uma sazonalidade desde o início do inverno até o final da primavera foi sugerida para casos de encefalite associada ao astrovírus na Suíça (Selimovic-Hamza et al., 2016). Curiosamente, o caso aqui descrito ocorreu em junho, correspondendo ao período de transição outono-inverno no Hemisfério Sul.
enquanto astrovírus neurotrópicos foram identificados na América do Norte (Li et al., 2013; Spinato et al., 2017), Europa (Bouzalas et al., 2014) e Ásia (Hirashima et al., 2018), Sua presença nunca foi relatada no hemisfério sul, de modo que esta comunicação amplia a distribuição geográfica da encefalite associada ao astrovírus. Para avaliar se a estirpe viral detectada no Uruguai poderia ter se originado na Europa, América do Norte, ou Ásia, estimamos a taxa de evolução e realizamos uma análise filogeográfica usando sequências neurominvasivas de Van GenBank. A taxa de evolução estimada usando a região de codificação completa foi de 4.27 × 10-4 (95% maior densidade de probabilidade-HPD-, 2, 19–6, 46 × 10-4) substituições nucleotídicas/local/ano, o que é esperado para um vírus ARN (Jenkins et al., 2002), mas inferior ao estimado para os astrovírus humanos entéricos (Babkin et al., 2012, 2014). O ORF1ab região mostrou uma semelhantes evolutiva taxa de (4.20 × 10-4, 95% HPD 1.66–6.46 × 10-4 substituições/site/ano) a completar a codificação de região, enquanto o ORF1a (2.92 × 10-4, 95% HPD 1.19 × 10-6-6.46 × 10-4 substituições/site/ano) e ORF2 (2.86 × 10-4, 95% HPD 4.13 × 10-6-5.79 × 10-4 substituições / local / ano) mostraram uma taxa evolutiva ligeiramente mais rápida, e a região genômica da polimerase parcial (ORF1b) mostrou uma taxa evolutiva ligeiramente mais lenta (5.39 × 10-4, 95% HPD 6.41 × 10-7-1.10 × 10-3 substituições/local/ano).
como determinado pela análise filogeográfica com a região de codificação completa, e mostrado no MCTT (Figura 3), Existem duas sub-linhagens (CH13 e Neuro1) com base em estirpes de referência, que têm um ancestral comum. O ancestral comum mais recente destas sub-linhagens (linhagem CH13/Neuro1) surgiu na Europa aproximadamente em 1885 (95% HPD, 1794-1940). No início dos anos 1900, as duas sub-linhagens divergiram, a sub-linhagem CH13 permaneceu circulando na Europa, enquanto a sub-linhagem Neuro1 se espalhou para a América e Ásia. O cenário mais provável é que a sub-linhagem Neuro1 foi introduzida no Uruguai a partir da Europa por volta do ano 1921 (95% HPD, 1849-1967), presumivelmente através do Comércio de gado, em seguida, se espalhou para a América do Norte, e mais tarde para o Japão (Figura 3). Devido à limitação no número de seqüências disponíveis no GenBank, o que pode ter enviesado a análise, os resultados obtidos utilizando-se a completar a codificação de região foram comparados com aqueles obtidos com outras regiões genômicas (ORF1ab, ORF2, ORF1a, e ORF1b) disponíveis para um maior número de cepas (i.é., o Canadense estirpes). Em todas as análises, o cenário mais provável é que a introdução do vírus no Uruguai tenha ocorrido a partir da Europa (números suplementares S1A–D). Além disso, a data estimada para esta introdução, obtidos com o ORF1ab e parcial polimerase genômica região (ORF1b) (Números Suplementares S1A,D), foi semelhante à obtida com a completar a codificação de região, considerando que a data estimada de introdução obtidos com ORF2 e ORF1a foi anteriores, mas com mais de 95% HPD (intervalo de Números Suplementares S1B,C). Uma introdução da sub-linhagem NeuroS1 diretamente para o Canadá a partir da Europa, com subsequente propagação para os Estados Unidos e Japão, também é plausível, s mostrado na figura suplementar S1D.

Figura 3. Maximum clade credibility tree obtained by analysis of the full-length coding region. A cor dos ramos representa o país mais provável onde os antepassados circularam, valores de probabilidade posteriores são mostrados nos ramos e os números em cada nó representam os anos de origem para cada clado com o intervalo HPD de 95%. As sub-linhagens são indicadas com os rótulos.
são necessárias mais investigações para avaliar a distribuição geográfica, os mecanismos patogénicos (em especial os mecanismos de transmissão e entrada), a Epidemiologia molecular e a potencial transmissão interespécie de astrovírus neuroinvasivas.
disponibilidade de dados
os conjuntos de dados gerados para este estudo podem ser encontrados em GenBank, MK386569.
contribuições dos autores
FG, RDC, e MC contribuíram com a concepção do estudo. A FG e a RDC realizaram o exame patológico e a amostragem. A PP realizou a hibridização in situ. O FU realizou a imunohistoquímica. LM, RC e MC realizaram testes de virologia molecular. MC realizou a sequência e análises filogeográficas e figuras associadas. FG e PP obtiveram as imagens histológicas. MF realizou as culturas bacterianas. FG e MC escreveram o primeiro rascunho do manuscrito. RDC, PP, FU, LM, MF e RC escreveram seções do manuscrito. Todos os autores contribuíram para a revisão do manuscrito, leram e aprovaram a versão submetida.
Funding
This work was funded by Grants PL-015 N-15156 from INIA and 158 from the “Programa de Iniciação à investigação 2017” from “Comissão Sectorial de Investigação Científica” (CSIC). MC and RDC acknowledge support from The “Agência Nacional de pesquisa e inovação” (ANII) and INIA, respetivamente, através do Ph.D. scholarships. FG ackno supledges support from ANII through Mobility grant MOV_CA_2018_1_150021.
Declaração de conflito de interesses
os autores declaram que a investigação foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.
the reviewer TS declared a past co-authorship with one of the authors PP to the handling editor.
Agradecimentos
Os autores agradecem Yisell Perdomo e Cecilia Monesiglio do INIA, e Karen Sverlow e Juliann Beingesser de CAHFS para a assistência técnica.
Material Suplementar
O Material Suplementar para este artigo podem ser encontradas online em: https://www.frontiersin.org/articles/10.3389/fmicb.2019.01240/full#supplementary-material
FIGURA S1 | Máximo clado credibilidade árvores (MCCTs) obtidos através da análise do comprimento total ORF1ab (A), com a duração total de ORF2 (B), com a duração total de ORF1a (C), e parcial ORF1b (D). A cor dos ramos representa o país mais provável onde os ancestrais circularam, valores de probabilidade posteriores são mostrados nos ramos, e os números em cada nó representam os anos de origem para cada clado com o intervalo HPD de 95%. As sub-linhagens são indicadas com os rótulos.
notas de Rodapé
- ^http://iqtree.cibiv.univie.ac.at
Al-Zoreky, N., e Sandine, W. E. (1990). Meio altamente selectivo para o isolamento de Listeria monocytogenes dos alimentos. Appl. Ambiente. Microbiol. 56, 3154–3157.
PubMed Resumo | Google acadêmico
Ashbaugh, S. E., Thompson, K. E., Belknap, E. B., Schultheiss, P. C., Chowdhury, S., & Collins, J. K. (1997). Detecção específica da excreção e latência do herpesvírus bovino 1 e 5 utilizando uma reacção de polimerase aninhada em cadeia. J. Vet. Diagn. Investir. 9, 387–394. doi: 10.1177/104063879700900408
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Babkin, I. V., Tikunov, A. Y., Sedelnikova, D. A., Zhirakovskaia, E. V., e Tikunova, N. V. (2014). Análise de recombinação baseada nos genomas completos HAstV-2 e HastV-4. Infectar. Genet. Evol. 22, 94–102. doi: 10.1016 / j. meegid.2014.01.010
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Babkin, I. V., Tikunov, A. Y., Zhirakovskaia, E. V., Netesov, S. V., e Tikunova, N. V. (2012). Alta taxa evolucionária de astrovírus humano. Infectar. Genet. Evol. 12, 435–442. doi: 10.1016 / j. meegid.2012.01.019
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Blomström, A. L., Widén, F., Martelo, A. S., Belák, S., e Berg, M. (2010). Detecção de um novo astrovírus no tecido cerebral de vison que sofre de síndrome de vison agitada através da utilização de metagenómica viral. J. Clin. Microbiol. 48, 4392–4396. doi: 10.1128 / JCM.01040-10
PubMed Resumo | CrossRef Texto Completo | Google Acadêmico
Boros,Á, Albert, M., Pankovics, P., Bíró, H., Pesavento, P. A., Phan, T. G., et al. (2017). Surtos de astrovírus neuroinvasivo associados a encefalomielite, fraqueza e paralisia entre suínos desmamados, Hungria. Esmerg. Infectar. S. 23, 1982–1993. doi: 10.3201/eid2312.170804
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Boujon, C. L. Koch, M. C., Wüthrich, D., Werder, S., Jakupovic, D., Bruggmann, R., et al. (2017). Indicação da transmissão cruzada de espécies de astrovírus associado a encefalite em ovinos e bovinos. Esmerg. Infectar. S. 23, 1604–1608. doi: 10.3201 / eid2309.170168
PubMed Resumo / CrossRef Texto Completo / Google acadêmico
Bouzalas, I. G., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Bruggmann, R., e Seuberlich, T. (2016). Caracterização molecular completa baseada no genoma de astrovírus bovino associado a encefalite. Infectar. Genet. Evol. 44, 162-168. doi: 10.1016 / J. meegid.06/052/2016
PubMed Resumo / CrossRef Texto Completo / Google acadêmico
Bouzalas, I. G., Wüthrich, D., Walland, J., Drögemüller, C., Zurbriggen, A., Vandevelde, M., et al. (2014). Astrovírus neurotrópico em bovinos com encefalite não supurativa na Europa. J. Clin. Microbiol. 52, 3318–3324. doi: 10.1128 / JCM.01195-14
PubMed Abstract | CrossRef Full Text | Google Scholar
Cantile, C., and Youssef, S. (2016). “Nervous System,” in Jubb, Kennedy, and Palmer’s Pathology of Domestic Animals, 6th Edn, ed. M. G. Maxie( Saint Louis, MO: Elsevier), 205-406.
Google Scholar
Deiss, R., Selimovic-Hamza, S., Seuberlich, T., and Meylan, M. (2017). Sinais clínicos neurológicos em bovinos com encefalite associada ao astrovírus. J. Vet. Estagiario. Med. 31, 1209–1214. doi: 10.1111 / jvim.14728
PubMed Abstract / CrossRef Full Text | Google Scholar
Donato, C., and Vijaykrishna, D. (2017). A vasta gama de hospedeiros e a diversidade genética dos astrovírus mamífero e aviário. Vírus 9:E102. doi: 10.3390/v9050102
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Drummond, A. J., Suchard, M. A., Xie, D., e Rambaut, A. (2012). Filogenética Bayesiana com BEAUti e a besta 1.7. Mol. Biol. Evol. 29, 1969–1973. doi: 10.1093/molbev/mss075
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Giannitti, F., Almeida, M., Miller, M., Rowe, J., Sverlow, K., Vasquez, M., et al. (2016). Clamídia pecorum: lesões fetais e placentárias no aborto esporádico dos Caprinos. J. Vet. Diagn. Investir. 28, 184–189. doi: 10.1177/1040638715625729
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Hirashima, Y. Okada, D., Shibata, S., Yoshida, S., Fujisono, S., Omatsu, T., et al. (2018). Análise completa do genoma de um novo astrovírus bovino neurotrópico detectado num boi negro japonês com encefalomielite não supurativa no Japão. Arco. Virol. 163, 2805–2810. doi: 10.1007 / s00705-018-3898-3
PubMed Abstract | CrossRef texto completo | Google Scholar
International Committee on Taxonomy of Viruses (2018). Master Species List( MSL32), Version 1. Disponível em: https://talk.ictvonline.org/files/master-species-lists/m/msl/7185(accessed March, 2019).
Jenkins, G. M., Rambaut, A., Pybus, O. G., and Holmes, E. C. (2002). Taxas de Evolução molecular nos vírus ARN: a quantitative phylogenetic analysis. J. Mol. Evol. 54, 156–165. doi: 10.1007/s00239-001-0064-3
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Kumar, S., Stecher, G., e Tamura, K. (2016). MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 33, 1870–1874. doi: 10.1093/molbev/msw054
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Li, L., Diab, S., McGraw, S., Ribeiro, B., Traslavina, R., Higgins, R., et al. (2013). Astrovírus divergente associado a doença neurológica em bovinos. Esmerg. Infectar. S. 19, 1385–1392. doi: 10.3201/eid1909.130682
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Lole, K. S., de Bollinger, R. C., Pereira, R. S., Gadkari, D., Kulkarni, S. S.; Novak, N. G., et al. (1999). Genomas do tipo 1 do vírus da imunodeficiência humana de longa duração, infectados pelo subtipo C, na Índia, com evidência de recombinação intersubtipo. J. Virol. 73, 152–160.
PubMed Abstract | Google Scholar
Naccache, S. N., Peggs, K. S., Mattes, F. M., Phadke, R., Garson, J. A., Grant, P., et al. (2015). Diagnóstico de infecção por astrovírus neuroinvasivo num adulto imunocomprometido com encefalite por sequenciação de próxima geração sem preconceitos. Clin. Infectar. S. 60, 919–923. Doi: 10.1093/cid/ciu912
PubMed Abstract | CrossRef Full Text | Google Scholar
Palmieri, C., Franca, M., Uzal, F., Anderson, M., Barr, B., Woods, L., et al. (2011). Patologia e resultados imunohistoquímicos da infecção pelo vírus do Nilo Ocidental em psittaciformes. Veterinario. Pathol. 48, 975–984. doi: 10.1177/0300985810391112
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Pfaff, F., Schlottau, K., Scholes, S., Courtenay, A., Hoffmann, B., Höper, D., et al. (2017). Um novo astrovírus associado a encefalite e ganglioneurite em ovelhas domésticas. Transindo. Esmerg. S. 64, 677–682. doi: 10.1111 / tbed.12623
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Quan, P. L., Wagner, T. A., Briese, T., Torgerson, T. R., Hornig, M., Tashmukhamedova, A., et al. (2010). Encefalite por astrovírus em rapaz com agamaglobulinemia associada a X Esmerg. Infectar. S. 16, 918–925. doi: 10.3201/eid1606.091536
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Rambaut, A., Lam, T. T., Max Carvalho, L., e Pybus, O. G. (2016). Exploring The temporal structure of heterochronous sequences using TempEst (formely Path-O-Gen). Vírus Evol. 2: vew007. Doi: 10.1093/ve/vew007
PubMed Abstract / CrossRef Full Text | Google Scholar
Reuter, G., Pankovics, P., and Boros, A. (2018). Meningoencefalomielite não-supurativa (asséptica) associada a infecções neurovirulentas por astrovírus em seres humanos e animais. Clin. Microbiol. Rev. 31, e00040-18. doi: 10.1128 / CMR.00040-18
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Schlottau, K., Schulze, C., Bilk, S., Hanke, D., Höper, D., Cerveja, M., et al. (2016). Detecção de um novo astrovírus bovino numa vaca com encefalite. Transindo. Esmerg. S. 63, 253–259. doi: 10.1111 / tbed.12493
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Selimovic-Hamza, S., Boujon, C. L., Hilbe, M., Oevermann, A., e Seuberlich, T. (2017a). Frequência e fenótipo patológico da infecção por astrovírus bovino CH13/NeuroS1 em bovinos com doença neurologicamente: para avaliação da causalidade. Vírus 9:E12. doi: 10.3390/v9010012
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Selimovic-Hamza, S., Sanchez, S., Philibert, H., Clark, E. G., e Seuberlich, T. (2017b). Infecção por astrovírus bovino em bovinos com doença neurológica no oeste do Canadá. Poder. Veterinario. J. 58, 601-603.
Google acadêmico
Selimovic-Hamza, S., Bouzalas, I. G., Vandevelde, M., Oevermann, A., e Seuberlich, T. (2016). Detecção de astrovírus em casos históricos de encefalite bovina esporádica Europeia, Suíça 1958-1976. Frente. Veterinario. Ciência. 3:91. doi: 10.3389 / fvets.2016.00091
PubMed Resumo | CrossRef Texto Completo | Google Acadêmico
Seuberlich, T., Wüthrich, D., Selimovic-Hamza, S., Drögemüller, C., Oevermann, A., Bruggmann, R., et al. (2016). Identificação de um segundo vírus da encefalite associado ao astrovírus no gado bovino. Esmerg. Micróbios Infectam. 5: e5. doi: 10.1038 / emi.2016.5
PubMed Abstract / CrossRef Full Text | Google Scholar
Spinato, M. T., Vince, A., Cai, H., and ojkic, D. (2017). Identificação do astrovírus bovino em casos de encefalite não supurativa bovina no leste do Canadá. Poder. Veterinario. J. 58, 607-609.
Google acadêmico
Stein, L. T., Rech, R. R., Harrison, L., e Brown, C. C. (2010). Estudo imunohistoquímico do vírus da raiva no sistema nervoso central das espécies domésticas e selvagens. Veterinario. Pathol. 47, 630–633. doi: 10.1177/0300985810370013
PubMed Resumo | CrossRef Texto Completo | Google acadêmico
Trifinopoulos, J., Nguyen, L. T., von Haeseler, A., e Minh, B. P. (2016). W-IQ-TREE: a fast online phylogenetic tool for maximum likely analysis. Nucleic Acids Res. 44, W232–W235. doi: 10.1093/nar / gkw256
PubMed Abstract | CrossRef Full Text | Google Scholar
Tse, H., Chan, W. M., Tsoi, H. W., Fan, R. Y., Lau, C. C., Lau, S. K., et al. (2011). Redescoberta e caracterização genómica dos astrovirus bovinos. J. Gen. Virol. 92 (Pt 8), 1888-1998. doi: 10.1099 / vir.0.030817-0
PubMed Abstract | CrossRef Full Text | Google Scholar