Biologi för Majors i
lärandemål
- förstå skillnaden mellan pre-mRNA och mRNA
efter transkription måste eukaryota pre-mRNA genomgå flera bearbetningssteg innan de kan översättas. Eukaryota (och prokaryota) tRNA och rRNA genomgår också bearbetning innan de kan fungera som komponenter i proteinsyntesmaskinen.
mRNA-bearbetning
den eukaryota pre-mRNA genomgår omfattande bearbetning innan den är redo att översättas. De ytterligare stegen som är involverade i eukaryot mRNA-mognad skapar en molekyl med en mycket längre halveringstid än ett prokaryot mRNA. Eukaryota mRNA varar i flera timmar, medan den typiska E. coli mRNA varar inte mer än fem sekunder.
pre-mRNA beläggs först i RNA-stabiliserande proteiner; dessa skyddar pre-mRNA från nedbrytning medan det bearbetas och exporteras ut ur kärnan. De tre viktigaste stegen för pre-mRNA-bearbetning är tillsatsen av stabiliserande och signalerande faktorer vid molekylens 5′ och 3′ ändar och avlägsnandet av mellanliggande sekvenser som inte specificerar lämpliga aminosyror. I sällsynta fall kan mRNA-transkriptet ”redigeras” efter att det har transkriberats.
5 ’Capping
medan pre-mRNA fortfarande syntetiseras, läggs en 7-metylguanosinlock till 5′ – änden av det växande transkriptet med en fosfatlänk. Denna del (funktionell grupp) skyddar det framväxande mRNA från nedbrytning. Dessutom känner faktorer som är involverade i proteinsyntes igen locket för att hjälpa till att initiera översättning av ribosomer.
3 ’ Poly-a-svans
när förlängningen är klar klyvs pre-mRNA av ett endonukleas mellan en aauaaa-konsensussekvens och en GU-rik sekvens och lämnar AAUAAA-sekvensen på pre-mRNA. Ett enzym som kallas poly-a-polymeras lägger sedan till en sträng av cirka 200 A-rester, kallad poly-a-svansen. Denna modifiering skyddar vidare pre-mRNA från nedbrytning och signalerar exporten av de cellulära faktorer som transkriptet behöver till cytoplasman.
pre-mRNA Splitsning
eukaryota gener består av exoner, som motsvarar proteinkodande sekvenser (ex-on betyder att de uttrycks) och mellanliggande sekvenser som kallas introner (intron betecknar deras mellanliggande roll), som kan vara involverade i genreglering men avlägsnas från pre-mRNA under bearbetning. Intronsekvenser i mRNA kodar inte funktionella proteiner.
upptäckten av introner kom som en överraskning för forskare på 1970-talet som förväntade sig att pre-mRNA skulle specificera proteinsekvenser utan vidare bearbetning, som de hade observerat i prokaryoter. Generna av högre eukaryoter innehåller ofta en eller flera introner. Dessa regioner kan motsvara reglerande sekvenser; emellertid är den biologiska betydelsen av att ha många introner eller ha mycket långa introner i en gen oklar. Det är möjligt att introner saktar ner genuttryck eftersom det tar längre tid att transkribera pre-mRNA med massor av introner. Alternativt kan introner vara icke-funktionella sekvensrester kvar från fusionen av forntida gener under hela evolutionen. Detta stöds av det faktum att separata exoner ofta kodar separata proteinunderenheter eller domäner. För det mesta kan sekvenserna av introner muteras utan att i slutändan påverka proteinprodukten.
alla pre-mRNAs introner måste avlägsnas fullständigt och exakt före proteinsyntes. Om processen felar med till och med en enda nukleotid, skulle läsramen för de återförenade exonerna skiftas, och det resulterande proteinet skulle vara dysfunktionellt. Processen att ta bort introner och återansluta exoner kallas skarvning (Figur 1). Introner avlägsnas och försämras medan pre-mRNA fortfarande finns i kärnan. Splitsning sker genom en sekvensspecifik mekanism som säkerställer introner kommer att tas bort och exoner återförenas med noggrannheten och precisionen hos en enda nukleotid. Splitsningen av pre-mRNA utförs av komplex av proteiner och RNA-molekyler som kallas spliceosomer.
öva fråga
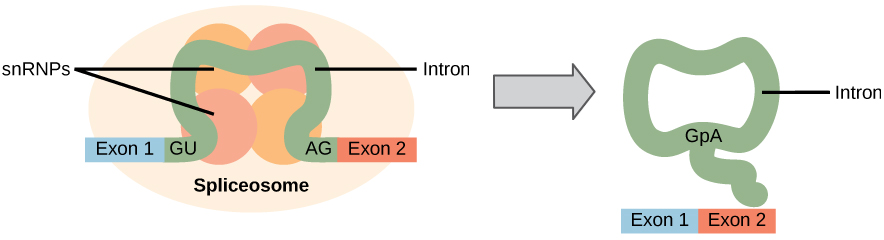
Figur 1. Pre-mRNA Splitsning innebär exakt avlägsnande av introner från det primära RNA-transkriptet. Splitsningsprocessen katalyseras av proteinkomplex som kallas spliceosomer som består av proteiner och RNA-molekyler som kallas snRNAs. Spliceosomer känner igen sekvenser vid 5′ och 3 ’ slutet av intron.
fel i skarvning är inblandade i cancer och andra mänskliga sjukdomar. Vilka typer av mutationer kan leda till skarvfel?
Observera att mer än 70 enskilda introner kan vara närvarande, och var och en måste genomgå splicingprocessen—förutom 5′ capping och tillsatsen av en poly-a—svans-bara för att generera en enda, översättbar mRNA-molekyl.
RNA-redigering i trypanosomer
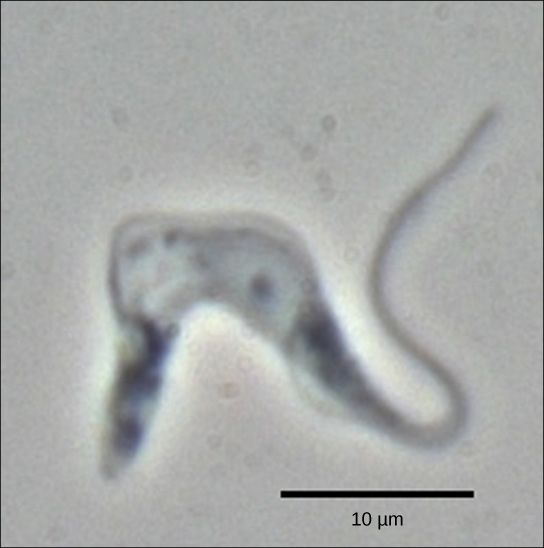
Figur 2. Trypanosoma brucei är orsakssambandet till sömnsjuka hos människor. MRNA för denna patogen måste modifieras genom tillsats av nukleotider innan proteinsyntes kan uppstå. (kredit: ändring av arbete av Torsten Ochsenreiter)
trypanosomerna är en grupp protozoer som inkluderar patogenen Trypanosoma brucei, vilket orsakar sömnsjukdom hos människor (Figur 2). Trypanosomer, och praktiskt taget alla andra eukaryoter, har organeller som kallas mitokondrier som förser cellen med kemisk energi. Mitokondrier är organeller som uttrycker sitt eget DNA och tros vara resterna av ett symbiotiskt förhållande mellan en eukaryot och en uppslukad prokaryot. Mitokondriellt DNA av trypanosomer uppvisar ett intressant undantag från den centrala dogmen: deras pre-mRNA har inte rätt information för att specificera ett funktionellt protein. Vanligtvis beror detta på att mRNA saknar flera u-nukleotider. Cellen utför ett ytterligare RNA-bearbetningssteg som kallas RNA-redigering för att avhjälpa detta.
andra gener i mitokondriellt genom kodar 40-till 80-nukleotidguide rna. En eller flera av dessa molekyler interagerar genom komplementär basparning med några av nukleotiderna i pre-mRNA-transkriptet. Guide-RNA har emellertid mer en nukleotider än pre-mRNA har u-nukleotider att binda med. I dessa regioner slingrar styr-RNA ut. 3 ’ – ändarna på styr-rna har en lång poly-U-svans, och dessa U-baser sätts in i regioner i pre-mRNA-transkriptet där styr-RNA är loopade. Denna process förmedlas helt av RNA-molekyler. Det vill säga guide rna—snarare än proteiner—fungerar som katalysatorer i RNA-redigering.
RNA-redigering är inte bara ett fenomen av trypanosomer. I mitokondrier hos vissa växter redigeras nästan alla pre-mRNA. RNA-redigering har också identifierats hos däggdjur som råttor, kaniner och till och med människor. Vad kan vara den evolutionära orsaken till detta ytterligare steg i pre-mRNA-bearbetning? En möjlighet är att mitokondrierna, som är rester av forntida prokaryoter, har en lika gammal RNA-baserad metod för att reglera genuttryck. Till stöd för denna hypotes skiljer sig ändringar av pre-mRNA beroende på cellulära förhållanden. Även om spekulativ, processen för RNA-redigering kan vara en kvarleva från en primordial tid då RNA-molekyler, i stället för proteiner, var ansvariga för katalyserande reaktioner.
Prova Det
Bidra!
förbättra denna sidalär dig mer