Creative Proteomics Blog
sekvensen av aminosyror i ett protein eller peptid kan identifieras genom Edman-nedbrytning, som utvecklades av Pehr Edman. Denna metod kan märka och klyva peptiden från N-terminal utan att störa peptidbindningarna mellan andra aminosyrarester. Edman-nedbrytningsreaktionen automatiserades 1967 av Edman och Beggs. Numera används den automatiserade Edman-nedbrytningen (proteinsekvensatorn) i stor utsträckning, och den kan sekvensera peptider upp till 50 aminosyror.
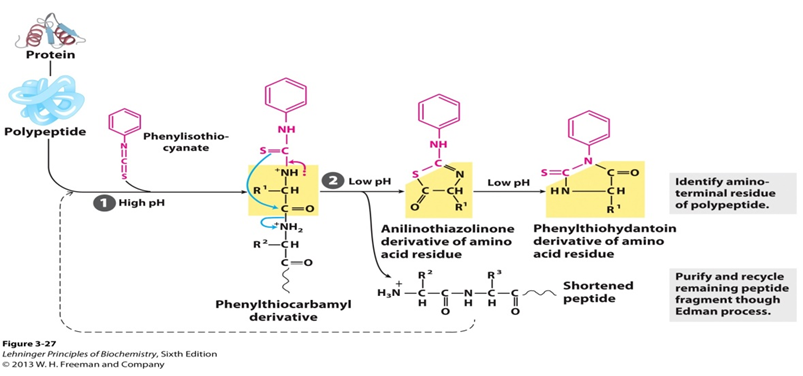
cyklisk nedbrytning av peptider baserat på reaktionen av fenylisotiocyanat med den fria aminogruppen i den N-terminala återstoden så att aminosyror avlägsnas en i taget och identifieras som deras fenyltiohydantoinderivat. Med tanke på den specifika processen reageras en oladdad peptid med fenylisotiocyanat (PITC) vid aminoterminalen under milt alkaliska betingelser för att ge ett fenyltiokarbamoylderivat (PTC-peptid). Därefter attackerar tiokarbonylsvavlet i derivatet under sura förhållanden karbonylkolet i den N-terminala aminosyran. Den första aminosyran klyvs som anilinotiazolinonderivat (ATZ-aminosyra) och resten av peptiden kan isoleras och utsättas för nästa nedbrytningscykel. En gång bildad är detta tiazolonderivat stabilare än fenyltiokarbamylderivat. ATZ-aminosyran avlägsnas sedan genom extraktion med etylakerat och omvandlas till ett fenyltiohydantoinderivat (PTH-aminosyra). Och kromatografin kan användas för att identifiera PTH-återstoden som genereras av varje cykel.
när det gäller den automatiserade Edman-nedbrytningen kan proteiner analyseras genom att applicera dem i lösning på ett TFA-filter och sedan laddas på Edman-sekvenseringsinstrumentet. Proteiner i blandningar separeras först med 1D-eller 2D-geler och blottas sedan på ett PVDF-membran. Proteinerna detekteras av coomassie blue, Amido black eller Poncau s färgning och proteinerna av intresse skärs ut och PVDF-membranstycket laddas på Edman sequencer.
med masspektrometri utvecklades började användningen av Edman-nedbrytningssekvensering minska. Det stillbilder förblir dock metoderna för flera typer av proteinstrukturanalysapplikationer. Det kan användas för att verifiera den N-terminala gränsen för rekombinanta proteiner eller bestämma N-terminalen för proteasresistenta domäner, särskilt när proteinet eller domänen är >40 till 80 kDa eller inte lätt kan renas. Det kan också användas för att identifiera det nya N-terminala och proteolytiska klyvningsstället i proteinfragmenten. Dessutom, när det gäller vissa nya proteiner och peptider där sekvensdatabaser inte är tillgängliga för MS/MS-databassökning, kan Edman-nedbrytning användas för analys.
den N-terminala aminosyran i proteinet kan klyvas av. Således identifierar den första cykeln i processen sålunda den exakta N-terminala aminosyran. Dessutom, eftersom de frisatta aminosyrorna identifieras och kvantifieras genom kromatografi, kan aminosyrorna med identisk molekylvikt identifieras. Isoleucin och leucin har till exempel en massa av 113 Da, men de har olika retentionstid. Dessutom kan Edman-sekvensering utföras på PVDF-blottar från 1D-och 2D-geler, vilket möjliggör N-terminal sekvensering av proteiner i blandningen. Emellertid kommer Edman-nedbrytningssekvensering inte att vara tillgänglig när peptiden vars N-terminal har modifierats kemiskt, såsom acetylering. Och eftersom PITC inte kan reaktivt med icke-asiatisk-aminosyra, sekvensering kommer att sluta om en icke-asiatisk-aminosyra påträffas som isoaspartiksyra. Dessutom kan större proteiner inte sekvenseras av Edman-sekvenseringen.
på Creative Proteomics ger vår professionella proteinsekvenseringsplattform N-terminal sekvensanalys av Edman-nedbrytning eller masspektrometri (MS), med komplementära fördelar med varandra. Vårt team av experter med lång erfarenhet kan hjälpa dig att förstå vad du försöker undersöka och uppfylla dina krav. Dessutom kan vi tillhandahålla andra proteinidentifieringstjänster, inklusive:
- peptid massa fingeravtryck (PMF) tjänst
- sekvensanalys av peptider eller proteiner
- de novo protein sekvensanalys tjänst
- Shotgun protein identifiering tjänst
- membran proteomics tjänst